Описание структуры гипотетического белка A.pernix
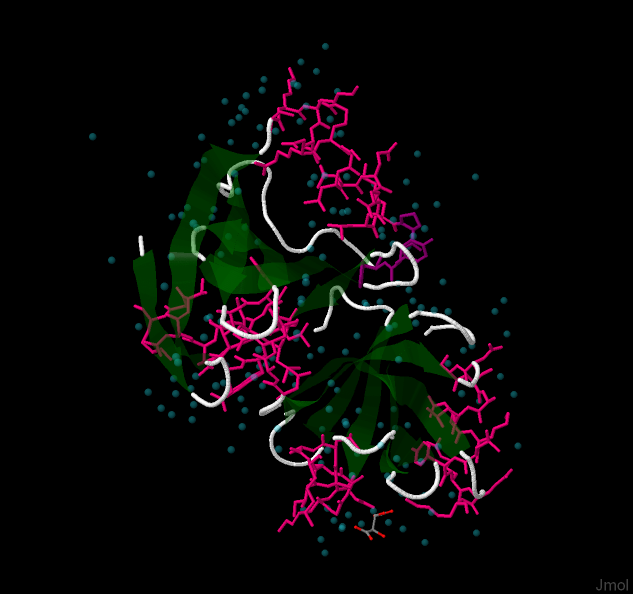
На рисунке 1 показана пространственная структура белка. Идентификатор белка в Protein Data Base — 2cx1.
Jmol-скрипт для создания данной схемы из стандартного изображения, прочитанного программой из pdb-файла:
$ background black
$ select water
$ cpk 100
$ color [36, 228, 242]
$ color translucent 0.7
$ select ligand
$ cpk 50
$ select sheets
$ color green
$ color translucent 0.5
$ select protein not (helix or sheet)
$ cartoons
$ select helix
$ cartoons off
$ wireframe 50
$ rotate x 50
Голубым цветом на рисунке 1 обозначены полупрозрачные молекулы воды, α-спиральные структуры (helix) проволочной моделью, β-листы (sheets) — зеленым цветом. Мелкие ионы (ligand) видны в нижней части картинки.
Из pdb-файла с информацией о составе белка можно извлечь эти прекрасные строчки:
Title crystal structure of a pua domain (ape0525) from the Aeropyrum pernix 2 K1 (tartrate complex) [Название структуры]
Compound mol_id: 1; molecule: hypothetical protein ape0525; chain: a; engineered: yes [Состав молекул белка]
Source mol_id: 1; organism_scientific: Aeropyrum pernix; organism_taxid: 272557; strain: K1; gene: ape0525;
expression_system: escherichia coli; expression_system_taxid: 562; expression_system_strain: b834(de3);
expression_system_vector_type: plasmid; expression_system_plasmid: pet15b
Experiment data x-ray diffraction [Метод расшифровки структуры]
Remark 2 resolution. 1.80 angstroms. [Разрешение для метода рентгеноструктурного анализа]
Formula 1 MSE 3(C5 H11 N O2 Se) [тут и далее: формулы малых молекул]
Formula 2 TLA C4 H6 O6
Formula 3 HOH *178(H2 O)
На этой странице рассмотрены внутримолекулярные взаимодействия в белке.