Заданиe 1
С помощью fiber из пакета 3DNA получим три файла: gatc-a.pdb, gatc-b.pdb и gc-z.pdb (программа не умеет делать только GATC структуру в Z-форме).
Заданиe 2
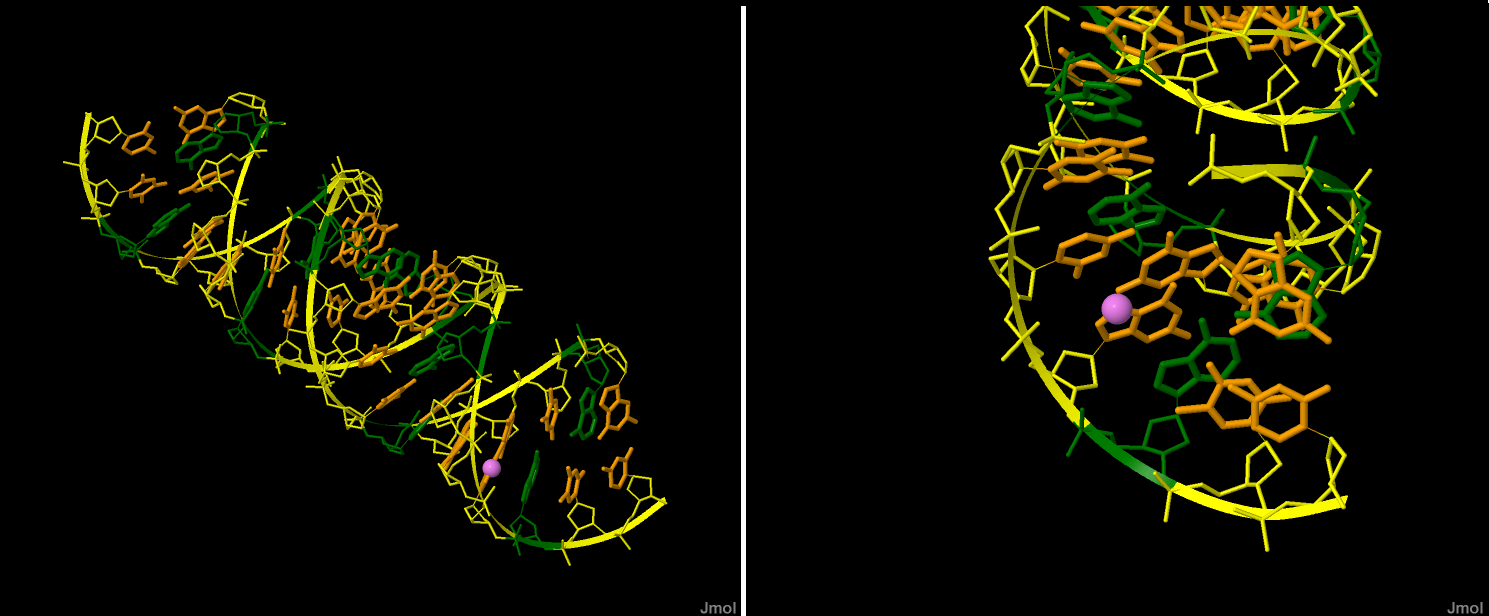
На рисунке 2 показана структура A-формы ДНК. С помощью средств Jmol желтым цветом выделен сахаро-фосфатный остов (backbone), зеленым — все аденины. Рыжим wireframe обозначены все азотистые основания. Фиолетовый шар — атом N7 на первом гуанине в последовательности ДНК.
Далее необходимо скачать pdb-файлы двух структур. 1DFM — это комплекс ДНК с белком эндонуклеазой рестрикции. 2СV0 — тРНК в комплексе с глутамил-тРНК синтетазой из Thermus thermophilus и L-глутаматом. Разрывов в нуклеиновых кислотах в этих двух структурах нет.
Для дальнейшей работы из этих структур нужно удалить все, что не является нуклеиновой кислотой. В разделе "REMARK" обоих файлов указано, что гетероатомы и аминокислоты находятся на цепях A и В, их-то мы и уберем. Получившиеся файлы: 1DFM-dna.pdb, 2CV0-rna.pdb.
Заданиe 3
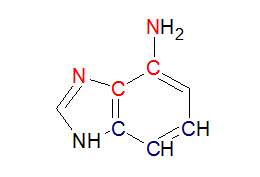
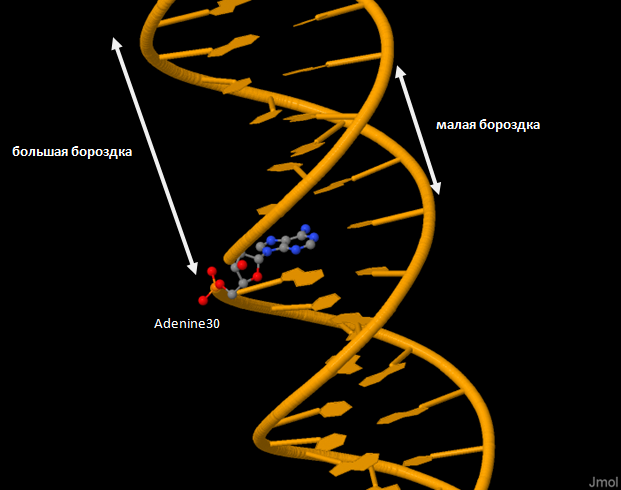
Необходимо выбрать любой аденин в цепи ДНК B-формы и разобраться, какие атомы смотрят в сторону большой бороздки, а какие — в сторону малой. Выбран остаток аденин под номером 30 (см. рисунок 4, 5). На рисунке 3 структурная формула с окраской по ориентации атомов. В сторону большой бороздки обращены атомы A30.C6, A30.N6, A30.C5 и A30.N7, A30.C8 (эти атомы отмечены красным цветом). В сторону малой бороздки обращены атомы A30.C2, A30.N3, A30.C4 (отмечены синим). Оставшиеся два атома расположены посередине, приблизительно между двумя бороздками.
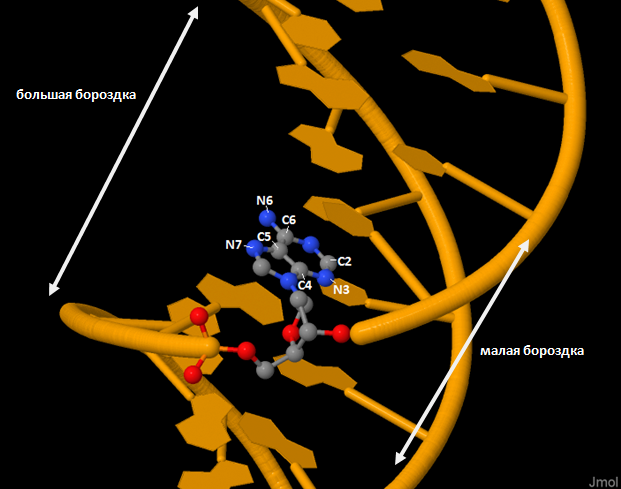
В A-форме ДНК ориентация атомов аденина получилась точно такая же. Стоит заметить, что в A-форме более глубокая бороздка (большая) заметно уже второй, малой. В структуре Z-формы нет аденинов, но только N2-атомы гуанина и O2-атомы цитозина обращены в сторону большой (более глубокой, но также более узкой) бороздки, все основание же направлено в сторону малой бороздки.
В следующем упражнении необходимо изучить все три формы ДНК. Результаты отражены в таблице 1.
Таблица 1. Информация о поиске гомологов в PsiBLAST.
| A-форма | B-форма | Z-форма | |
| Закрученность | правая | левая | левая |
| Шаг спирали, A | 28.05 | 33.80 | 43.56 |
| Оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.96 | 17.21 | 7.20 |
| Ширина малой бороздки | 16.61 | 11.69 | 17.60 |
В таблице 2 приведены значения торсионных углов аденина, посчитанные с помощью Jmol.
Таблица 2. Торсионные углы для аденина.
| α (O3'–P–O5'–C5') | β (O5'–C5') | γ (O5'–C5'–C4'–C3') | δ (C4'–C3') | ε (C4'–C3'–O3'–P) | ζ (O3–P) | χ (O4'–C1'–N9–C4) | |
| А-ДНК | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-ДНК | -60.0 | -163.4 | 31.1 | 143.4 | -140.8 | -160.6 | -98.0 |
Значения явно получились не такими, как в презентации. Возможно, причиной послужило то, что там даны углы в теоретической молекуле, а здесь кристаллизованная ДНК из комплекса.
Заданиe 4
Упражнение 1
С помощью программ find_pair и analyze получим файлы с разной информацией о нуклеиновых кислотах в 1DFM и 2CV0.
Средние значения торсионных углов отражены в таблице 3.
Таблица 2. Средние значения торсионных углов. (Excel-файлы со значениями всех углов: для 1DFM, для 2CV0)
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| 1DFM-DNA | -60,2 | 3,4 | 51,1 | 135,6 | -63,6 | -102,2 | -108,6 |
| 2CV0-tRNA | -50,5 | 65,1 | 47,0 | 86,2 | -150,6 | -72,1 | -139,6 |
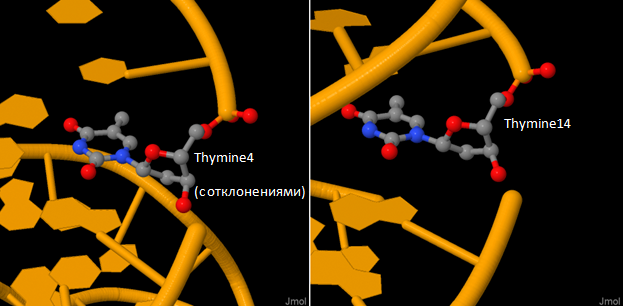
В ДНК наибольшие отклонения от среднего по трем углам (alpha, gamma, chi) имеет тимин 4 (см. рисунок 6).
В РНК это разные атомы по отклонению каждого угла: С85, G22, U53, C111, G39, C92 и G11.
Упражнение 2
Рассмотрим структуру тРНК. Из выходного файла find_pair можно выяснить позиции нуклеотидов, образующих водородные связи.
Акцепторный стебель состоит из участка 501-507 и комплементарного ему участка 566-572:
(0.011) C:.501_:[..G]G——-C[..C]:.572_:C (0.008)
(0.008) C:.502_:[..G]G-*—-U[..U]:.571_:C (0.007)
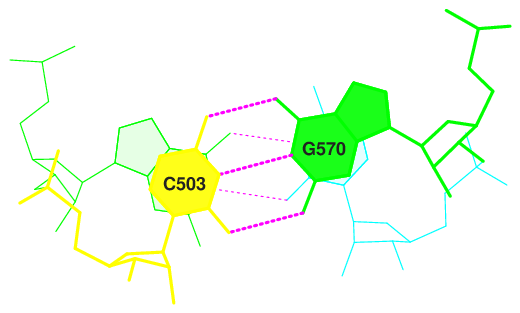
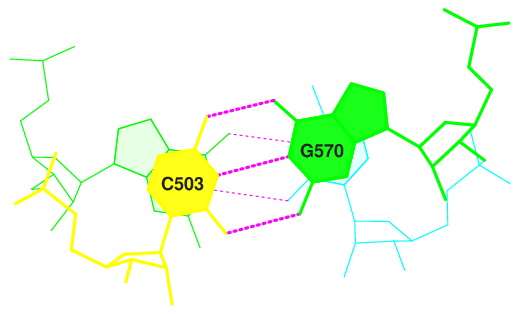
(0.005) C:.503_:[..C]C——-G[..G]:.570_:C (0.004)
(0.005) C:.504_:[..C]C——-G[..G]:.569_:C (0.008)
(0.007) C:.505_:[..C]C——-G[..G]:.568_:C (0.009)
(0.006) C:.506_:[..C]C——-G[..G]:.567_:C (0.004)
(0.004) C:.507_:[..A]Ax——U[..U]:.566_:C (0.004)
Т-стебель из 549-553 и 561-565:
(0.008) C:.549_:[..G]G——-C[..C]:.565_:C (0.003)
(0.006) C:.550_:[..G]G——-C[..C]:.564_:C (0.005)
(0.005) C:.551_:[..G]G——-C[..C]:.563_:C (0.002)
(0.003) C:.552_:[..G]G——-C[..C]:.562_:C (0.004)
(0.008) C:.553_:[..G]G——xC[..C]:.561_:C (0.004)
D-стебель из 510-513 и 522-525:
(0.012) C:.510_:[..G]G——-C[..C]:.525_:C (0.005)
(0.005) C:.511_:[..U]U——-A[..A]:.524_:C (0.006)
(0.005) C:.512_:[..C]C——-G[..G]:.523_:C (0.010)
(0.007) C:.513_:[..U]U-*—xG[..G]:.522_:C (0.013)
Антикодоновый стебель из 539-544 и 526-531:
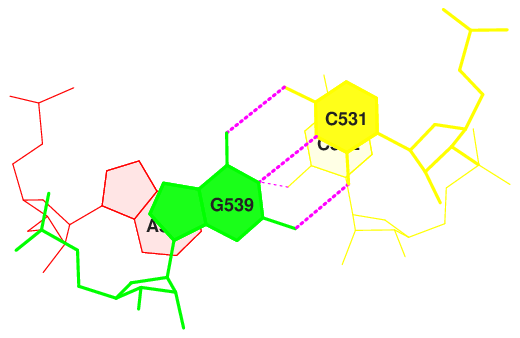
(0.007) C:.539_:[..G]G——-C[..C]:.531_:C (0.003)
(0.009) C:.540_:[..G]G——-C[..C]:.530_:C (0.007)
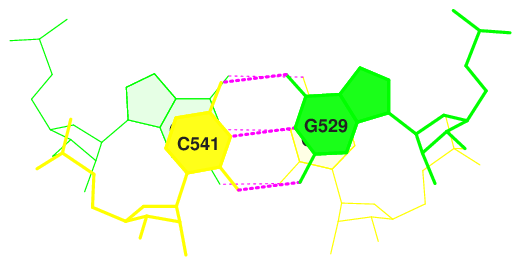
(0.003) C:.541_:[..C]C——-G[..G]:.529_:C (0.006)
(0.008) C:.542_:[..C]C——-G[..G]:.528_:C (0.007)
(0.007) C:.543_:[..G]G——-C[..C]:.527_:C (0.009)
(0.011) C:.544_:[..A]Ax*—-G[..G]:.526_:C (0.009)
Остальные водородные связи, стабилизирующие третичную структуру тРНК (не образуют стебли):
(0.009) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.014)
(0.024) C:.515_:[..G]Gx**+xC[..C]:.548_:C (0.013)
(0.012) C:.519_:[..G]Gx—-xC[..C]:.556_:C (0.003)
Также в структуре есть 5 неканонических пар оснований: 520G-571U, 513U-522G, 544A-526G, 555U-518G, 538A-532C.
Упражнение 3
В выходном файле pair_align выделим данные о величине площади "перекрывания" двух последовательных пар азотистых оснований. Пары с наибольшими значениями имеют большую площадь "перекрывания", наиболее вероятное стекинг-взаимодействие:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 2 GC/GU 7.14( 4.30) 0.00( 0.00) 0.00( 0.00) 6.63( 4.10) 13.76( 8.40) 17 GC/GC 6.19( 3.17) 0.00( 0.00) 0.00( 0.00) 6.51( 3.37) 12.69( 6.53) 30 GC/GU 7.17( 4.39) 0.00( 0.00) 0.00( 0.00) 6.49( 4.01) 13.66( 8.40) 43 AG/CC 4.74( 2.33) 0.00( 0.00) 0.00( 0.00) 5.68( 2.31) 10.42( 4.64)
Для получения изображений использовались следующие запросы:
ex_str -i stacking.pdb stepi.pdb, где i - номер шага в stacking.pdb
stack2img -cdolt stepi.pdb stepi.ps - получение изображения
convert stepi.ps stepi.png - конвертирование изображения в png