Предсказание генов эукариот.
Задание 1. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды.
Мне выдана плазмида CP016281. Она принадлежит организму Clostridium tyrobutyricum штаму W428 плазмиде pW428. Она имеет размер 62833 bp, 128 генов из которых 64 кодирующих.
С помощью программы EMBOSS я получила файлы моей плазмиды в fasta и gff форматах.
Команда: seqret embl:cp016281 -feature plasmid.gff для получения файла plasmid.gff с особенностями. Параметр -feature требуется для того, чтобы в файле были указаны особенности, embl - входная база данных.
Команда: seqret embl:cp016281 pr11.fasta для получения файла pr11.fasta
Я скачала Prodigal и запустила егочерез командную строку командой: prodigal.windows.exe -i pr11.fasta -o plasmid.pro -f sco. Параметр -f указывает на формат, -i и -o задают входной файл и выходной соответственно, минималистический формат sco для удобства дальнейшей работы. Получен файл plasmid.pro
Для сравнения предсказаний генов в базе данных и программой Prodigal я использовала Excel. Проэкт Excel. На первом листе данные из базы данных (номер по порядку, 5' конец, 3' конец, направление), на втором листе данные предсказанные программой Prodigal (так же). Для сравнения значений использовалась функция ВПР, она ищет значение из заданной ячейки в заданной таблице в первом из этой таблицы столбце и выводит заданное значение, если оно есть в заданном столбце. В столбце Е записаны совпадающие начала генов, в столбце F записаны значения концов генов из листа Prodigal для начал из Prodigal, которые есть в Genbank. В столбце G записаны значения концов из Genbank совпадающие со значениями столбца F, внизу посчитано количество совпадений. В столбце I записаны начала с учетом направления, в J столбце - концы.
| Совпадающие гены | Несовпадения по N концу | Несовпадения по C концу | Несовпадающие гены | |
| Число | 54 | 6 | 0 | 4 |
| Процент,% | 84.375% | 9.375% | 0 | 6.25% |
Анализ несовпадений
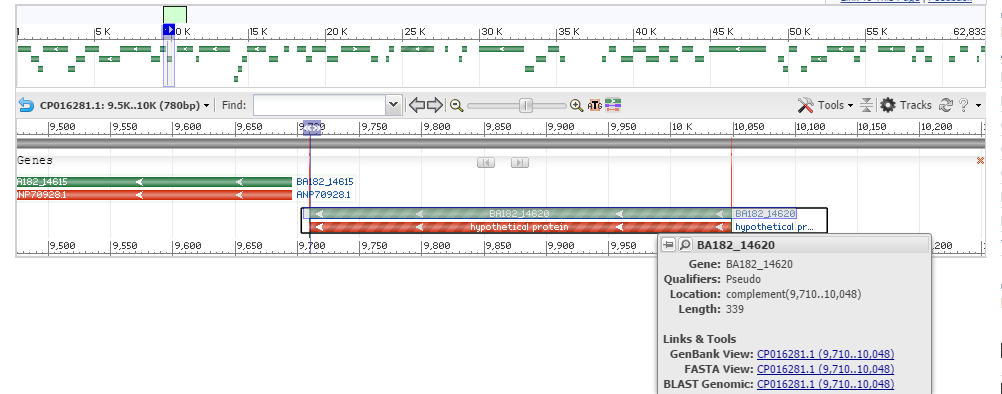
Было обнаружено 6 несовпадений по N концу, рассмотрим одно из них. Ген с координатами 9710-10048 (-). Программа Prodigal предсказала ген короче с координатами 9710-9988.

На рисунке ниже представлена последовательность этого гена. Голубым цветом выделен ген предсказанный программой Prodigal, а синим цветом кусок гена, который не был предсказан, но существует в гене. Так как направление (-) порядок обратный и цепь комплиментарна. Непредсказанный фрагмент начинается с кодона tac(aug), а предсказанный с aac(uug). Для бактерий и архей чаще всего стартовым кодоном явдяется aug, также стартовыми кодонами являются uug, gug, cug и некоторые другие. Скорее всего ошибка в предсказании гена произошла из-за того, что программа посчитала началом гена реже используемый стартовый кодон aac(uug), а не tac(aug).

Рассмотрим также ещё одно несовпадение по N концу. Ген с координатами 17348-17650 (-). Программа Prodigal предсказала ген короче с координатами 17348-17590.
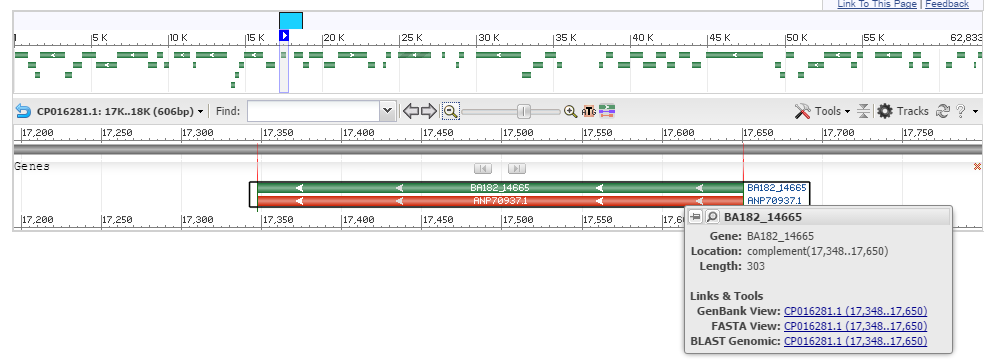
На рисунке ниже представлена последовательность этого гена. Тёмно-розовым цветом выделен ген предсказанный программой Prodigal, а светло-розовым фрагмент, который не был предсказан, но существует в гене. Так как направление (-) порядок обратный и цепь комплиментарна. И предсказанный и непредсказанный фрагменты начинаются с кодона aac(uug). Почему программа пропустила первый кодон aac и предсказала ген только со второго непонятно.

Ссылки:
© Кузнецова Ксения, 2015