
Предсказание генов эукариот.
Задание 1. Описание выданного контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг.
Мне был выдан контиг NW_006890265. Он принадлежит организму Callorhinchus milii (слоновая акула). Систематика: Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Chondrichthyes; Holocephali; Chimaeriformes; Callorhinchidae; Callorhinchus; Callorhinchus milii.

Размер контига 771106 пар оснований. Контиг содержит 26 генов, из которых 25 белок-кодирующие.
Я выбрала ген cdip1 complement(332274-401790) для которого предсказывается альтернативный сплайсинг. Длина 69,517. Ниже на рисунке представлены транскрибируемые с него мРНК и соответсвующие им изоформы белков.
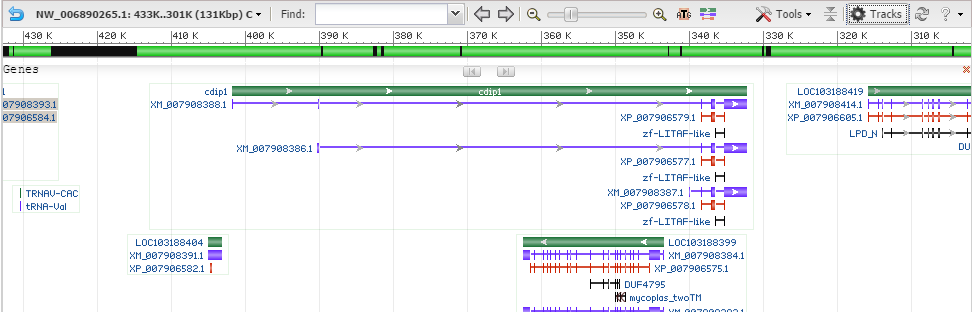
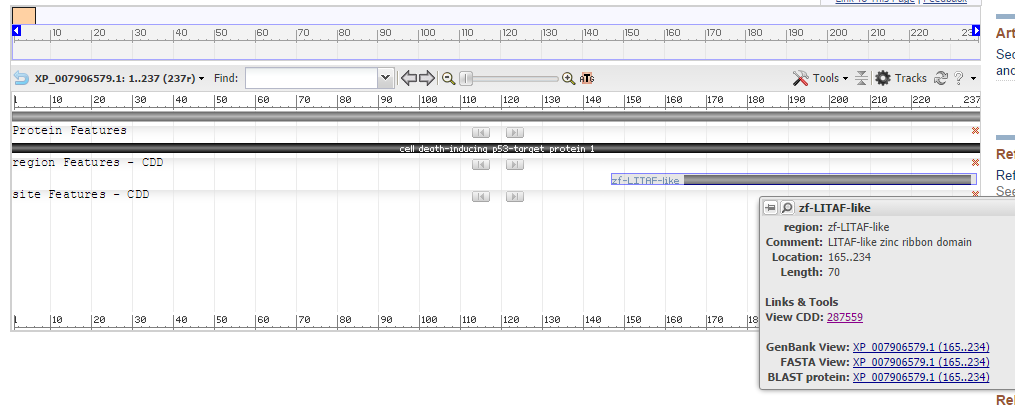
Из рисунка видно, что для этого гена возможны 3 транскрибируемые мРНК разной длины. Этим мРНК соответствуют 3 изоформа белка. На рисунке ниже представлен изоформ одного из белков.
Задание 2. Предсказание гена и белок-кодирующих областей в выданном контиге.
Предсказание гена и белок-кодирующих областей было выполнено с помощью web-сервера AUGUSTUS, используя режим Prediction. В качестве параметров модели сначала был использован мой организм Callorhinchus milii, но программа по непонятной причине выдала только 3 файла (из 6) и 2 из них с пустым содержанием. Поэтому в качестве близкого организма была выбрана рыба Danio rerio.
Параметры запуска:

Описание выбранных параметров запуска. Параметр AUGUSTUS parameter project identifier: zebrafish - это выбранный организм. Genome file - последовательность контига. User set UTR prediction - предсказание UTR, которое аннонсирует нетранслируемые участки, выбранно false по умолчанию. Report genes on: both strands - предсказывать гены на обоих цепях по умолчанию. Alternative transcripts: medium - показывает разное количество альтернативных транскриптов, было выбрано medium, для того чтобы учесть альтернативные транскрипты. Allowed gene structure: predict any number of (possibly partial) genes - предсказывать любое количество (возможно частичных) генов, для учета всех генов. Ignore conflictes with other strand: false - не учитывать перекрытие генов на комплиментарных цепях, по умолчанию.
Программа AUGUSTUS выдает архив predictions.tar.gz, в котором содержится 6 файлов: augustus.aa, augustus.cdsexons, augustus.codingseq, augustus.gbrowse, augustus.gff, augustus.gtf.
| Название | Содержимое |
| augustus.aa | для каждого предсказанного гена последовательность белков в формате FASTA |
| augustus.cdsexons | для предсказанного гена белок-кодирующие последовательности после сплайсинга (экзоны) в FASTA формате |
| augustus.codingseq | кодирующие последовательности (CDS) предсказанных генов в FASTA формате с учетом цепи |
| augustus.gff | Таблицы предсказанных генов с их различными характеристиками. Название (ген,транскрипт, старт-кодон,CDS,terminal...), координаты начала и конца, направление, идентификатор. После для каждого гена указана кодирующая последовательность и последовательность предполагаемого белка |
| augustus.gbrowse | более сжатая таблица как в augustus.gff, в которой вместо транскрипта есть мРНК |
| augustus.gtf | Таблица как в augustus.gff, но нет строчек помеченных символом # |
С сайта NCBI я скачала файл sequence.gff3 для моего контига и выбрала из него только CDS, для сравнения с предсказанием AUGUSTUS. Также CDS были выбраны из файла augustus.gbrowse. Данные я сравнивала при помощи программы excel, как в предыдущем практикуме. Ссылка на проект Excel.
В результате полностью предсказанных генов 224 (в GenBank 442 гена, а предсказано AUGUSTUS 593 гена, количество предсказаных отличается аж на порядок и это странно), причем 203 ошибки по N концу и 193 ошибки по C концу.
Предсказание AUGUSTUS для выбранного мною гена cdip1 complement(332274-401790) не совпадает с данными базы данных.
Ссылки:
© Кузнецова Ксения, 2015