IC.
Я брала информацию о выравнивании из варианта 7 пракрикума. Результа и матрицу можно найти
в таблице ниже. Для начала я нашла частоты букв в выравнивании, которые содержатся в первой
рамке таблицы. Частоты букв GC у данио-рерио (38.6%) были нам даны в прошлом практикуме.
Далее был построены матрицы с информационным содержанием отдельных букв
и колонок(просуммировала колонку). Итоговое информационное содержание получилось равным 10.1324.
Формулы для вычисления также приведены в таблице в виде рамочки.
Матрица.
Картинка LOGO для выравнивания:

3.Проверка PWM

Найденные последовательности (красными блоками выделены нужные)
 Вся выдача meme: meme
Данный мотив был получен в практикуме 6.
Вся выдача meme: meme
Данный мотив был получен в практикуме 6.
Задание 3.
a-b
В предыдущем пункте показан получившийся у меня мотив "AACTAAA". Он состоит из 7 нуклеотидов,
но предположительно CS- "AACTAA". Каждый мотив(обозначен красным прямоугольником) был найден
один раз перед поздними генами и в лидерной последовательности. Участок поиска был ограничен 100
нуклеотидами.

Видно, что вероятность найти такой мотив внутри гена не мала. Но сам мотив все еще соответствует критериям, описанным выше:
он встречается один раз до полипротеина и один раз до генов. В CS перед геном 3 присутствует замена нуклеотда A на C.
Это как раз отражено на картике Logo для мотива в предыдущем пункте. E-value был задан = 0.001, так как при более низких значениях
результата не было.
Как видно вероятность найти данную или похожую последовательность в геноме не особо маленькая, но тем не менее меньше E-value=0,001.
Однако вероятность найти мотив в нужных областях гораздо меньше этого значения.
c
Козак поздних генов.
 Козак генов человека.
Козак генов человека.

Чтобы получить Logo, я взяла область старт-кодонов вирусов с 6 нуклеотидами до и после ATG. Если сравнивать полученный мной
результат с последовательностью козак генов человека, можно отметить, что они весьма схожи в нуклеотидах
на -3, -2 позициях. Чаще всего на этих позициях встречаются A и C. Сама последовательность козак вируса
более длинная после старт-кодона.
d
Чтобы выполнить это задание, я взяла 2 наиболее близких вируса к моему в пределах рода, т. к. штамм не был найден.
BtRf-AlphaCoV/HuB2013 и BtRf-AlphaCoV/YN2012. После загрузки геномов и мотива в Fimo, были получены следующие таблицы:
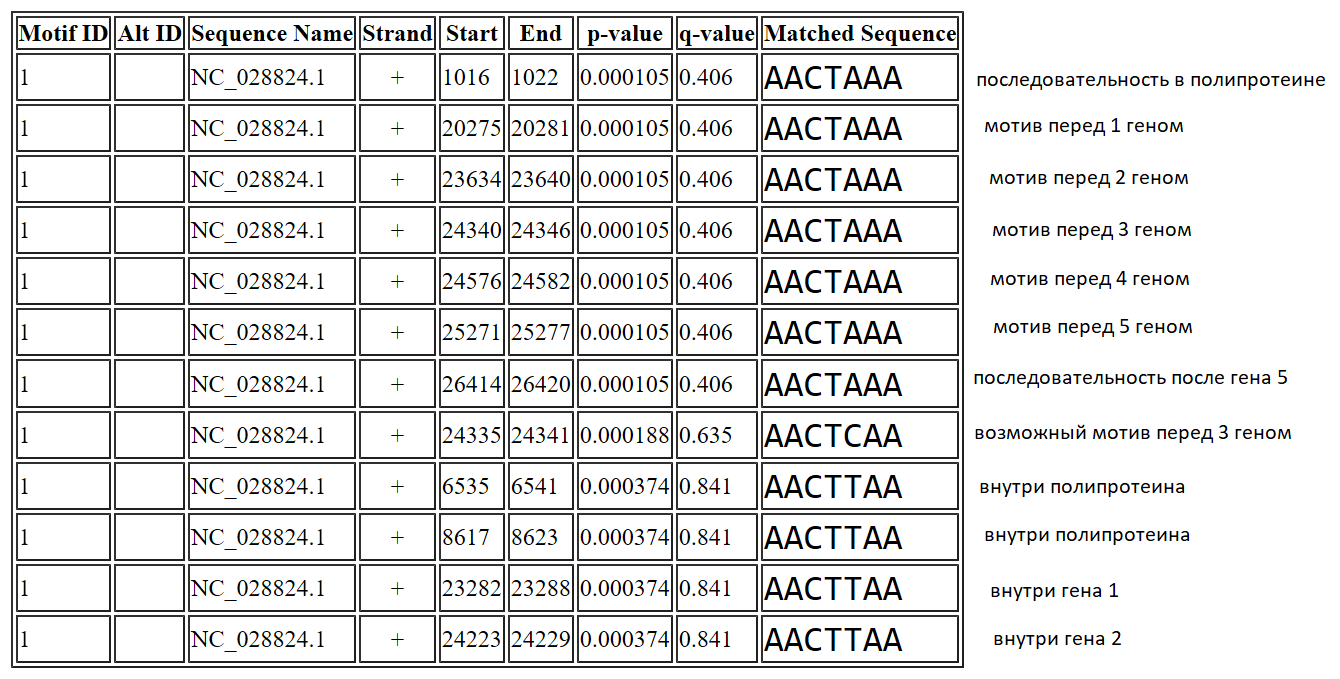
BtRf-AlphaCoV/YN2012
BtRf-AlphaCoV/HuB2013
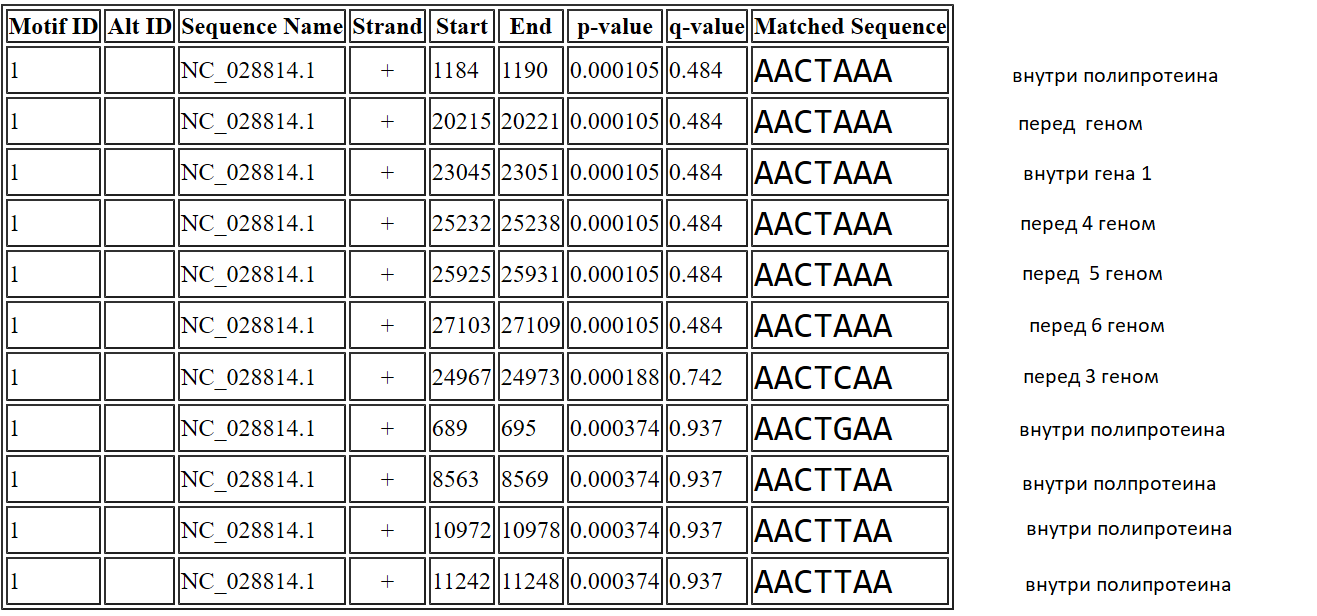
Таким образом можно сказать, что мотив не особо специфичен для вида, так как вероятности его наличия у близкородственных организмов высока,
особенно хорошо это выражено для вируса BtRf-AlphaCoV/YN2012. По результатам можно предположить, что у него идентичная CS.

