Белки Uniprot с заданным составом доменов Pfam.
1.Домен: это цинк-связывающая петля Fold group, обнаруженная в эндо-дезокси-рибонуклеазах и нуклезах HNH.
1) ID: HNH_3
2)AC: PF13392
3)Название: HNH endonuclease
4)Число последовательностей среди бактерий: 1485
2.Домен: этот домен из 60 остатков аминокислот может связываться с ДНК и находится в белках фактора транскрипции.
1) ID: AP2
2)AC: PF00847
3)Название: AP2 domain
4)Число последовательностей среди бактерий: 249
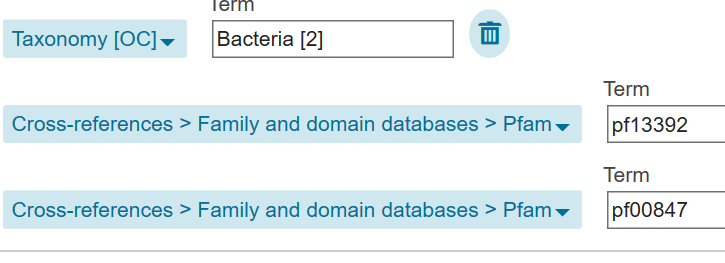
Запрос в Uniprot выглядил так:
На данном изображениии представлена архитектура домена с описанием:
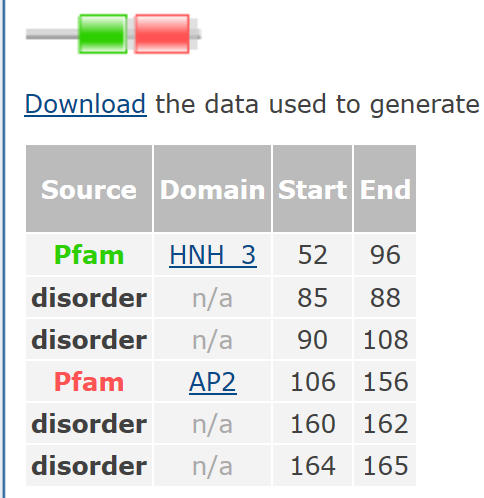
Pham нашел 164 последовательности с данной архитектурой:HNH_3, AP2 .
После загрузки доменов в Uniprot я получила уже 693 белка. Вполне ожидаемо произошло увеличение количества, по сравнению с Pham.
После очистки вручную таблицы получилось 687 белков с двумя заданными домаенами.
Таблицаф с белками.
Задание 3.
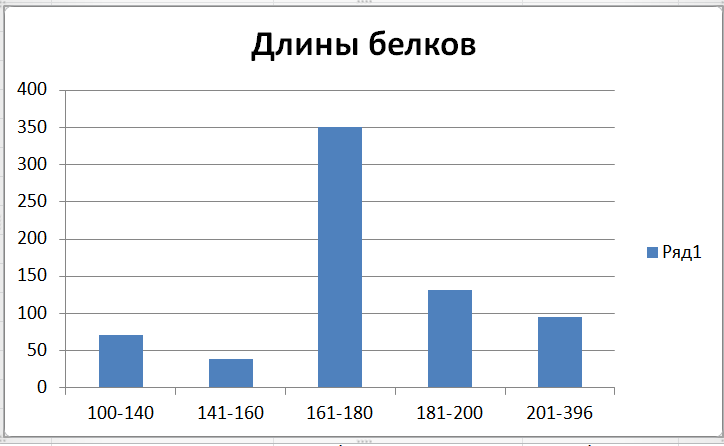
В выборку были взяты белки длиной от 161 до 180 из различных отделов.
Было взято 2-5 представителей рода. Всего было выбрано 49 белков.Их я отмечала "+" в столбце.(Лист 2)
Таблица с белками.
Таблица с белками.
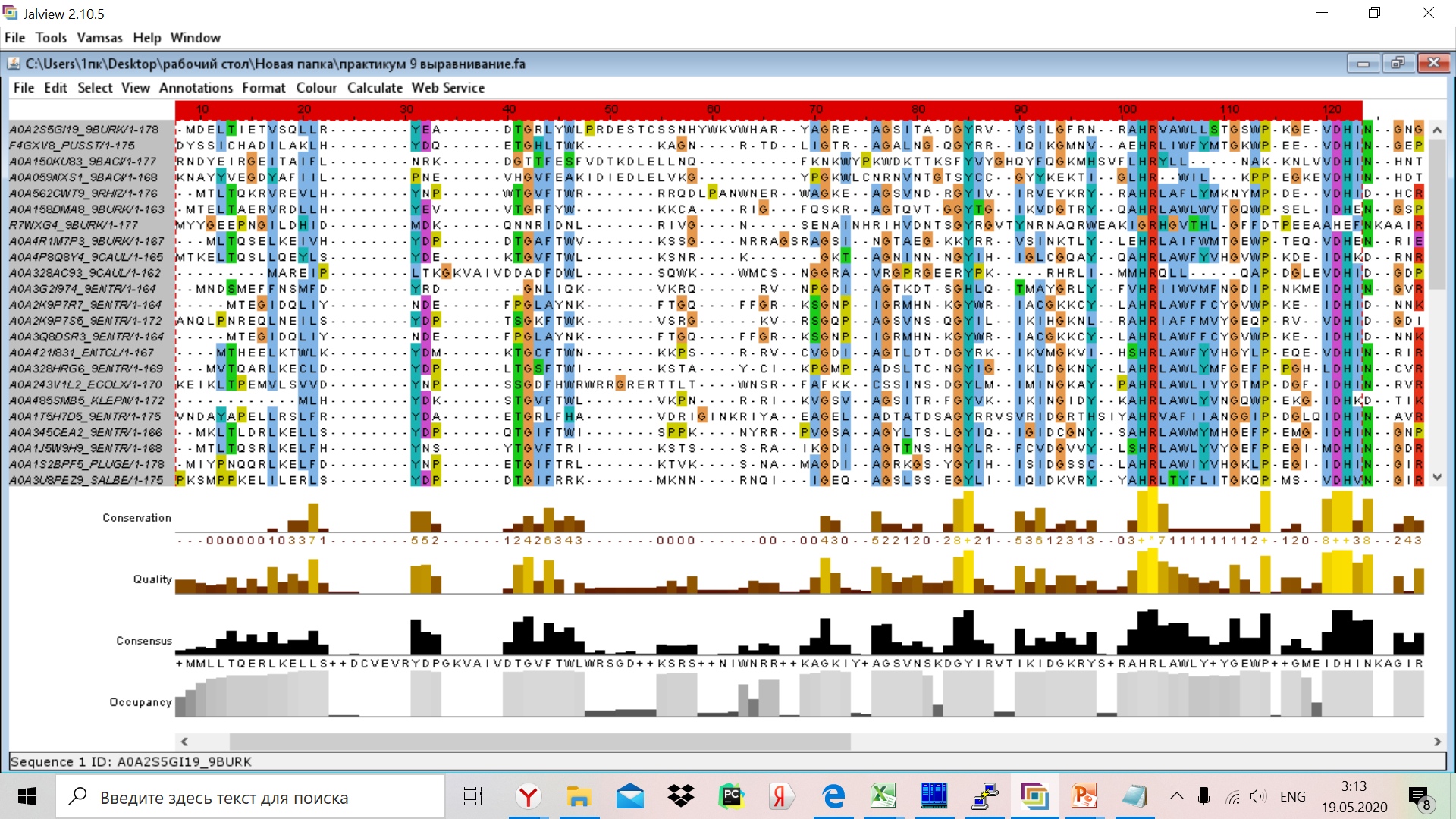
С помощью программы Jalview были загружены последовательности с данными AC и выравнены(Muscle with defaults).
Далее в программе Jalview я удалила 124 нуклеотида до первого N-концевого консервативного блока и 26 нуклеотидов после С-концевого
консервативного блока. (Ориентировалась на презентацию)
N-концевой
C-концевой
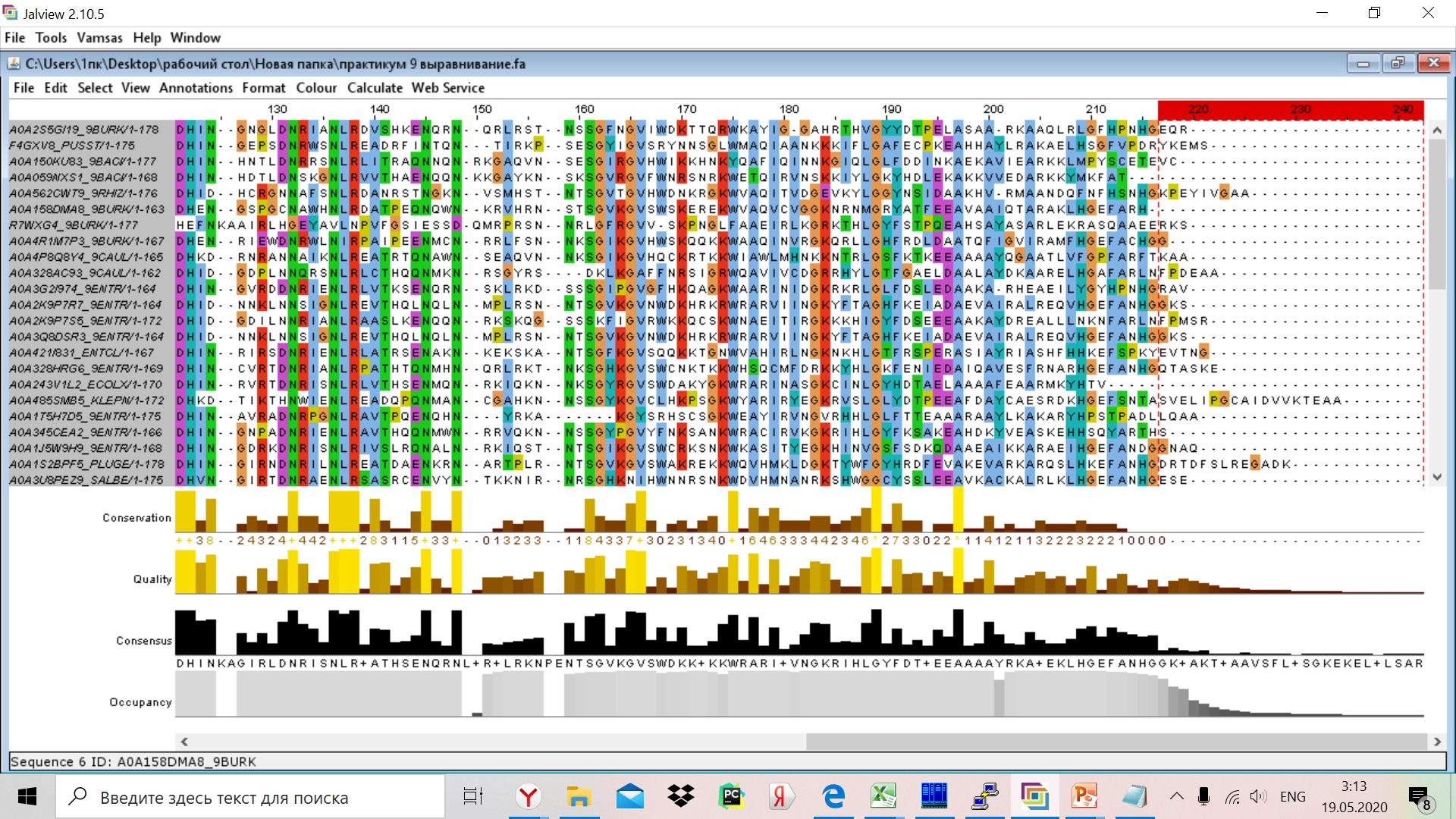
Файл с выравниваниями после обработки:
Выравнивание.
Я скачала из Uniprota домен PF00847(1508), так как число бактерий среди бактерий
с таким же доменом оказалось меньше, чем с PF13392. Запрс в Uniprot - "taxonomy:"Bacteria [2]" database:(type:pfam pf00847)"
Скачать файл с последовательностями, содержащими этот домен.
Uniprot белки.
Команды для построение HMM профиля:
1)hmm2build -g prophile public_html/term4/pr9/vyr.fa
2)hmm2calibrate prophile
3)hmm2search -E 0.1 prophile download.fasta > pr9table1.txt -
поиск среди всех белков с первый домен, тех, что содержат зданную доменную архитектуру с E-value 0.1 (рекомендованное значение).
В Excel был добавлена нужная таблица из получившегося файла и там же я вычисляла нужные значения.
HMM профиль.
Вычисления, белки, графики.
Сравнение исходной таблицы и таблицы белков (HMM профиля) сравнивались функцией Excel(одинаковые выделены цветом, а затем
1 отмечались одинаковые AC, а 0 несовпадающие).
График весов.
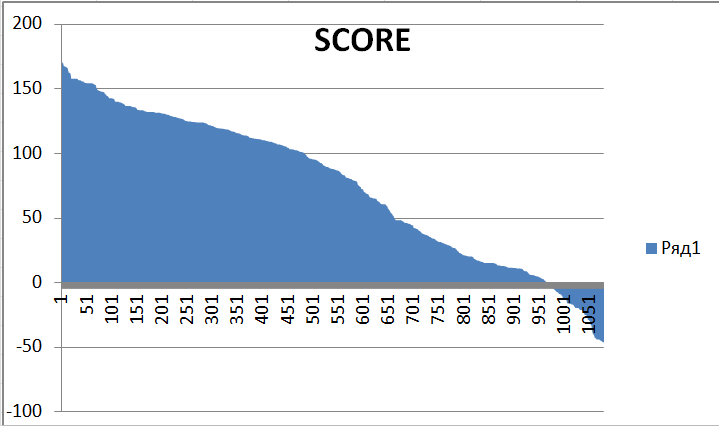
На графике была выбрана точка с весом 45,7и E-value 1,40E-11.Она расположена в месте,
где график начинает стремиться к асимптоте.(Чувствительность - 0,965625
Специфичность - 0,16553288). Определяя этут точку, я ориентировалась на наибольшее значение F1(0,928625094).Это строка 691 на
странице "Расчеты.."
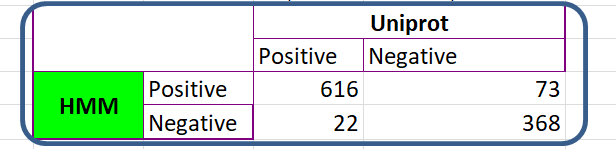
Ее значения и стали пороговыми, по которым заполнялась таблица предсказаний и истины.
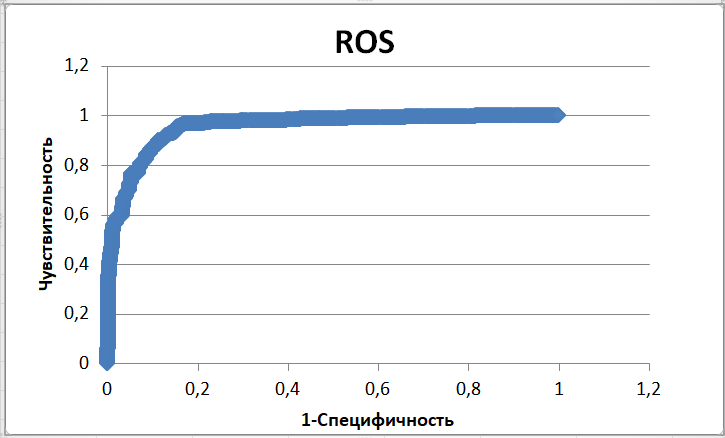
Таблица предсказаний. Порог - вес 45,7и E-value 45,7 и 1,40E-11.По ней можно судить о качестве предсказания.