В выбранных бактериях были найдены гомологи белка CLPX_BACSU. Для этого сначала был создан единый файл с протеомами бактерий proteomes.fasta . По нему с помощью команды
makeblastdb -in proteomes.fasta -out db.fasta -dbtype prot
была создана база данных для blastp.
Затем командой
blastp -query CLPX_BACSU.fasta -db db.fasta -evalue 0.001 -out result.txt
был получен файл result.txt , содержащий среди прочего мнемоники последовательностей, дающих с нашим белком выравнивания с e-value не хуже заданного (0.001). Всего их оказалось 23.
При помощи fetch sequences последовательности были извлечены в JalView, и сервисом Muscle было постороено их выравнивание (par_modern.fasta ). Для удобства в названиях последовательностей были оставлены только мнемоники.
Выравнивание было открыто в Mega, и по нему методом Maximum Likelihood method было построено филогенетическое дерево. Считая, что данное дерево реконструировано верно, можно попытаться найти на нем ортологов и паралогов.
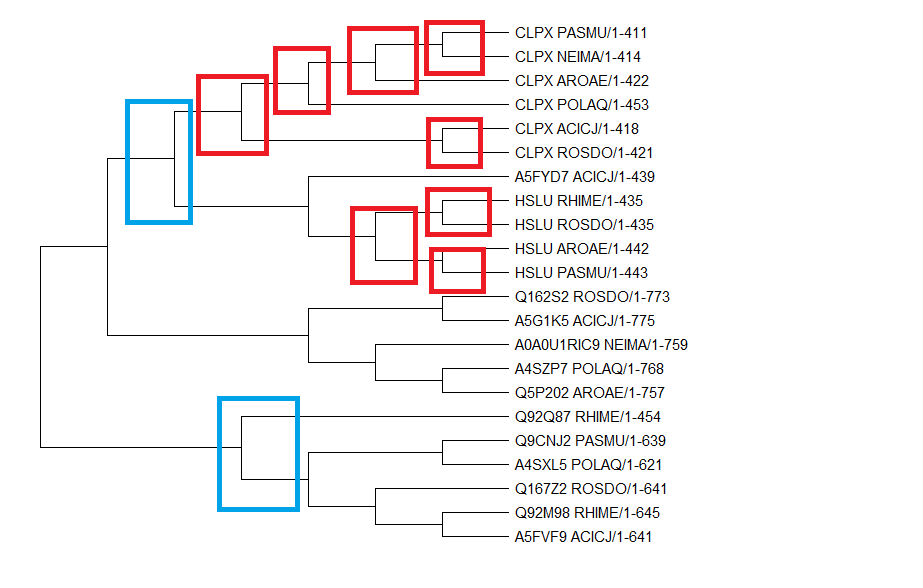
парологи: CLPX_AROAE и HSLU_AROAE Q92M98_RHIME и Q92Q97_RHIME ортогоги: CLPX_AROAE и CLPX_NEIMA HSLU_RHIME и HSLU_ROSDO
Также по имеющемуся дереву можно попытаться восстановить эволюционные события. На рисунке красными рамочками выделены некоторые примеры разделения путей эволюции в результате видообразования, а голубыми рамочками - дупликации генов.