Второй семестр
Второй семестрФилогенетические деревья, реконструированные разными способами
Эталонное выравнивание доменов
benchmark.msf:
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
|
|
|
2 |
0 |
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
|
|
|
4 |
0 |
|
|
|
|
|
|
|
|
|
* |
|
|
|
|
|
| P |
S |
M |
R |
_ |
A |
R |
C |
F |
U |
|
: |
|
E |
K |
A |
P |
S |
I |
I |
F |
I |
D |
E |
L |
D |
A |
I |
A |
A |
R |
R |
T |
N |
S |
D |
T |
S |
G |
D |
R |
E |
V |
Q |
R |
T |
- |
- |
M |
M |
Q |
L |
L |
A |
E |
L |
D |
G |
F |
D |
P |
R |
G |
|
: |
|
4 |
8 |
| T |
E |
R |
A |
1 |
_ |
C |
A |
E |
E |
|
: |
|
K |
N |
Q |
P |
A |
I |
L |
F |
I |
D |
E |
I |
D |
A |
I |
A |
P |
K |
R |
E |
K |
T |
N |
G |
E |
V |
E |
R |
R |
I |
V |
- |
- |
- |
- |
- |
S |
Q |
L |
L |
T |
L |
M |
D |
G |
V |
K |
G |
R |
S |
|
: |
|
4 |
5 |
| V |
P |
S |
4 |
_ |
S |
C |
H |
P |
O |
|
: |
|
E |
Q |
K |
P |
S |
I |
I |
F |
I |
D |
E |
I |
D |
S |
L |
C |
G |
S |
R |
S |
E |
G |
E |
S |
E |
S |
S |
R |
R |
I |
K |
T |
- |
- |
- |
- |
E |
F |
L |
V |
Q |
M |
N |
G |
V |
G |
K |
D |
E |
S |
|
: |
|
4 |
6 |
| F |
T |
S |
H |
3 |
_ |
S |
Y |
N |
Y |
|
: |
|
K |
Q |
A |
P |
C |
I |
V |
F |
I |
D |
E |
L |
D |
A |
I |
G |
K |
S |
R |
A |
S |
G |
A |
F |
M |
G |
G |
N |
D |
E |
R |
E |
Q |
T |
L |
N |
Q |
L |
L |
T |
E |
M |
D |
G |
F |
S |
A |
A |
G |
A |
|
: |
|
5 |
0 |
| R |
I |
X |
7 |
_ |
Y |
E |
A |
S |
T |
|
: |
|
S |
L |
A |
P |
C |
L |
V |
F |
F |
D |
E |
I |
D |
A |
I |
T |
P |
K |
R |
D |
G |
G |
A |
Q |
R |
E |
M |
E |
R |
R |
I |
V |
A |
Q |
L |
L |
T |
S |
M |
D |
E |
L |
T |
M |
E |
K |
T |
N |
G |
K |
|
: |
|
5 |
0 |
| |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
P |
|
6 |
6 |
F |
i |
D |
E |
6 |
D |
a |
6 |
|
|
|
R |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
6 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Построение дерева по
алгоритму UPGMA.
Вычисления и их результаты находятся в файле
UPGMA.xls
На листе DISTANCES - матрица попарных расстояний
между последовательностями, а на листе UPGMA - построение скобочной структуры дерева.
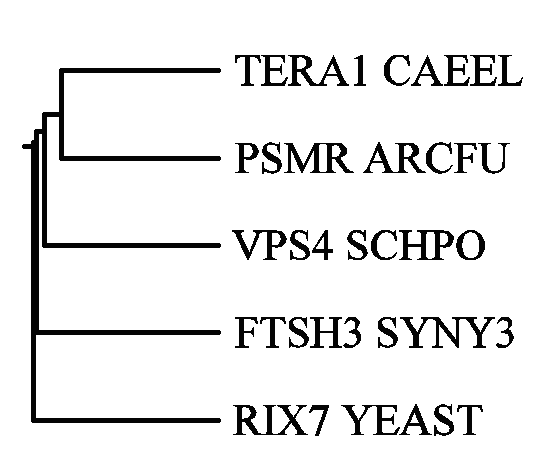
Правильная скобочная структура:
((((TERA1_CAEEL:0.315, PSMR_ARCFU:0.315):0.0325,VPS4_SCHPO:0.3475):0.0175,FTSH3_SYNY3:0.365):0.005,RIX7_YEAST:0.37);
По данной структуре дерево было визуализировано с помощью программ drawtree и drawgram (все параметры по умолчанию) из
Online-версии пакета Phylip .
При использовании программы drawtree получается неукорененное дерево, при использовании программы drawgram - укорененное.
Неукорененное дерево:
TERA1_CAEEL и PSMR_ARCFU имеют одного предка.
RIX7_YEAST и FTSH3_SYNY3 имеют общего предка.
VPS4_SCHPO отстоит от этих 2-х групп.
Укорененное дерево:
Предковый белок образовал две ветви: одну для RIX7_YEAST и FTSH3_SYNY3, другую для белка А, который образовал 2 ветви: белок Б и
VPS4_SCHPO, и сам белок Б дал 2 ветви: TERA1_CAEEL и PSMR_ARCFU.
Построение дерева по
методу Neighbour-joining
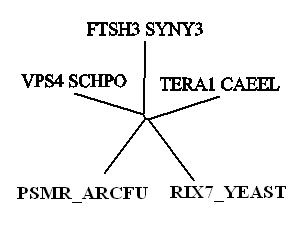
Правильная скобочная структура дерева:
((PSMR_ARCFU:0.33508,
(VPS4_SCHPO:0.34568,FTSH3_SYNY3:0.34997):0.02259):0.01415,
TERA1_CAEEL:0.35731,RIX7_YEAST:0.37378);
drawtree- неукорененное дерево, drawgram - укорененное дерево.
Неукорененное дерево:
TERA1_CAEEL и RIX7_YEAST имеют одного предка.
VPS4_SCHPO и FTSH3_SYNY3 имеют общего предка.
PSMR_ARCFU отстоит от этих 2-х групп.
Укорененное дерево получилось таким же, как в методе UPGMA.(только с разными эволюционными расстояниями)
Выводы.
Неукорененные деревья получились разными ( в
неукорененном дереве расхождение аминокислотных последовательностей произошло в другом месте) и нельзя судить об их достоверности, так как веточки очень короткие
(дерево похоже на звезду).
Деревья дают нам представление об эволюции аминокислотных последовательностях, а не организмах.
©Лавыш Дарья