 Четвертый семестр
Четвертый семестрКлассификация функций. Коды ферментов.
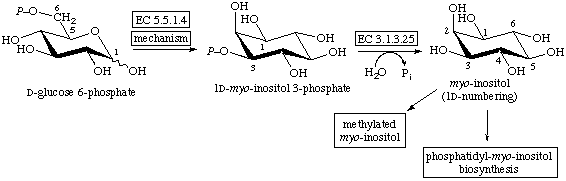
Объяснение, что значит код фермента в классификации IUPAC: 3.1.3.25
EC 3 Гидролазы.
EC 3.1 Действуют на эфирные связи.
EC 3.1.3 Гидролаза моноэфиров фосфорной кислоты.
EC 3.1.3.25 инозитфосфат (C6H13O9P) фосфатаза.
Гидролазы-ферменты, которые катализируют гидролиз различных связей.
Сравнение последовательности ферментов с одинаковым кодом из эволюционно далеких организмов
C помощью SRS по заданному коду нашла в UniProt все ферменты
из 3-х хорошо изученных модельных организмов - кишечной палочки Escherichia
coli K-12, археи Methanococcus jannaschii и человека Homo sapiens. Использовала принятые короткие имена,
что позволило легко отфильтровать разные штаммы Ecoli.
Запрос:
((([uniprot-ID:*_ECOLI] | [uniprot-ID:*_METJA]) | [uniprot-ID:*_HUMAN]) & [uniprot-ECNumber:3.1.3.25])
При поиске по коду убрали wildcards.
Выдача: 6 записей, 3 из человека, 2 из кишечной палочки, 1 из археи.
Имени гена нет только у одной последовательности A2UJH0_ECOLI.
При помощи просмотра результата SW_InterProMatches, который позволяет быстро посмотреть на все
мотивы в последовательностях, были обнаружены по одному домену Pfam в каждом белке,
кроме Q9UK71_HUMAN, доменная структура кторого, по-видимому, неизвестна.
Сравнение доменной структуры ферментов из далеких организмов
| |
UniProt ID |
UniProt AC |
Имя гена |
Первый домен |
Идентификатор Pfam |
Положение в последовательности |
| 1 |
IMPA1_HUMAN |
P29218 |
IMPA1 |
PF00459 |
5-277 |
| 2 |
IMPA2_HUMAN |
O14732, Q9UJT3 |
IMPA2 |
PF00459 |
15-282 |
| 3 |
SUHB_ECOLI |
P0ADG4, P22783, P77511, Q8X2E6 |
SUHB |
PF00459 |
1-263 |
| 4 |
SUHB_METJA |
Q57573 |
SUHB |
PF00459 |
2-249 |
Таким образом, все последовательности содержат одинаковый Pfam домен, незначительные различия наблюдаются в длине и положении.
Оценка сходства последовательностей аминокислот в гомологичных доменах.
Благодаря встроенной в SRS программе NeedleP получили попарное выравнивание доменов.
Страничка с выдачей результата.
Таблица, отражающая попарное сходство доменов PF00459:
| Домены из |
Identity |
| IMPA1_HUMAN - IMPA2_HUMAN |
141/274 (51.5%) |
| IMPA1_HUMAN - SUHB_ECOLI |
88/278 (31.7%) |
| IMPA1_HUMAN - SUHB_METJA |
70/292 (24.0%) |
| IMPA2_HUMAN - SUHB_ECOLI |
88/272 (32.4%) |
| IMPA2_HUMAN - SUHB_METJA |
72/288 (25.0%) |
| SUHB_ECOLI - SUHB_METJA |
66/280 (23.6%) |
Ферменты из разных организмов имеют довольно низкие проценты идентичности.
Интересно, что низкое попарное сходство, не является определяющим фактором в функциональной активности домена.
Ферменты с разными первичными структурами выполняют одинаковую функцию в разных организмах, т.к. они имеют
сходныме 3D структуры, которые образуют активные центры, и в большей степени определяют функции белка.
©Лавыш Дарья
