Скелеты в тулупах с красной звездой
Седлают оленей козлиной ногой.
На них с любопытством смотрел оцелот,
Пока не устроился в Аэрофлот.
Кобыла и трупоглазые жабы, "Азиатский Способ Производства"
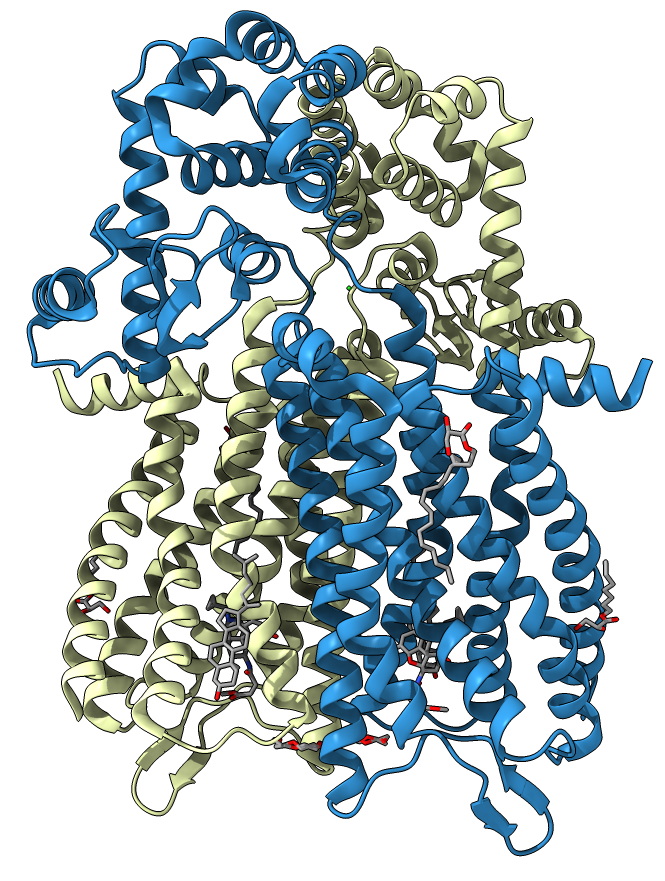
Кристаллизованный белок представляет собой димер из одинаковых субъединиц. По сравнению с углубленным карманом связывания, наблюдаемым в большинстве опубликованных к настоящему времени рецепторов, связанных с G-белками, лиганд глубоко связывается в большом, открытом кармане[2].
| Лиганд | Химическая формула | Брутто-формула | Название в номенклатуре IUPAC | Молярная масса, г/моль | Индикатор PubChem | |
| SO4 | 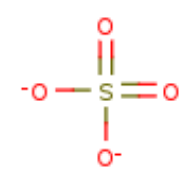 |
SO4 | SULFATE ION | 96.07 | 1117 | |
| Cl |  |
Cl | CHLORIDE ION | 35.45 | 312 | |
| MPG | 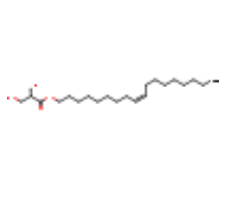 |
C21H40O4 | [(Z)-octadec-9-enyl] (2R)-2,3-bis(oxidanyl)propanoate | 356.5 | 17754086 | |
| BF0 | 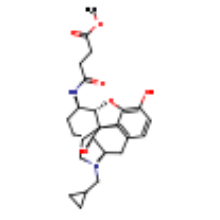 |
C25H32N2O6 | methyl 4-{[(5beta,6alpha)-17-(cyclopropylmethyl)- 3,14-dihydroxy- 4,5-epoxymorphinan-6-yl]amino}-4-oxobutanoate |
456.5 | 137348997 | |
| 1PE | 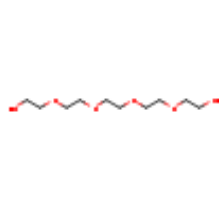 |
C10H22O6 | PENTAETHYLENE GLYCOL | 238.28 | 62551 | |
| CLR | 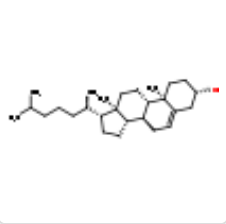 |
C27H46O | CHOLESTEROL | 386.7 | 5997 |
Для нахождения дисульфидных мостиков были выделены все цистеины белка, после был произведен ручной поиск цистеинов, образующих ковалентную связь.
При рассмотрении структуры белка был найден один цистиновый мостик, образованный остатками цистеина Cys 140 и Cys 217. Увидеть его можно в апплете, выбрав скрипт «Цистиновый мостик». Данная связь стабилизирует внеклеточный участок белка, оставляя открытым центр связывания.
Поиск водородных связей в белке производился через выделение всех атомов белка, не входящих во вторичные структуры, с последующим расчетом водородных связей.
Белок, в целом, имеет большое количество альфа-спиралей, которые поддерживаются водородными связями. Помимо водородных связей, служащих для поддержания вторичной структуры, существуют также водородные связи между боковыми радикалами аминокислот: например, между треонином Thr 1054 и валином Val 1057, между аспарагином Asn 1020 и тирозином Tyr 1024 (всего водородных связей вне вторичных структур 14). Также в объекте существует водородная связь между боковыми радикалами треонинов обоих цепей (белок представляет собой димер). Эти водородную связь можно увидеть в апплете, выбрав скрипт «Водородные связи». Параметры этих связей (Таблица 2) соответствуют общепринятым[12, 13].
Функциональная роль связи между треонинами может состоять в стабилизации структуры димера.
| Взаимодействующие аминокислоты | Атомы-участники водородной связи | Длина, Å | Угол, градусы | |
| Thr 249:1 - Thr 249:2 | O - O | 2.6 | 134.5 | |
| Thr 1054 - Val 1057 | O - N | 3.2 | 145 | |
| Asn 1020 - Tyr 1024 | N - O | 2.8 | 142.8 | |
| Asn 1020 - Tyr 1024 | O - N | 2.4 | 111.7 |
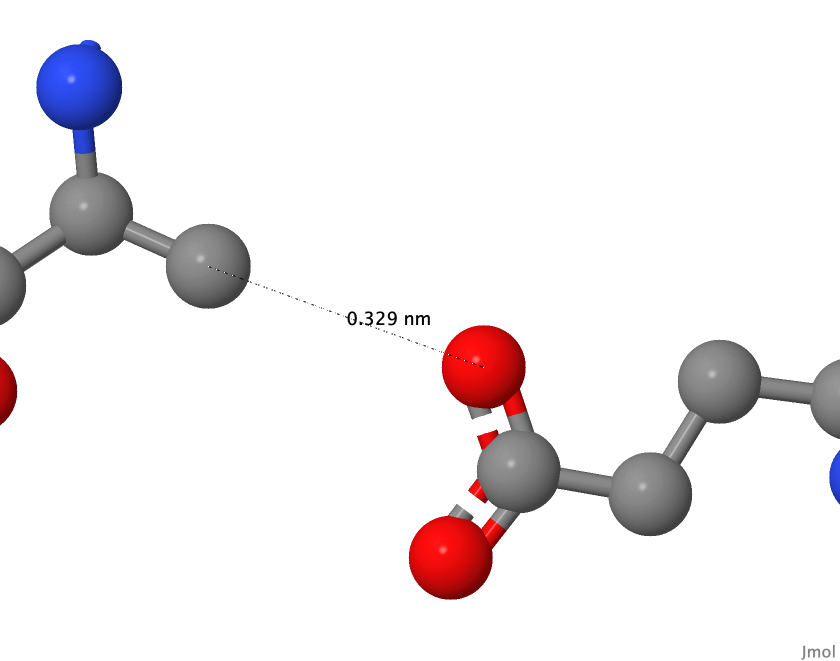
| Остатки, формирующие мостик | Расстояние, Å | |
| 1 | Glu 341 - Lys 344 | 3.5 и 2.9 |
| 2 | Glu 229 - Lys 233 | 3.2 |
| 3 | Glu 1128 - Arg 1125 | 3.5 и 2,8 |
| 4 | Glu 270/1 - Arg 263/2 | <3.3 |
В качестве демонстрационного аминокислотного остатка был выбран фенилаланин Phe 1153.
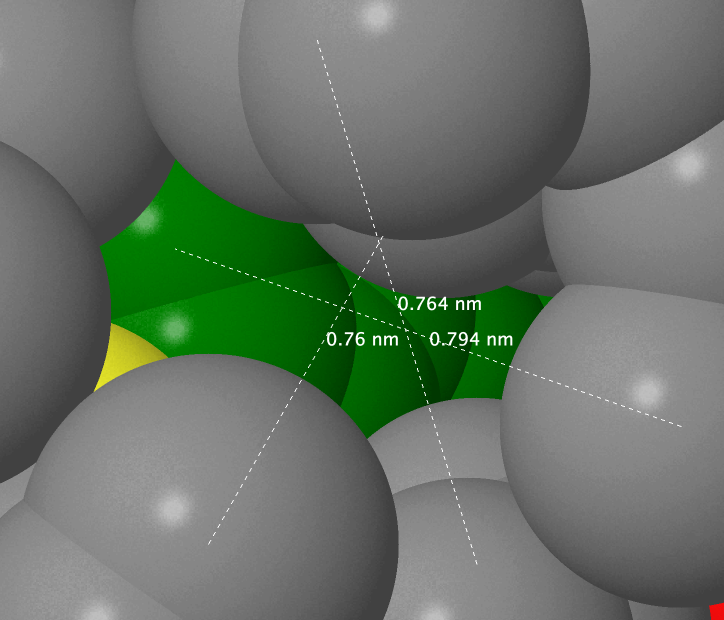
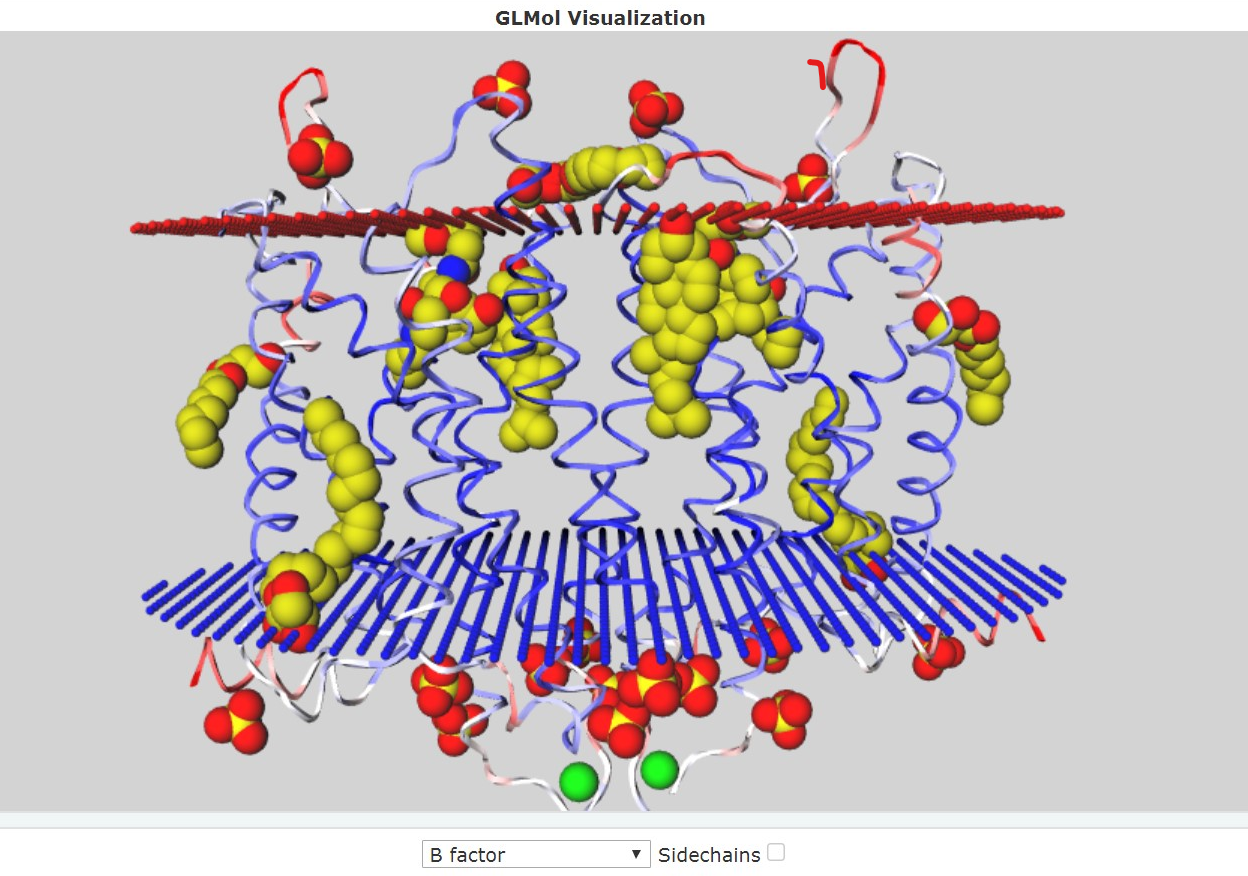
Прогнав скрипт в третьем апплете, можно увидеть, что расстояние в 7 ангстрем является минимальным для полного сокрытия аминокислоты в других атомах. В среднем, характерное расстояние между ядрами соседних не связанных ковалентно атомами в белке не больше, чем 6.1 ангстрем, т.е. в такое расстояние не поместится молекула воды, поскольку расстояния между поверхностями атомов не больше 2.2Å, а диаметр атома кислорода составляет 2.8Å[16]. Однако, вблизи атома серы метионина Met 1102 существует просвет(Рис.3), который ничто не занимает и который вполне может вместить одну молекулу воды.
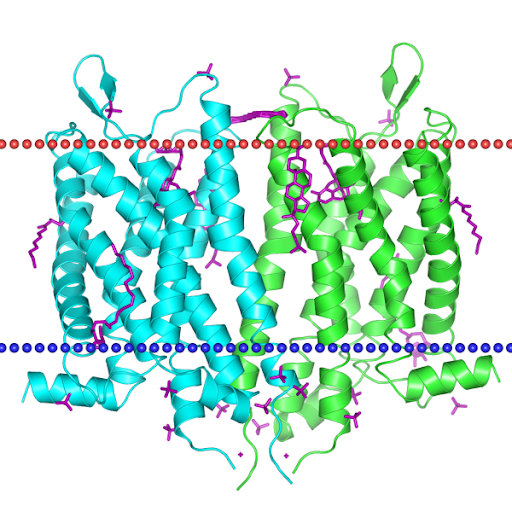
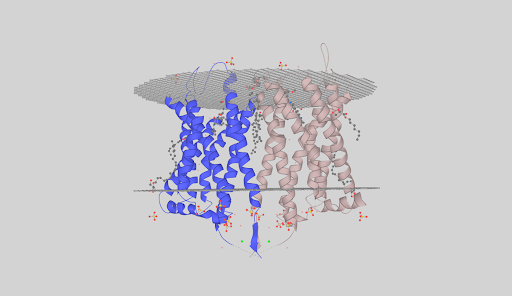
Белок относится к семейству рецепторов, связанных с G-белком. Согласно предсказанию TMHMM(Рис. 4), у него 7 трансмембранных участков в виде альфа-спиралей, находящихся N-концом наружу. Расположение белка по аминокислотам можно увидеть в Таблице 4. Видно, что у белка есть внутренние, внешние и трансмембранные домены. Очевидно, что внутриклеточные домены нужны рецепторы для связи с G-белками. Также, участок белка с 206 по 384 аминокислоту является самым большим по протяженности из внутриклеточных, а значит, скорее всего является участком связи с G-белками. Энергия ассоциации всех трансмембранных альфа-спиралей (т.е. энергетических выигрыш процесса их ассоциирования) −65 kcal\mol, что является энергетически выгодным. Поэтому белок компактно располагается в мембране.
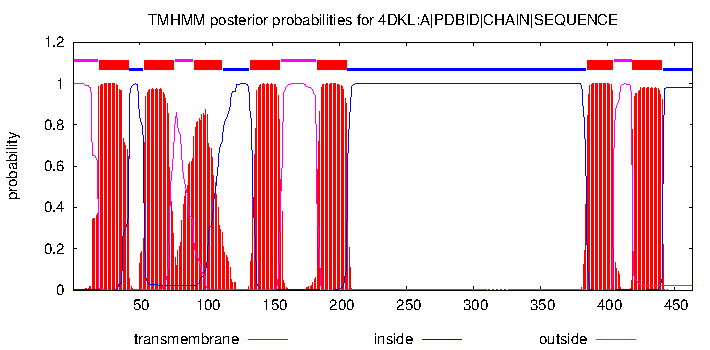
| Вне клетки | 1- 19 |
77- 90 |
156- 182 |
405- 418 |
|||||||||||
| Трансмембиранное пространство |
20- 42 |
54- 76 |
91- 112 |
135- 155 |
183- 205 |
385- 404 |
419- 441 |
||||||||
| Внутри клетки | 43- 53 |
113- 132 |
206- 384 |
442- 464 |
Дмитрий Босов: скрипты апплета (лиганды), смог сформулировать неоценимый вклад Евгения Егорова. Перевод на язык эмодзи.
Евгений Егоров: большая часть работы выполнена тренером нашего состава по киберспортивной дисциплине Dota 2. Сложно выделить какие-то ключевые моменты в его деятельности, так как любой его совет направлял нас в верное русло. Мы долго не могли понять как двигаться по карте. Именно его качественный анализ — просмотр всех наших соло матчей (со случайными игроками в обычном игровом режиме) и также наши командные игры. Он дал нам множество советов, касательно этого аспекта, к ним относятся:
– расстановка линий: именно он расставил роли. Объяснил нам, что керри должен идти на легкую линию, а не в лес.
– объяснил саппортам, особенно 4-ке — Артемию (далее Шейкеру) о особенностях перемещения по карте в первые минуты игры.
– показал нам на важность хождения впятером после стадии лайнинга (от англ. line — линия).
Он разбирал с нами не только макро, но и наши микро. Уверенно подмечал все ошибки. Мы были уверены, что эти ошибки свойственны даже тир-1 игрокам, но на это заявление он просто показал нам EvilArthas, величайшего игрока в Dota 2.
Это самый лучший игрок, идеально чувствующий игровую ситуацию. Он способен выиграть игру в соло (от англ. solo — в одиночку). Помимо весьма качественного анализа игровой ситуации, он никогда не принижает свой вклад в игру. Именно он доказал существование «скрытого пула» на своём примере.
Артас (EvilArthas) смотря профессиональные матчи (The International) отметил, что в игре важны 3 аспекта: первый и самый важный — пик (от англ. pick — выбирать), второй — командное взаимодействие, и третье — личный скилл игрока(умение вовремя нажать БКБ и как говорил сам EvilArthas: «БКБ купил, прожать не купил»).
Непросто так была упомянута стадия пиков — именно над ней Евгений Егоров работал большую часть времени. У нас есть множество связок героев, благодаря его советам по выбору персонажей мы уверенно отстаиваем линии.
Самые важные советы Тренера:
– Не пикать Pudge
– Банить Techies и Pudge
– Пикать Rubick саппорта в 2045 — быть дауном (в этом также уверен EvilArthas)
– Legion Commander — худший герой (Лучшая внешне, худшая на деле)
– Shadow Fiend проигрывает мид уже как 10 лет
Помимо определения слабых героев Тренер также выделяет Wraith King, Luna, Windranger.
Сложно даже предположить, где бы мы были, если бы нас не согласился тренировать Евгений Егоров. Мои споры с саппортом Владимиром Н. периодически доходили до драк, но благодаря Тренеру это прекратились. Когда он пришёл к нам, мы были на гране распада: вечный скрытый пул, не понимая в команде, плохая отыгровка от Phantom Lancer; но он, в соляного, сумел нас настроить, раскрыть скрытый потенциал каждого из нас. Он был не просто Тренером — он был психологом.
Нам не удалось попасть на первые квалификации Major из-за контрольной по химии, хотя безусловно, мы были топ-1 СНГ. Помимо химии, мы прекрасно понимали, что если бы мы поехали в Чэнду (Китай), то пропустили бы коллоквиум по математическому анализу (хорошо что мы учли это и сделали выбор в пользу Na’Vi (дали им шанс пройти)).
Помнится, он рассказал мне, как правильно стоять хардлайн на Windranger. Я понял, что до этого допускал множество ошибок и не представлял что нужно делать. Он указал Артемию П. как играть на Earthshaker. После его гайда я не удивлюсь, что Артемий даст свой Echo Slam на миллион долларов ( это отсылка к The International 2016, когда EG — североамериканская команда — в решающей драке на рошане выдали свою связку: Echo Slam + Ice Blast, убив всю команду противника).
Логичный вопрос: как всё это относится к Атласу контактов?
Знаете, всё рассказанное выше никак не относится к Атласу контактов, Евгений Егоров просто хороший тренер. На самом деле если бы не его опыт, мы бы вряд ли организовались и так слаженно сделали эту работу.
Владимир Ноздрин: поиск межбелковых взаимодействий, скрипты апплетов (межбелковые взаимодействия, гидрофобное ядро), сопровождение отчета скриншотами из JMol, создание веб-страницы с отчётом.
Артемий Пигиданов: работа с TMHMM, получение изображения белка в мембране с помощью соответствующих сервисов, выводы о функциональной принадлежности найденных межбелковых контактов.
Даниил Хлебников: текст отчёта (Ru + En), описание полученных результатов, поиск водородных связей.