Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. Построение дерева по нуклеотидным последовательностям
| Название | Мнемоника | AC записи EMBL | Координаты 16S rRNA | Цепь |
| Bacillus subtilis | BACSU | AL009126 | 9810..11364 | + |
| Enterococcus faecalis | ENTFA | AF039902 | 1..1494 | + |
| Finegoldia magna | FINM2 | AP008971 | 197837..199361 | + |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 | + |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160..46720 | + |
| Listeria monocytogenes | LISMO | FM242711 | 234080..235590 | + |
| Staphylococcus aureus | STAA1 | CP000253 | 448819..450374 | + |
| Streptococcus pyogenes | STRP1 | AM295007 | 17043..18549 | + |
Последовательности были воровнены с помощью Muscle.
файл jar
Выравнивание было импортировано в MEGA, дерево реконструировано методом Neighbour-Joining.
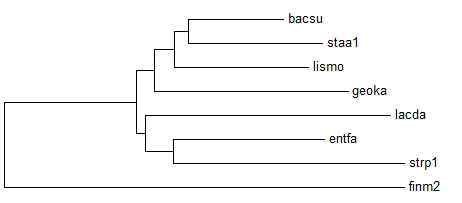
Изображение дерева:
Правильное дерево:
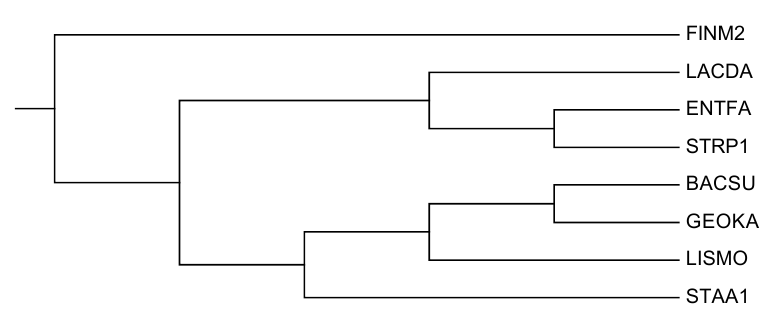
Построенное дерево с правильным не совпадает. Отсутствуют ветви {BACSU, GEOKA} vs {FINM2, LACDA, ENTFA, STRP1, LISMO, STAA1} и {BACSU, GEOKA, LISMO} vs {FINM2, LACDA, ENTFA, STRP1, STAA1}, и появились 2 неверные новые: {BACSU, STAA1} vs {FINM2, LACDA, ENTFA, STRP1, LISMO, GEOKA} и {BACSU, STAA1, LISMO} vs {FINM2, LACDA, ENTFA, STRP1, GEOKA}. Качество реконструкции однозначно сравнить сложно, и в задании 5 второго практикума результаты получались не ближе к правильным.
2. Построение и анализ дерева, содержащего паралоги
Найдем в своих бактериях достоверные гомологи белка CLPX_BACSU и построим дерево этих гомологов.
Сначала воспользуемся командой:
makeblastdb -in proteo.fasta -out proteins -dbtype prot
Теперь проводим поиск гомологов белка из CLPX.fasta по базе proteins:
blastp -query CLPX.fasta -db proteins -out homologs.txt -evalue 0.001
Из полученного набора белков отбираю только относящиеся к моим бактериям белки, извлекаю из соответствующих баз данных их последовательности в отдельлный файл. В Jalview строим выравнивание программой Muscle, импортируем его в MEGA, методом Neighbour-Joining строим дерево:
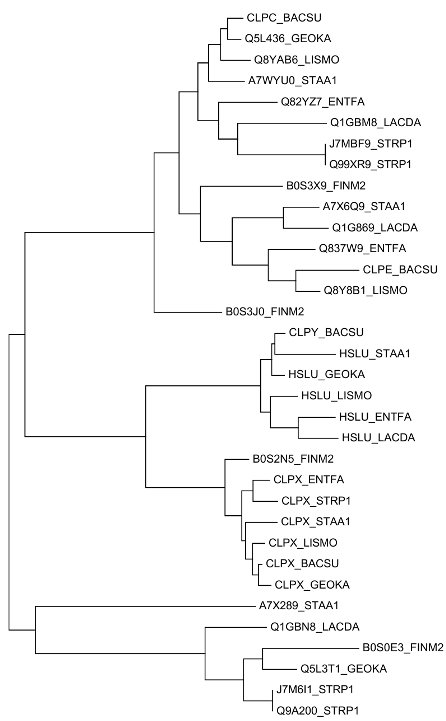
Будем считать дерево реконструированным верно.
Два гомологичных белка будем называть ортологами, если они а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами.
Находим несколько ортологов: HSLU_ENTFA и HSLU_LACDA, CLPX_ENTFA и CLPX_STRP1, CLPX_BACSU и CLPX_GEOKA.
Есть и паралоги, например: J7MBF9_STRP1 и Q99XR9_STRP1, CLPX_BACSU и CLPY_BACSU, B0S3X9_FINM2 и B0S3J0_FINM2.