Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
Выравнивание представителей домена, построенное в практикуме 9, было разделено на две группы по таксономии. Такое решение приняла после анализа деревьев, на которых хорошо отделились таксоны, и нет явного разделения всего дерева по архитектурам белков. Интересно также будет посмотреть, как качество быборки отразится на результатах.
Построение профиля
Использовались выравнивания: 1.msf (Ecdysozoa) и 2.msf (Chordata).
seqret 1.msf msf::1_1.msf
noreturn -infile 1_1.msf -outfile 1_2.msf
pfw 1_2.msf > 1_weighted.msf
pfmake 1_weighted.msf /usr/share/pftools23/blosum62.cmp > 1.prf
То же самое было проделано для выравнивания 2.msf.
Поиск по профилю в исходных последовательностях
Был создан файл со всеми последовательностями в формате fasta. Затем проведен поиск по профилю в исходных последовательностях. Порог веса поставим маленьким (-C 0.0), чтобы все последовательности оказались в выдаче:
pfsearch –C 0.0 –f 1.prf selected.fa | sort -nr > 1.xls
pfsearch –C 0.0 –f 2.prf selected.fa | sort -nr > 2.xls
Построение ROC-кривой по полученным данным и анализ результатов поиска
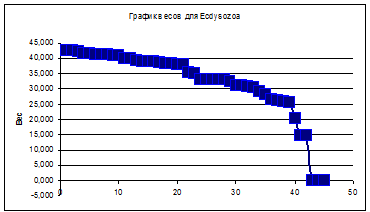
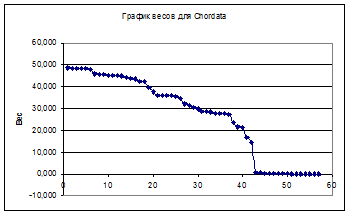
Отметим в таблицах последовательности, которые действительно принадлежат профилям. Дубликаты удалены. Построим графики весов находок pfsearch, отсортированных по убыванию:
ROC-кривые получились довольно странные:
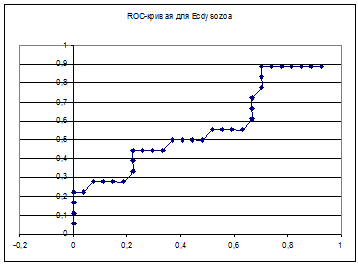
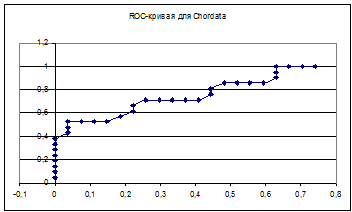
По графикам для нормализованного веса все-таки можно установить порог - 25 для Ecdysozoa и 27 для Chordata. Ошибки первого и второго рода для обоих подтаксонов оказались довольно значительными. Для Chordata:
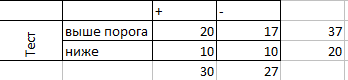
Для Ecdysozoa:
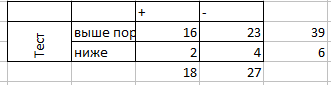
Можно сделать вывод, что такое разбиение белков оказалось неоптимальным. Повлиял и маленький размер выборки, и неравноценное представительство подтаксонов (Ecdysozoa изначально брали меньше). И, думаю, можно заключить, что по таким профилям различать последовательности Линяющих и Хордовых совсем не следует.