Учебный сайт Фоменко Елены
| Главная | Семестры | Проекты | Заметки |
1. A- и В- формы ДНК. Структура РНК
Нужно построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
Пакет 3DNA - один из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот
(работает под операционной системой LINUX).
С помощью Рutty, используя ssh, подсоединяемся к kodomo.cmm.msu.ru. В директории Term3/Practice2 вводим следующие команды, чтобы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
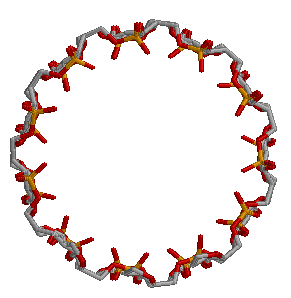
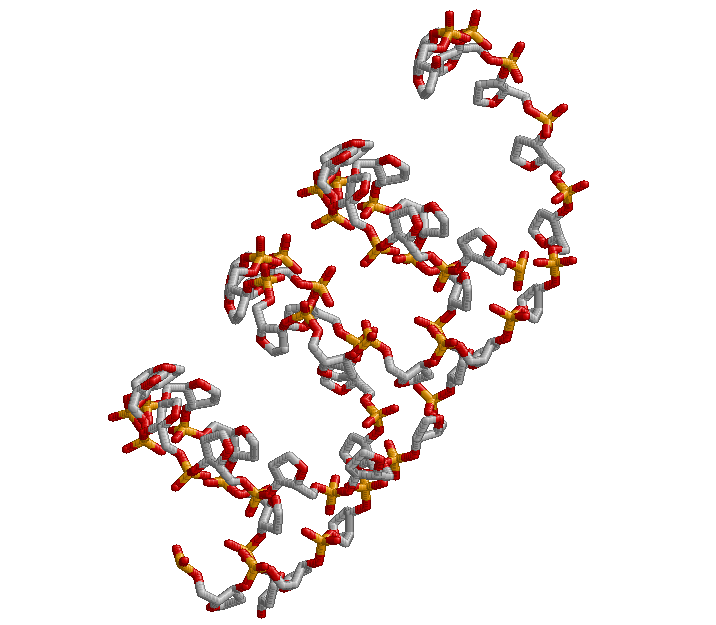
С помощью программы fiber пакета 3DNA построила A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc". (Для z-формы повторяющейся единицей выбрана последовательность "gc".) Для начала работы с программой fiber запустила команду:
fiber -h
Полученные файлы:
gatc-a.pdb
gatc-b.pdb
gatc-z.pdb
Были использованы команды:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Repeating unit: gatc
Number of repeats: 10
2. Средства RasMol для работы со структурами нуклеиновых кислот.
Упр.1.
Нужно научиться выделять разные атомы и химические группировки, используя предопределенные множества RasMol.
Выделены сахарофосфатный остов ДНК, нуклеотиды (аденин-красным, гуанин-зеленым, цитозин-розовым,
тимин-синим), аденины, атом №7 во всех гуанинах.
Вот скрипт.
А-форма.
Сахарофосфатный остов:
Выделены нуклеотиды:
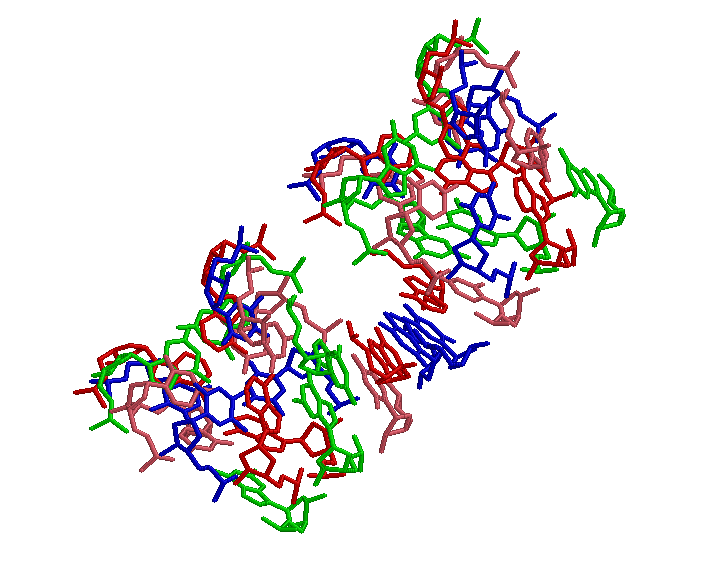
Выделены аденины:
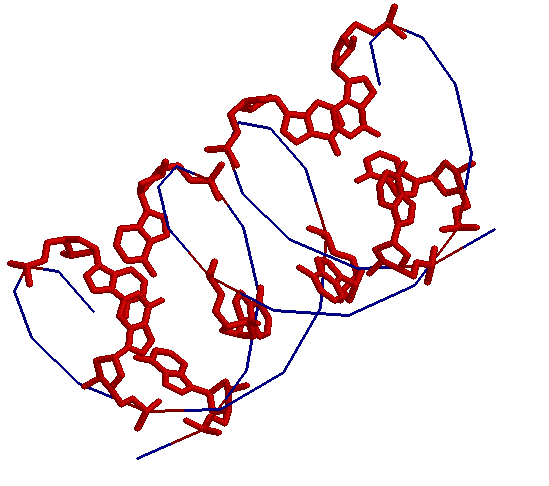
Выделены гуанины и атомы №7 в них:
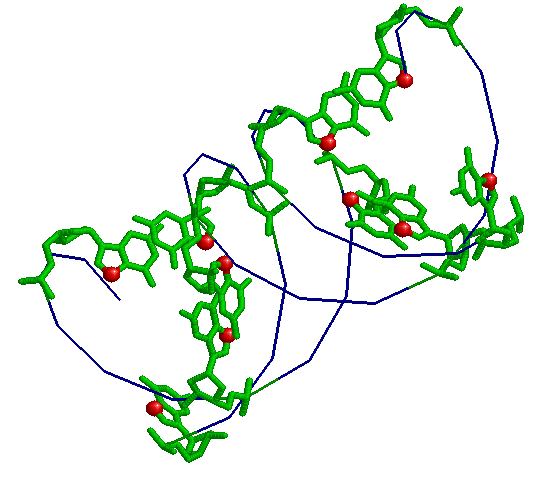
B-форма.
Сахарофосфатный остов:

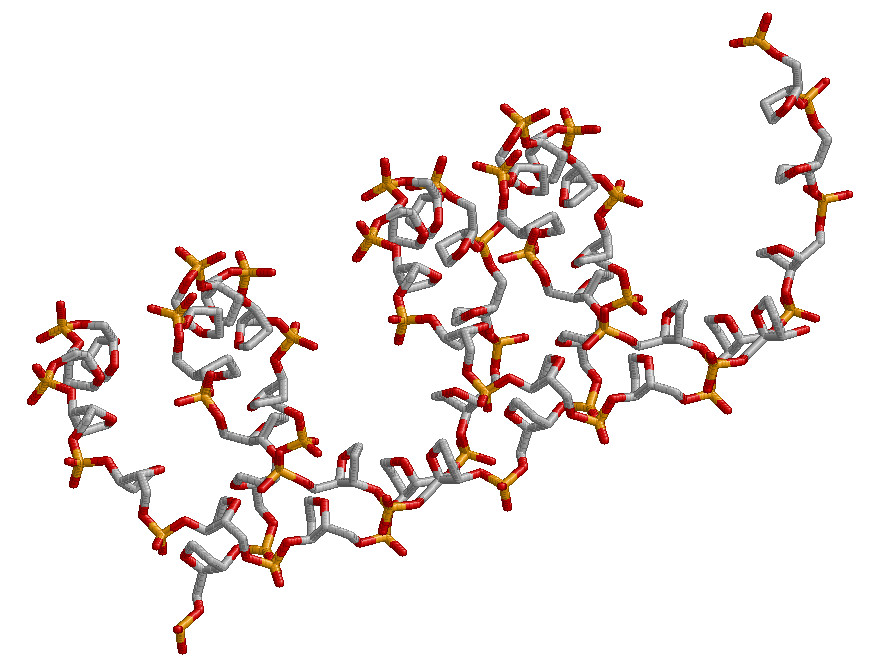
Выделены нуклеотиды:
Выделены аденины:
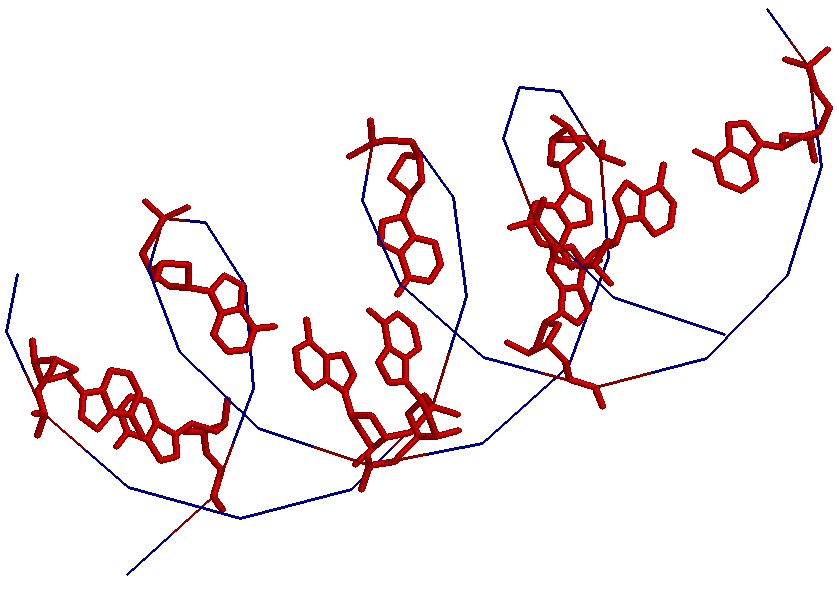
Выделены гуанины и атомы №7 в них:
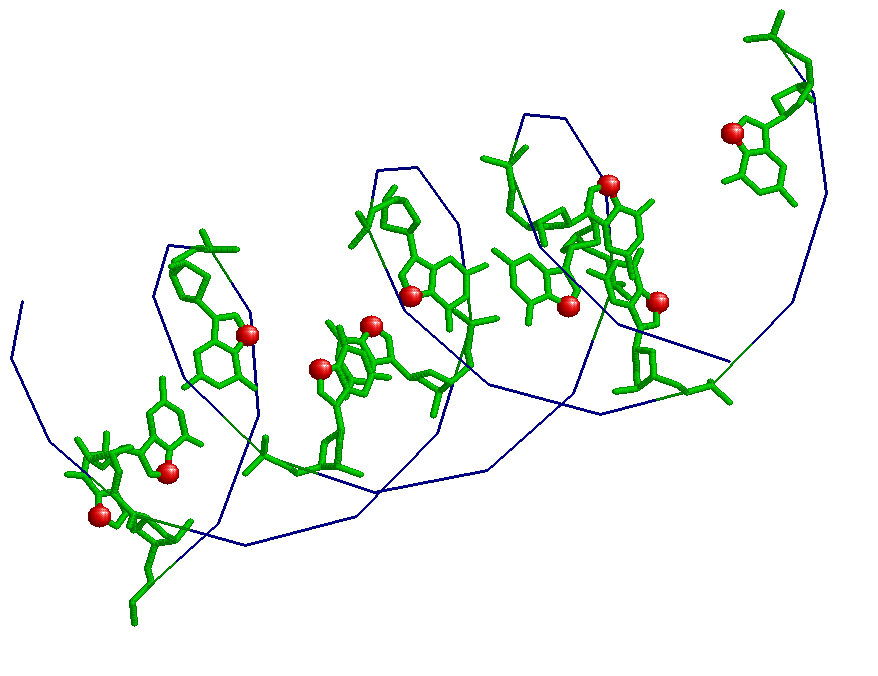
Z-форма.
Сахарофосфатный остов:
Выделены нуклеотиды:
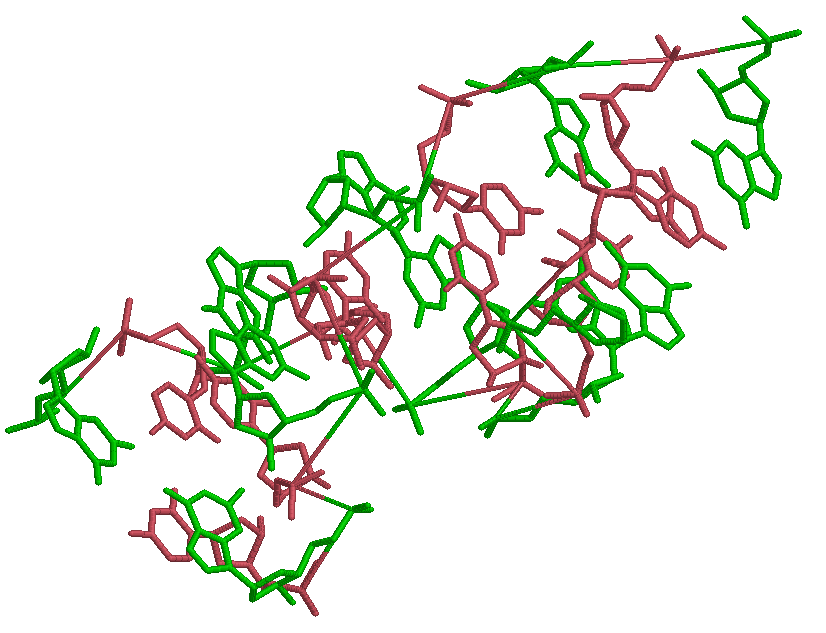
Аденинов тут нет и быть не может:
Выделены гуанины и атомы №7 в них:
Упр. 2
На сайте PDB нашла структуры 1qrt и 1ksx. Сохранила изображения структур.
1qrt (Glutaminyl-tRNA synthetase mutant D235G complexed with glutamine transfer RNA):
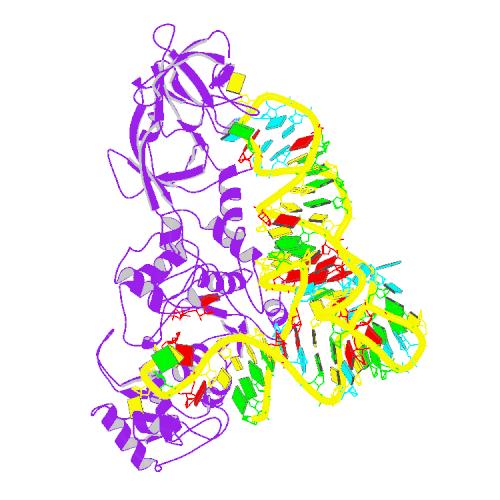
1ksx (Assembly of the Papillomavirus Replication Initiation Complex)
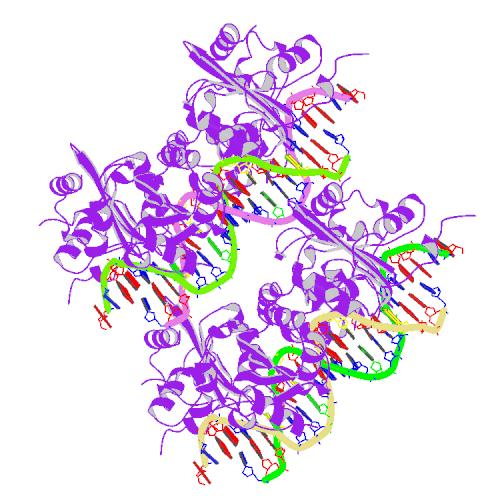
Сохранила в рабочей директории файлы pdb: 1QRT.pdb, 1KSX.pdb.
Упр.3
Нужно проверить ДНК и РНК на наличие разрывов. Получаем изображения только нуклеиновых кислот в проволочной модели и смотрим.
1qrt:
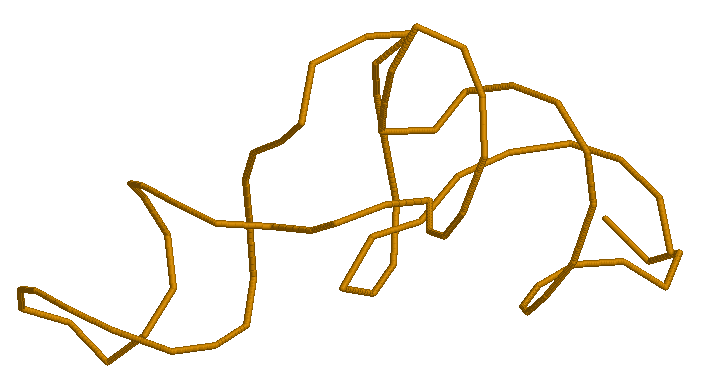
1ksx:

Разрывов не обнаружено!
Чтобы сохранить координаты атомов только ДНК и РНК, получив изображения, представленные выше, используем команду save <имя файла.pdb>. Получаем: 1QRTN.pdb и 1KSXN.pdb.
3. Сравнение структур 3-х форм ДНК с помощью средств RasMol
Упр.1
Нужно научиться находить большие и малые бороздки. Открываем в RasMol файл gatc-b.pdb, полученный при выполнении задания 1. Рассматриваем структуру, определяем большие и малые бороздки. Выбираем цитозин №12, определяем направление его атомов. На изображении: атомы, обращенные в сторону большой бороздки, окрашены красным, обращенные в сторону малой бороздки - синим.
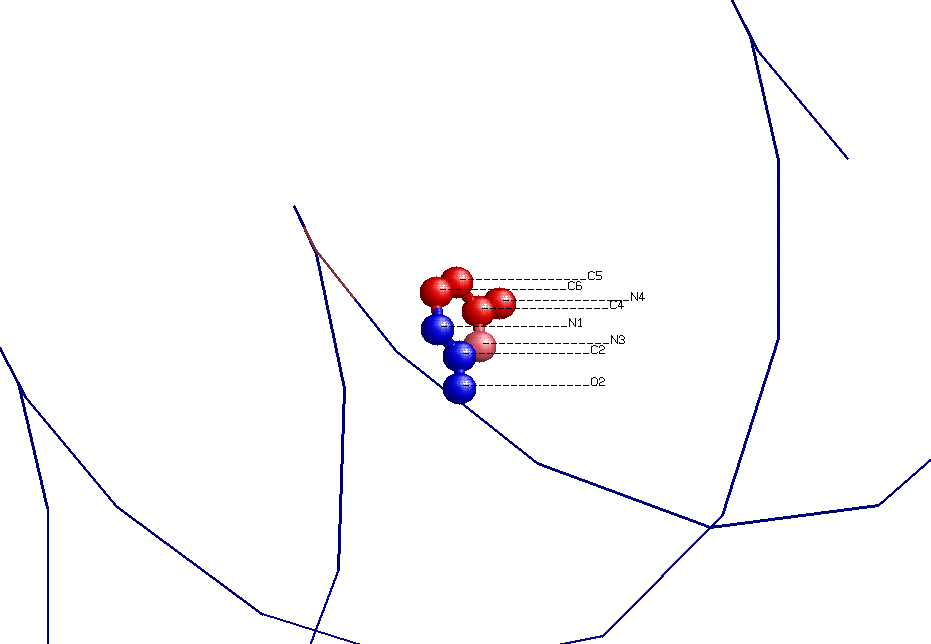
С помощью ChemSketch получаем изображение основания:
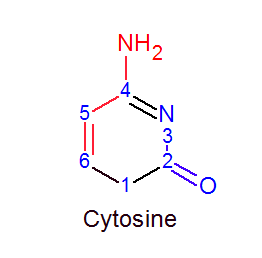
В сторону большой бороздки обращены атомы с12.c5, c12.c6, c12.с4, c12.n4
В сторону малой бороздки обращены атомы c12.c2, c12.n1, c12.o2
Остальные атомы основания не имеют четкого направления.
Посмотрим, как направлены те же атомы в A- и Z-структурах.
| направление | A | Z |
| в сторону малой бороздки | c12.c5, c12.c4, c12.n4 | c6.n1, c6.c2, c6.o2 |
| в сторону большой бороздки | c12.n1, c12.c2, c12.o2, c12.n3 | c6.c5, c6.c4, c6.n4 |
| не имеют четкого направления | c12.c6 | c6.c6, c6.n3 |
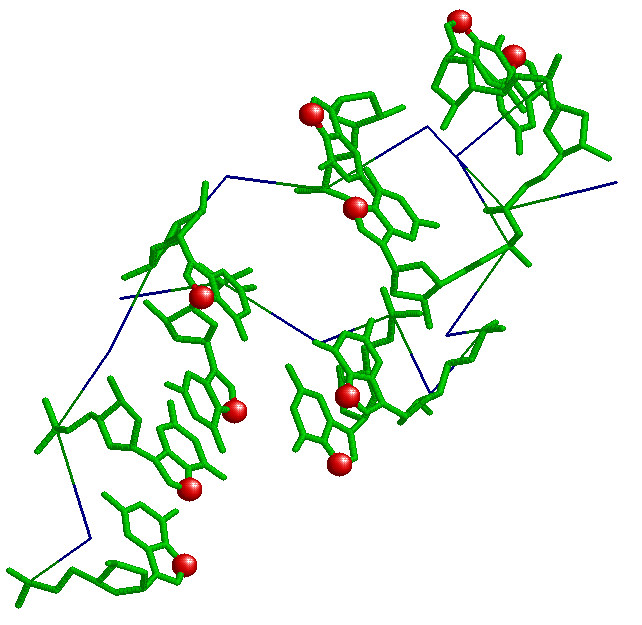
Упр.2
Сравниваем основные спиральные параметры разных форм ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (A) | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
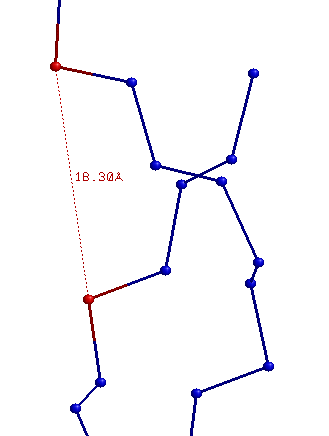
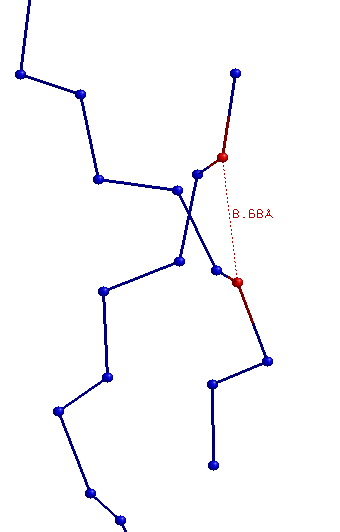
| Ширина большой бороздки | 16,81 (T35B.P - C8A.P) | 17,91 (C28B.P - G9A.P) | 18,30 (C6A.P - C12B.P) |
| Ширина малой бороздки | 7,98 (A10A.P - G25B.P) | 11,69 (G13A.P - C32B.P) | 8,68 (G17B.P - G9A.P) |
Изображения измерений:
A-форма:
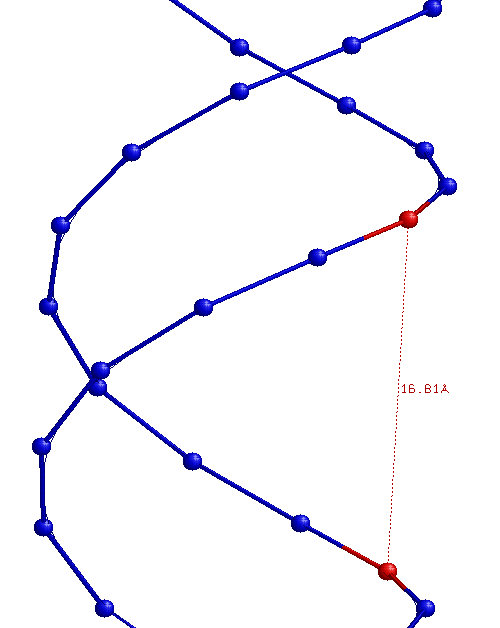
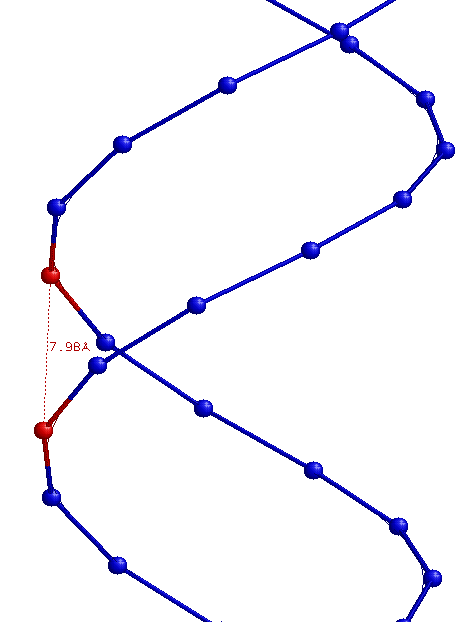
B-форма:
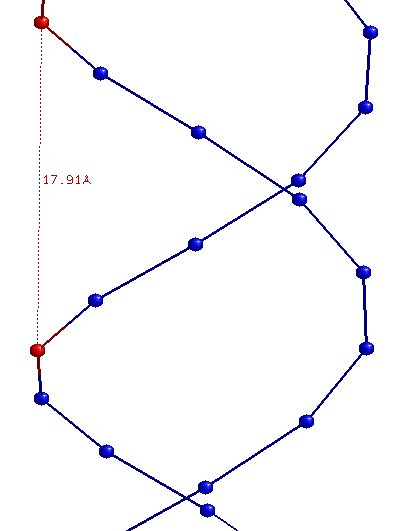
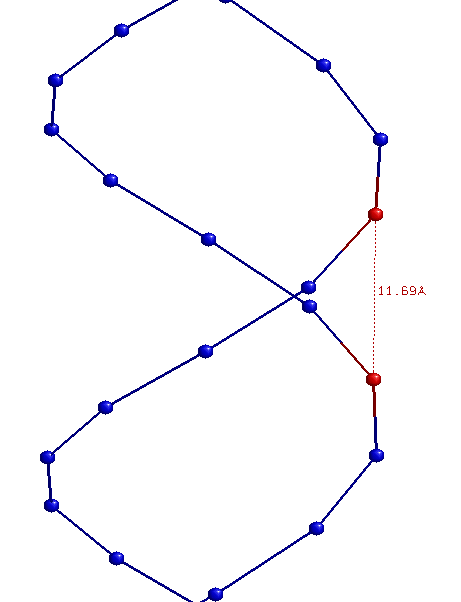
Z-форма:
Упр.3
Нужно cравнить торсионные углы в структурах А- и В-форм.
Таблица по результатам измерений:
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) | |
| A-форма | -51.67 | 174.79 | 41.73 | 79.11 | -147.75 | -75.15 | -157.18 |
| B-форма | -29.89 | 136.32 | 31.17 | 143.34 | -140.77 | -160.50 | -97.97 |
Значения торсионных углов из презентации:
| A-форма | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-форма | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Упр.1
Нужно научиться определять торсионные углы нуклеотидов.
Для анализа структур нуклеиновых кислот будем использовать программы find_pair и analyze. Выполняем команды:
find_pair -t gatc-a.pdb stdout | analyze
find_pair -t gatc-b.pdb stdout | analyze
find_pair -t gatc-x.pdb stdout | analyze
В рабочей директории получаем файлы с описанием разных параметров структуры, в файле gatc-x.out можно найти описание водородных связей, значения всех торсионных углов, ширину малой и большой бороздки.
В этом файле приведены данные о торсионных углах в каждой из структур. В наибольшей степени отличаются значения углов delta, zeta и chi.
Поскольку пакет 3DNA пока работает только со старым форматом PDB, для перевода файлов в старый формат используем программу remediator, установленную на kodomo:
remediator --old ''1QRT.pdb'' > ''1qrt_old.pdb
remediator --old ''1KSX.pdb'' > ''1ksx_old.pdb
Далее воспользуемся программами find_pair и analyze. Значения торсионных углов в заданной структуре тРНК - в отдельноом файле. С помощью Excel определяем среднее значение каждого из торсионных углов ДНК (краевые нуклеотиды не рассматриваем), номер самого "деформированного" нуклеотида. Результаты - в таблице Excel.
Упр.2
Учимся определять структуру водородных связей.
Определяем номера нуклеотидов, образующих стебли во вторичной структуре тРНК.
Акцепторный стебель образован комплементарными участками 2-7 и 71-66.
T-стебель образован участками 49-53 и 65-61.
Антикодоновый стебель - участками 37-44 и 33-26.
D-стебель - участками 10-12 и 25-23.
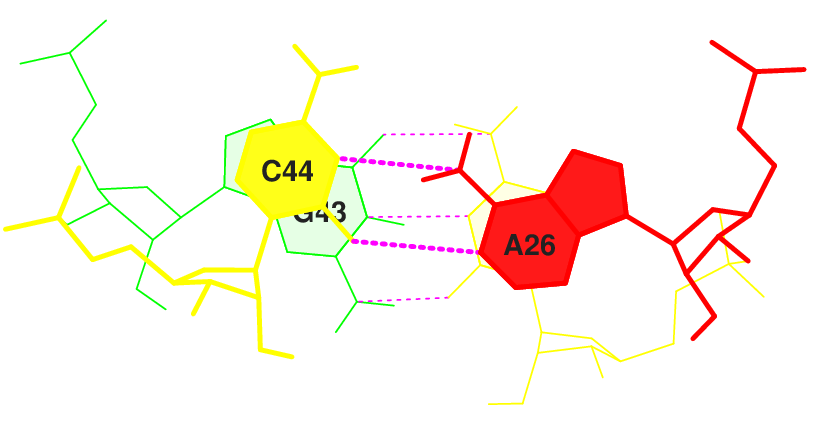
Найдены дополнительные водородные связи, стабилизирующие ее третичную структуру:
B:..38_:[..U]U-*---U[..U]:..32_:B B:..44_:[..C]Cx*---A[..A]:..26_:B B:..13_:[..A]A-**+xA[..A]:..45_:B B:..46_:[..U]U-**+-U[..U]:..47_:B B:..19_:[..G]Gx---xC[..C]:..56_:B B:..14_:[..A]A-*--xU[..U]:...8_:B B:..15_:[..G]Gx**+xC[..C]:..48_:B B:..19_:[..G]Gx---xC[..C]:..56_:B B:..54_:[..U]U-*--xA[..A]:..58_:B B:..55_:[..U]Ux**+xG[..G]:..18_:B
Из них 5 - неканонические (2 U-U, C-A, A-A, U-G).
Упр.3
Учимся находить возможные стекинг-взаимодействия.
В файле 1qrt_old.out находим таблицу с информацией о перекрывании последовательных пар оснований:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GG/CC 4.24( 2.95) 0.00( 0.00) 0.19( 0.00) 0.39( 0.00) 4.82( 2.95) 2 GG/CC 3.73( 2.42) 0.00( 0.00) 0.44( 0.00) 0.15( 0.00) 4.32( 2.42) 3 GG/CC 3.92( 2.54) 0.00( 0.00) 0.41( 0.00) 0.00( 0.00) 4.32( 2.54) 4 GU/AC 6.54( 3.63) 0.00( 0.00) 0.00( 0.00) 4.14( 2.56) 10.67( 6.19) 5 UA/UA 0.99( 0.09) 0.00( 0.00) 1.29( 1.04) 0.16( 0.00) 2.43( 1.13) 6 AC/GU 1.82( 0.78) 0.00( 0.00) 0.00( 0.00) 6.19( 3.15) 8.01( 3.93) 7 CG/CG 0.00( 0.00) 0.00( 0.00) 6.16( 3.35) 0.00( 0.00) 6.16( 3.35) 8 GA/UC 4.25( 2.45) 0.00( 0.00) 0.00( 0.00) 3.19( 0.93) 7.44( 3.38) 9 AG/CU 3.91( 3.53) 0.00( 0.00) 0.14( 0.00) 0.01( 0.00) 4.07( 3.53) 10 GG/CC 2.66( 1.16) 0.00( 0.00) 1.92( 0.11) 0.00( 0.00) 4.58( 1.27) 11 GU/AC 6.35( 3.70) 0.00( 0.00) 0.00( 0.00) 5.74( 2.68) 12.09( 6.38) 12 UU/GA 3.81( 1.83) 0.00( 0.00) 0.00( 0.00) 4.48( 2.48) 8.29( 4.31) 13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 14 AU/UU 4.76( 1.35) 0.00( 0.00) 0.00( 0.00) 5.85( 3.17) 10.62( 4.52) 15 UU/AU 2.07( 0.74) 0.00( 0.00) 0.00( 0.00) 5.12( 3.41) 7.19( 4.15) 16 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.71( 1.19) 2.71( 1.19) 17 CC/GG 0.01( 0.00) 0.00( 0.00) 1.39( 0.00) 3.13( 1.62) 4.53( 1.62) 18 CG/CG 0.59( 0.00) 0.00( 0.00) 4.11( 1.37) 0.40( 0.00) 5.10( 1.37) 19 GG/CC 1.95( 0.57) 0.00( 0.00) 0.93( 0.00) 0.16( 0.00) 3.04( 0.57) 20 GC/AC 6.70( 3.73) 0.00( 0.00) 0.00( 0.00) 5.56( 3.63) 12.26( 7.36) 21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.15( 0.00) 1.40( 0.80) 1.55( 0.80) 22 GC/GC 4.71( 1.81) 0.00( 0.00) 0.00( 0.00) 6.10( 3.04) 10.81( 4.85) 23 CC/GG 0.00( 0.00) 0.00( 0.00) 0.76( 0.00) 2.89( 1.35) 3.64( 1.35) 24 CA/AG 2.40( 0.85) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.40( 0.85) 25 AA/UA 0.00( 0.00) 1.71( 0.00) 0.00( 0.00) 0.00( 0.00) 1.71( 0.00) 26 AG/CU 4.24( 1.52) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.24( 1.52) 27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 28 GU/UC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Выбираем пару с наибольшим значением площади перекрывания:
20 GC/AC 6.70( 3.73) 0.00( 0.00) 0.00( 0.00) 5.56( 3.63) 12.26( 7.36)
Далее в файле stacking.pdb находим номер этой структуры и вырезаем ее командой
ex_str -20 stacking.pdb step20.pdb
Получаем изображение командой
stack2img -cdolt step20.pdb step20.ps