Эволюция белков митохондриальных рибосом
Одной из теорий возникновения митохондрий является теория симбиогенеза. Целью данного блока являлось исследование возможности происхождения митохондрий от альфапротеобактерий или от гаммапротеобактерий. В работе я рассматривала белок L14 большой субъединицы митохондриальной рибосомы. Последовательность белка по различным критериям сравнивалась с последовательностями рибосомальных белков альфа- и гаммапротеобактерий, на основании чего делались некоторые выводы.- Поиск бактериальных гомологов среди альфа- и гаммапротеобактерий по нормированному профилю белков L14 митохондриальных рибосом
- Филогенетический анализ рибосомальных белков L14 из эукариотических миохондрий и 3-х групп бактерий Анализировались последовательности восьми исследуемых рибосомальных белков из эукариотических митохондрий, последовательности альфа- и гаммапротеобактерий, найденные по профилю, а кроме того - последовательности из созданной out-группы - последовательности рибосомальных белков из разных родов Firmicutes.
- Резюме В ходе работы по различным критериям сравнивались последовательности митохондриального рибосомального белка эукариот L14 с последовательностями рибосомальных белков альфа- и гаммапротеобактерий. В целом, результаты различных этапов работы вполне согласуются между собой. Распределение нормированных весов находок в протеомах двух групп бактерий и тест Вилкоксона показали, что профилю, построенному по митохондриальным белкам, лучше удовлетворяют некоторые гомологичные белки из альфа-, а не гаммапротеобактерий. В свою очередь, филогенетический анализ показал эволюционную близость белка L14 митохондрий белкам альфапротеобактерий. В частности, филогенетическое дерево выявило, что наиболее близкими альфапротеобактериями к рассматриваемым эукариотическим белкам являются Rickettsiaceae. Поэтому можно сделать вывод о том, что происхождение митохондрий эукариот от альфапротеобактерий более вероятно, чем от гаммапротеобактерий.
Выбор последовательностей для исследования
Для начала нужно было создать выборку ортологичных белков из рибосом митохондрий эукариот. С помощью SRS в UniProt были найдены последовательности рибосомального белка L14, принадлежащего восьми разным организмам.Для этого в поисковой системе SRS по базе данных UniProtKB производился поиск по следующему запросу:
Description Ribosomal Protein L14 Description 39S Taxonomy Eukaryota
Система выдала 8 находок, все последовательности которых были сохранены в формате FASTA. Отдельно можно просмотреть их коды доступа и идентификаторы.
Найденные ортологичные аминокислотные последовательности имеют приблизительно равную длину - около 145 аминокислотных остатков. Единственной находкой, слегка выбивающейся из стройного ряда, является белок B0WL56_CULQU из Southern house mosquito, длиной в 157 АК. Такое различие в длине нельзя считать значительным, тем более что этот белок достаточно хорошо выравнивается с другими, как будет показано ниже.
В каждой из записей банка UniProt, посвященной находкам, имеется ссылка на базу Pfam, причем на один и тот же домен PF00238.
Доменная структура белка из митохондрий человека
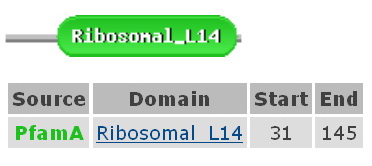
Белок L14 человека состоит из 145 аминокислотных остатков, остатки 31-145 составляют домен Ribosomal_L14.
Выравнивание Pfam
В описании каждой из 8 найденных последовательностей имеется ссылка на домен PF00238. Однако в выравнивании, которое генерирует Pfam, присутствуют только 5 последовательностей. Их выравнивание можно просмотреть в Fasta-формате, либо в виде, выдаваемом GeneDoc. Также можно скачать файл GeneDoc.Выравнивание полных белковых последовательностей
Все 8 найденных последовательностей белка L14 были выровнены программой muscle на kodomo-count. В выравнивание добавлялись веса с помощью pwf из пакета pftools. Получилось следующее выравнивание: в Fasta-формате или раскрашенное; файл GeneDoc.Важно отметить, что выравнивание полных белковых последовательностей практически полностью соответствует выравниванию доменов, сгенерированному Pfam. Не наблюдается сдвигов последовательностей в выравнивании друг относительно друга.
В первую очередь такое хорошее совпадение, пожалуй, объясняется большой схожестью восьми исследуемых последовательностей: в выравниваниях мы наблюдаем значительное число позиций, консервативных на 100%.
Построение профилей
По взвешенному выравниванию восьми белковых последовательностей при помощи pfmake был построен профиль, который затем нормировался относительно случайной базы малого объема программой autoscale. Простой и нормированный профили отличаются значениями SCORE в поле MA (CUT_OFF) и R1/R2 в поле MA (NORMALIZATION), где R1 и R2 - коэффиценты функции нормирования.Поиск гомологов по профилю
C помощью программы pfsearch пакета PFTOOLS производился поиск гомологичных белков среди альфа- и гаммапротеобактерий. Каждый поиск сначала повторили по 3 раза с разными значениями порога 5.0, 10.0, 30.0.С пороговым значением 30 не было найдено ни одного гомолога ни в одной группе. С порогом 10 нашлись 3 гомолога среди альфапротеобактерий и ни одного - среди гаммапротеобактерий. Для порогового значения 5 было обнаружено 218 гомологичных белков альфапротеобактерий и 588 - гаммапротеобактерий.
Такое несогласие количества находок побудило меня провести поиск дополнительно для пороговых значений 6,8 и 9. Получилось, что для пороговых значений 8, 9 и 10 гомологов из альфапротеобактерий больше, чем из гаммапротеобактерий. Для порогов в 5 и 6 результат был противоположным.
Это немного смущало, т.к. не совсем понятно, какие пороговые значения лучше выбрать для дальнейшей работы. В итоге руководствуясь тем, что для порога в 9 уже сравнительно мало находок, я выбрала значения 5, 6 и 8.
Сравнение вариантов поиска
Итак, мы рассматриваем 6 вариантов поиска гомологов - по 3 с различными значениями порога на каждую группу бактерий.Для каждого варианта при помощи скрипта определялось количество находок с GO идентификаторами "клеточный компонент" и "большая субчастица рибосомы".
Все результаты занесены в таблицу:
Результаты поиска по двум группам бактерий с разными значениями порога.
| Группа | Альфапротеобактреии | Гаммапротеобактерии | ||||
|---|---|---|---|---|---|---|
| Порог | 5 | 6 | 8 | 5 | 6 | 8 |
| Общее количество находок | 218 | 90 | 60 | 588 | 168 | 22 |
| С GO "cellular component (С)" | 177 | 86 | 60 | 450 | 157 | 22 |
| С GO "C:large ribosomal subunit" | 88 | 79 | 59 | 139 | 130 | 22 |
По данным таблицы можно заключить, что пороговое значение
5 не обеспечивает достаточной
"чистоты" выборки: при этом значении среди альфапротеобактерий
записей с GO "C:large ribosomal subunit" менее половины от общего числа,
а среди гаммапротеобактерий - менее трети.
При пороге в 6 уже меньшее число найденных гомологов не имеют
необходимого описания GO. А при пороге в 8 это количество стремится к 0.
Таким образом, пороговое значение 8 позволяет
находить гомологичные последовательности, почти все из которых имеют
пометку GO в описании. Поэтому для поиска по профилю было выбрано именно
значение 8.
Распределение нормированных весов находок в протеомах двух групп бактерий
Для сравнения весов гомологичных находок альфа- и гаммапротеобактерий в Excel была построена диаграмма распределения значений весов.Диаграммы распределения
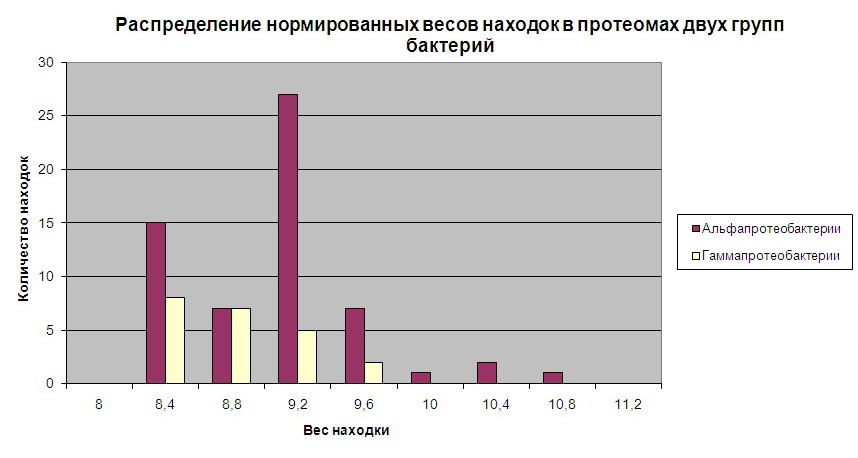
Количество находок среди альфапротеобактерий почти в
3 раза больше, чем среди гаммапротеобактерий, поэтому сравнивать абсолютные
количества находок среди 2 групп бактерий для разных карманов не имеет смысла.
Важно посмотреть на форму распределений. Из-за относительно
малого числа находок форма получается довольно расплывчатой и далекой от
нормального распределения. Однако, можно приблизительно оценить
медианы распределений. И кажется, что медиана распределения весов находок
среди альфапротеобактерий лежит правее медианы для гаммапротеобактерий. Так ли
это на самом деле проверялось при помощи программы STADIA.
Рабочие файлы: находки среди альфапротеобактерий и гаммапротеобактерий, книга Excel
Тест Вилкоксона
По распределениям весов находок в двух группах бактерий был в программе STADIA выполнен тест Вилкоксона.Его результат:
Вилкоксон=2675, Z=-1,938, Значимость=0,0263, степ.своб = 60,22 Гипотеза 1: <Есть различия между медианами выборок>Таким образом, распределения весов находок в 2 группах бактерий различны.
Для получения дополнительной информации воспользовались функцией "Описательная статистика":
Для альфапротеобактерий:
| Размер | <---Диапазон---> | Среднее---Ошибка | Дисперс | Ст.откл | Сумма |
| 60 | 8,035 10,55 | 8,844 0,06589 | 0,2605 | 0,5104 | 530,7 |
| Медиана | <--Квартили--> | ДовИнтСр. | <-ДовИнтДисп-> | Ош.СтОткл | |
| 8,864 | 8,397 9,034 | 0,1301 | 0,2131 0,2605 | 0,1547 | |
| Асимметр. | Значим | Эксцесс | Значим | ||
| 0,7652 | 0,0055 | 4,153 | 0,0126 |
Для гаммапротеобактерий:
| Размер | <---Диапазон---> | Среднее---Ошибка | Дисперс | Ст.откл | Сумма |
| 22 | 8,001 9,211 | 8,629 0,0848 | 0,1582 | 0,3977 | 189,8 |
| Медиана | <--Квартили--> | ДовИнтСр. | <-ДовИнтДисп-> | Ош.СтОткл | |
| 8,788 | 8,219 8,957 | 0,1742 | 0,09366 0,3231 | 0,1558 | |
| Асимметр. | Значим | Эксцесс | Значим | ||
| -0,366 | 0,2115 | 1,742 | 0,0917 |
Сравнивая статистические данные по 2 выборкам, можно сделать следующие выводы. Первоначально бросается в глаза тот факт, что диапазон значений весов находок для альфапротеобактерий шире, чем для гаммапротеобактерий (хотя по своей сути это наблюдение не является достаточно значимым). Больше нас интересует то, что выборочное среднее и медиана для альфабактерий больше, чем для гаммапротеобактерий Это говорит о том, что сравнивая на глаз гистограммы, мы были правы. Смещение средних значений весов свидетельствует о том, что в целом находки среди альфапротеобактерий "лучше". Дисперсии выборок показывают, что разброс значений весов находок меньше у гаммапротеобактерий (отчасти это связано с меньшим, по сравнению с альфапротеобактериями, объемом выборки).
В общем, и тест Вилкоксона, и сравнение гистограмм показали, что находки среди альфапротеобактерий "лучше", чем среди гаммапротеобактерий. То есть наш профиль, построенный по митохондриальным белкам, находит в 3 раза больше "гомологичных" белков среди альфапротеобактерий, причем в среднем эти находки лучше удовлетворяют профилю, чем белки гаммапротеобактерий. В какой-то степени это свидетельствует о близости митохондриального белка L14 белкам альфа-, а не гаммапротеобактерий.
Последовательности из Firmicutes были получены в результате поиска по SRS по запросу
Description L14 Ribosomal Taxonomy FirmicutesИз 135 находок были выбраны 6 последовательностей из разных родов.
Кратко информацию об исследуемых данных можно просмотреть в таблице:
Описание выборки
| Группа источников рибосом | Число последовательностей |
| Альфапротеобактерии | 60 |
| Гаммапротеобактерии | 22 |
| Фирмикуты | 6 |
| Митохондрии | 8 |
При помощи программы muscle было построено множественное выравнивание всех исследуемых последовательностей. По нему программой proml из пакета PHYLIP было построено филогенетическое дерево методом максимального правдоподобия.
Изображение дерева (из TreeView):
Пометку "e" имеют митохондриальные белки, "a", "g" -
соответственно альфа- и гаммапротеобактерий, "o" - белки из out-группы.
Видно, что белки фирмикут на дереве
представляют собой отдельную группу, хорошо выделенную из остальных белков.
Так же обстоит дело с гаммапротеобактериями. Белки же альфапротеобактерий
нельзя по какой-либо одной ветви отделить от других. Если же говорить об
эукариотических белках, они на дереве отнесены очень далеко от других
последовательностей. Однако можно сказать, что они эволюционно находятся
ближе к альфапротеобактериям, чем к гаммапротеобактериям.
Причем 2 наиболее близкие к эукариотам последовательности альфапротеобактерий
принадлежат одному и тому же организму
Orientia tsutsugamushi
с таксономией Bacteria; Proteobacteria; Alphaproteobacteria; Rickettsiales;
Rickettsiaceae; Rickettsieae; Orientia.
Эволюционные расстояния
Для 96 исследуемых последовательностей были определены попарные эволюционные расстояния по модели JTT с помощью программы protdist пакета PHYLIP.Полученную матрицу импортировали в Excel, где построили гистограммы распределения попарных расстояний между митохондриальными белками и белками из альфа- и гаммапротеобактерий.
Гистограмма:
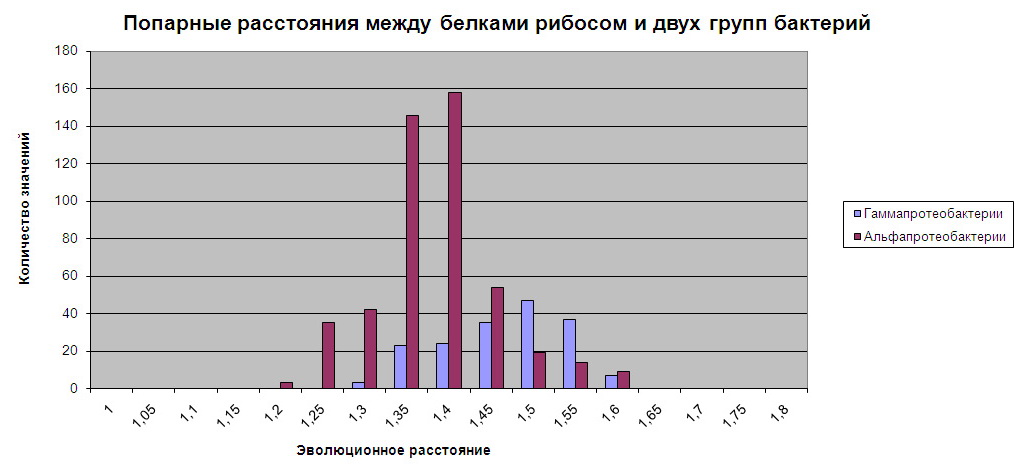
Отчетливо видно, что
медиана распределения количества последовательностей с определенным
эволюционным расстоянием между ними по расстоянию для альфапротеобактерий
лежит левее, чем для гаммапротеобактерий.
То есть в среднем
попарное эволюционное расстояние между митохондриями и гаммапротеобактериями
больше, чем между митохондриями и альфапротеобактериями. А это, в свою
очередь, говорит о том, что эволюционно митохондрии ближе альфапротеобактериям
(результат совпадает с выводами, сделанными по структуре дерева, и
это не удивительно: и матрица, и дерево строились по одному выравниванию).
Рабочие файлы: книга Excel,
матрица расстояний.
Однако следует заметить, что исследуемые последовательности митохондриальных рибосом эукариот принадлежат очень близким организмам, вследствие чего слишком похожи. На практике это привело к созданию профиля, удовлетворяющего в большей степени лишь некоторой части митохондриальных рибосомальных белков L14.