Трансмембранные белки
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения задания был выбран белок LptE. Вместе с белком LptD он образует комплекс сборки липоплисахарида (5iv9).
Английское название: LPS-assembly protein complex.
Uniprot: A0A0J4W1Y0_KLEVA.
Из организма Klebsiella pneumoniae.
TM segments: 1( 205- 210), 2( 218- 225), 3( 233- 238), 4( 246- 254), 5( 261- 268), 6( 287- 296), 7( 303- 310), 8( 332- 340), 9( 344- 353),10( 366- 376),11( 382- 391),12( 405- 416),13( 423- 432),14( 458- 467),15( 481- 490),16( 528- 538),17( 548- 557),18( 580- 587),19( 593- 600),20( 610- 618),21( 624- 631),22( 655- 664),23( 671- 676),24( 685- 693),25( 700- 707),26( 724- 731)
Последовательность, взятая из базы данных PDB, была загружена в DeepTMHMM для определение трансмембранный участков. Были найдены бета-листы со следующими координатами: 1(207-213), 2(217-223), 3(233-241), 4(245-253), 5(287-294), 6(261-275), 7(287-294), 8(302-310), 9(331-339), 10(344-351), 11(368-375), 12(384-390), 13(407-414), 14(424-431), 15(459-465), 16(484-490), 17(528-535), 18(551-557), 19(578-585), 20(596-602), 21(608-614), 22(627-632), 23(654-660), 24(673-679), 25(683-690), 26(702-708), 27(723-729). Полную выдачу программы можно посмотреть по ссылке. Полученное изображение показано на рисунке 1.
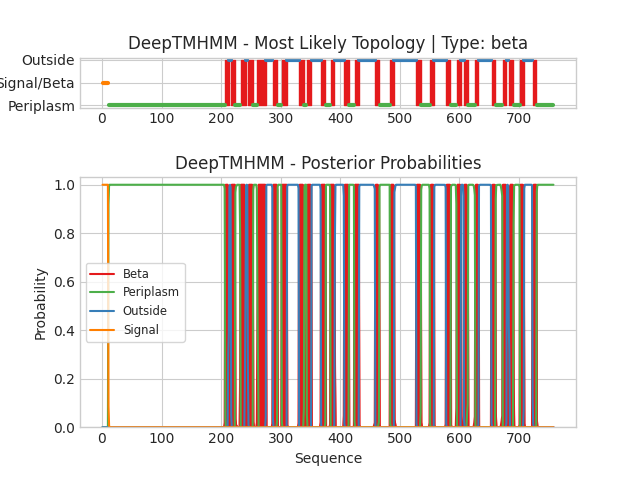
Данные из базы данных почти точно совпадают с выдачей DeepTMHMM. Количество бета-листов одинаковое. Немного отличаются координаты, т.к. в DeepTMHMM даны координаты самих бета-листов, а в базе данных - координаты их трансмембранных участков. Небольшие фрагменты бета-листов выходят за пределы мембраны, поэтому они длиннее трансмембранных участков.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Для задания был взят белок Y1470_HAES1 - предполагаемый транспортный белок из Haemophilus somnus.
С помощью DeepTMHMM были найдены трансмембранные спирали со следующими координатами: 1(4-22), 2(31-47), 3(61-83), 4(91-112), 5(160-177), 6(370-387), 7(401-415), 8(429-447), 9(463-484), 10(526-544). На рисунке 2 показан результаты работы DeepTMHMM. Выдачу можно посмотреть по ссылке.
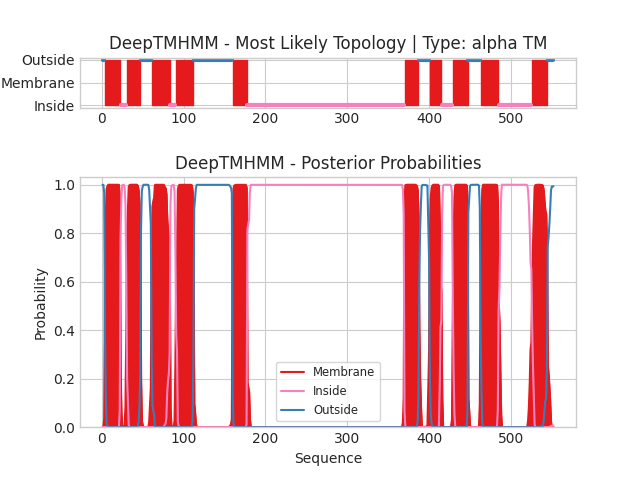
Далее файл сос структурой был скачан и загружен на сервер PPM со следующими параметрами: Type of membrane - Undefined (т.к. функция белка точно не известна); Allow curvature - YES; Topology (N-ter) - out (из рисунка 2 видно, что начало находится снаружы); Include heteroatoms, excluding water and detergents, for positioning in membrane - yes (гетероатомы могут участвовать во взаимодействии с мембранными липидами). В результате была получена модель. Были найдены трансмембранные участки со следующими координатами: 1( 4- 23), 2( 33- 50), 3( 56- 79), 4( 91- 112), 5( 120- 133), 6( 134- 143), 7( 151- 177), 8( 370- 385), 9( 401- 415),10( 430- 455),11( 462- 484),12( 529- 548). Выдачу программы можно посмотреть по ссылке.
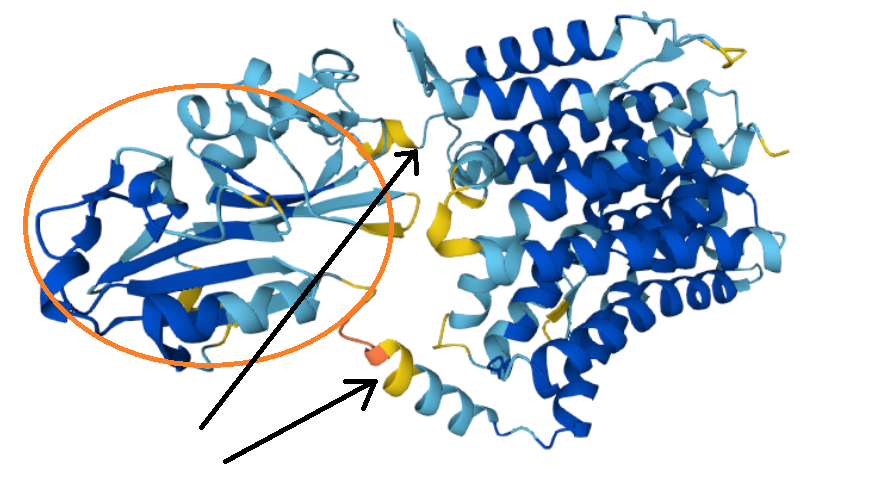
Результаты, полученные двумя методами отличаются: РРМ нашел 12 трансмембранных участков, а DeepTMHMM - 10. РРМ нашел трансмембранные участки 5( 120- 133), 6( 134- 143), которые не нашла вторая программа. Они расположены середине последовательности. Этот участок обозначен оранжевым овалом на рисунке 3. Мне кажется, PPM считает, что белок изгибается, и этот участок тоже попадает в мембрану. Отличия могут быть связаны с тем, что "перемычки" между двумя структурными блоками белка (черные стрелочки) предсказаны менее точно по сравнению с другим участками белка (они покрашены в желтый цвет).