Структура предполагаемой цинк-зависимой пептидазы (THE PUTATIVE ZN-DEPENDENT PEPTIDASE) бактерии G.sulfurreducens, полученная в редакторе Jmol
На текущих занятиях по информатике мы начали проходить программы для просмотра и редактирования структур химических молекул, в нашем случае, белков. Программа, с которой мы познакомились, - Jmol. Именно с помощью неё я получила структуру моего белка Zn-зависимой пептидазы, которая изображена на рисунке 1.
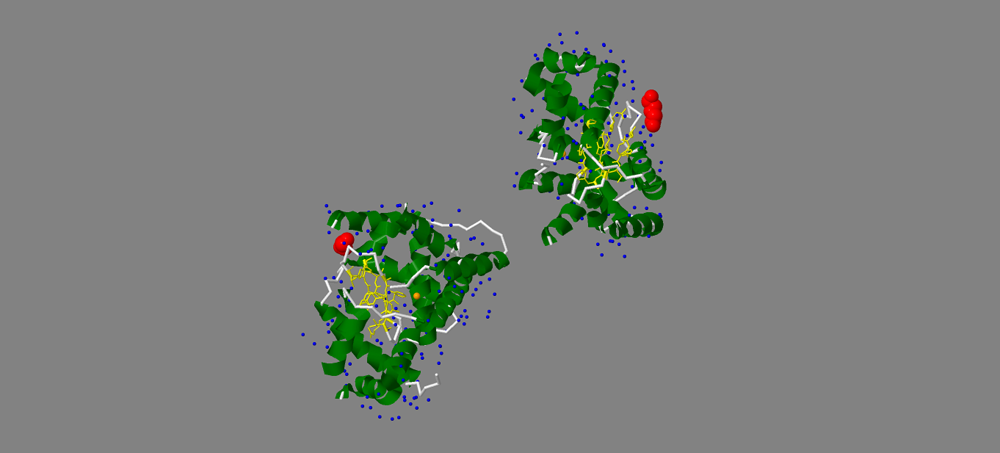
Рисунок 1. Структура белка предполагаемая Zn-зависимая пептидаза G.sulfurreducens штамм PCA в программе Jmol.
Данный белок состоит из 2 цепей: A и B.
Цепь А состоит из 1922 атомов и включает такие структуры как α-спираль (отмечена зелёным цветом, выполнена в стиле ribbons)и β-слой (отмечен жёлтым цветом, выполнен в стиле wireframe). Также в цепи А есть такие элементы как Zn (оранжевый, cpk), вода (HOH, синий, cpk) и лиганд PEG (красный, cpk). Строение цепи B аналогично, но состоит она из 1900 атомов.
Если Вы хотите получить в Jmol'е такую же структуру белка, Вам достаточно запустить в нём следующий скрипт:
load 3C37.pdb background grey cartoons off cpk off wireframe off select all backbone 100 color white select helix color green ribbons backbone off select sheet wireframe 100 backbone off wireframe 50 color yellow select water color blue cpk 100 select zn color orange cpk 200 select peg color red cpk rotate z -134.34 rotate y 102.84 rotate z -101.01 zoom 49.72
Pdb-файл может использоваться не только для построения визуальных структур, но и для передачи информации о данном веществе. Например, из файла может быть получена следующая информация (была получения с помощью команды egrep '^SOURCE|EXPDTA|TITLE|COMPND|REMARK' 3C37.pdb > Mukhaleva_pr1.txt в bash):
1. Название: Рентгеновская структура предпологаемой цинк-зависимой пептидазы Q74D82 в разрешении 1.7 А, цель GSR143A северо-западного консорциума по геномике - TITLE X-RAY STRUCTURE OF THE PUTATIVE ZN-DEPENDENT PEPTIDASE Q74D82 AT THE RESOLUTION 1.7 A. NORTHEAST STRUCTURAL GENOMICS CONSORTIUM TARGET GSR143A
2. Заголовок: гидролаза 27 января 2008 (дата создания файла) 3C37 (код в Protein Data Bank) -HEADER HYDROLASE 27-JAN-08 3C37
3. Состав: молекула: пептидаза из семьи пептидаз M48 - COMPND 2 MOLECULE: PEPTIDASE, M48 FAMILY;
4. Состав: цепи: А;В - COMPND 3 CHAIN: A, B;
5. Способ получения: дифракция рентегновских лучей кристаллической структурой вещества - EXPDTA X-RAY DIFFRACTION
6. Лиганды в белке: цинк (Zn), селенметионин (MSE) и дигидроксилэтиловый эфир (PEG)
7. Местонахождения: организм: G.sulfurreduncens PCA - SOURCE 2 ORGANISM_SCIENTIFIC: GEOBACTER SULFURREDUCENS PCA
За более подробной информации можете обратиться к файлу: Информация о Zn-зависимой пептидазе. А на рисунке 2 показана структура белка, прокрашенная в направлении от начала к концу следующим образом: красный - оранжевый - жёлтый - зелёный - голубой - синий - фиолетовый - малиновый - розовый - коралловый - сиреневый.
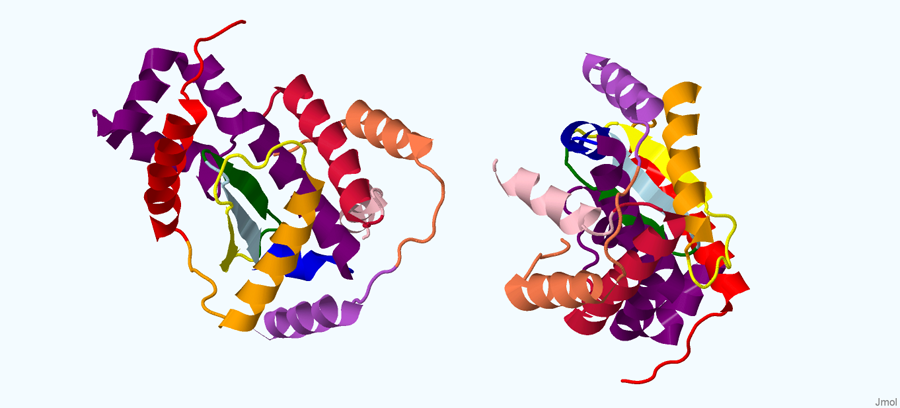
Рисунок 2. Структура белка предполагаемая Zn-зависимая пептидаза G.sulfurreducens штамм PCA, прокрашенная в направлении начало-конец.
Дата последнего изменения: 16.03.2014
