Понятие о выравнивании. Построение выравниваний в Jalview
После успешного освоения правил пользования базами данных, мы приступили к изучению понятия "выравнивания". Выравнивание - это расположение друг под другом несколько последовательностей (ДНК, РНК, аминокислотных) с целью обнаружения гомологичных участков. В начале года мы пытались сделать это вручную в Microsoft Office Word, теперь для этого нам рассказали о специальной программе - JalView(сайт разработчика), с помощью которой выполнялись последующие задания.
Для выполнения задания потребовалось множественное выравнивание: загрузить исходное выравнивание (я выбрала aln26_1.fa), проект JalView. Окраску я выбрала ClustalX с параметром консервативности (Above identity threshold ) > 70%.
Для начала были определены вертикальные блоки - части выравнивания, где совпадают аминокислотные последовательности всех белков. Гомологичность выбранных мною белков оказалась высокой, поэтому я выбрала 2 блока, представленных на рисунках 1 и 2. Все рисунки в дальнейшем были получены с помощью JalView.
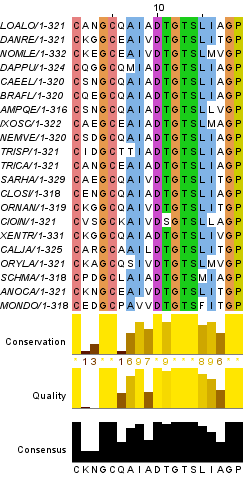
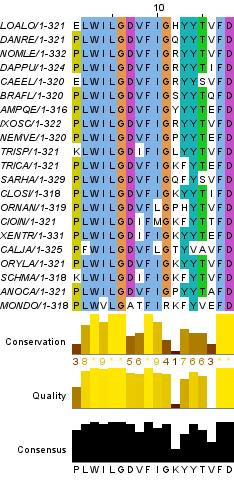
Рисунок 1. Первый вертикальный блок выравнивания (позиции №№ 219-237). Рисунок 2. Второй вертикальный блок выравнивания (позиции №№ 316-333).
Рассмотрим подробнее первый блок выравнивания. Для него были посчитаны параметры, оценивающие гомологичность последовательностей. Результаты представлены в таблице 1.
Таблица 1. Таблица параметров, оценивающих гомологичность участка.
| Количество | № позиций | Процент позиций | |
| Общее количество позиций | 19 | 219-237 | 100% |
| Абсолютно консервативные позиции | 9 | 219, 222-223, 228, 230-232, 236-237 | 47,4% |
| Абсолютно функционально консервативные позиции | 3 | 227, 229, 234 | 15,8% |
| Консервативные на 70% позиций | 5 | 225, 226, 229, 233-234 | 26,3% |
| Функционально консервативные на 70% позиций | 2 | 225, 235 | 10, 5% |
Далее был определён участок, где какие-то последовательности являются гомологичными, а о гомологичности остальных данных нет. Для этого было построено дерево, определяющее сходство последовательностей по времени их расхождения в процессе эволюции (представлено на рисунке 3). Согласно нему, последовательности BRAFL и TRICA имеют самое близкое родство (один из участков гомологии представлен на рисунке 4).
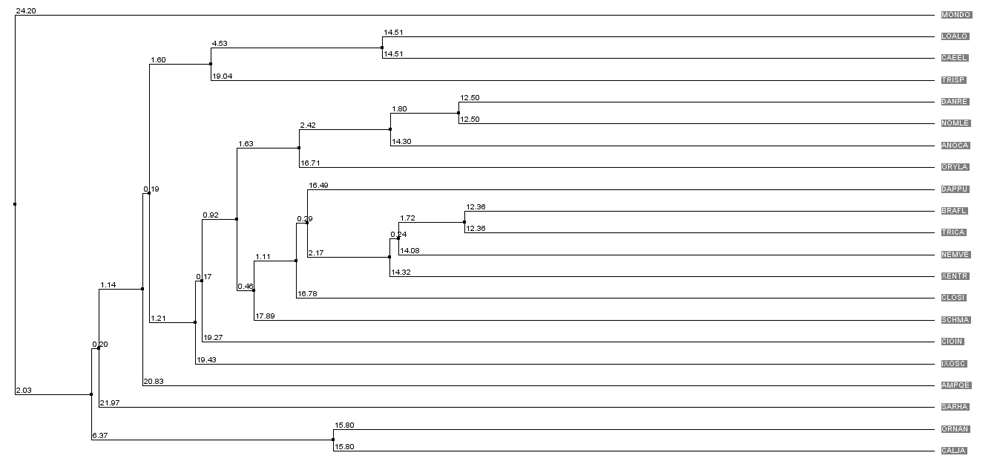
Рисунок 3. Дерево сходства последовательностей.

Рисунок 4. Участок выравнивания (позиции №№ 31-40).
Следующим заданием было рассмотрение близко расположенных вертикальных блоков. На рисунке 5 изображён участок,в который вошли те самые блоки, представленные на рисунках 1 и 2, т.к. между ними 80 позиций, а это входит в диапозон "близости". Рассмотрим участок, начиная с позиции 219 и заканчивая позицией 333, то есть всего 115 позиций. Из них 8 позиций с гэпами, что составляет 6,96% от всего количества.
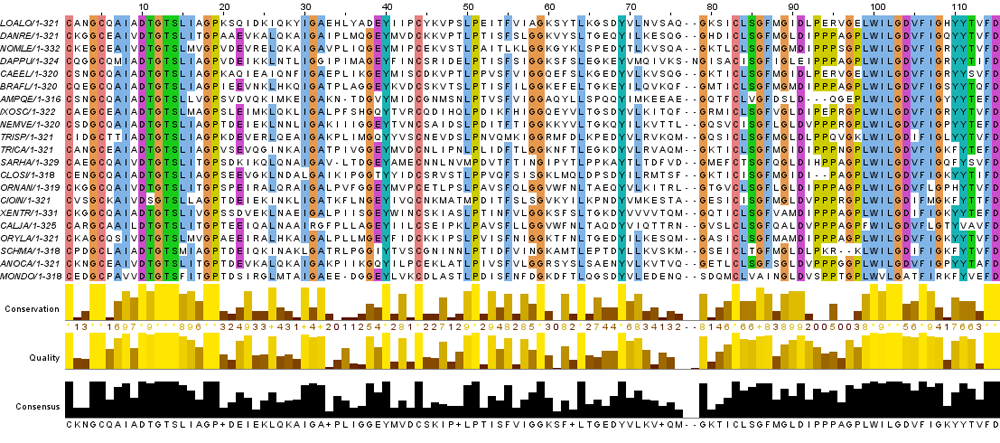
Рисунок 5. Близко расположенные вертикальные участки(позиции №№ 219-333).
Затем нам было предложено добавить последовательность и выровнять её относительно первого блока. Для этого даже не пришлось двигать добавленную последовательность стрелками - она сразу встала в свою лучшую позицию.
Само выравнивание по ней представлено на рисунке 6.
Консенсусная последовательность данного выравнивания:
QYYGEISIGTPPQNFTVVFDTGSSNLWVPSKKCSFTDIACLLHNKYDSSKSSTYKKNGTEFAIQYGSGSLSG FLSQDTVSI-----------GGIKVKNQTFAEATSEPGLTFVAAKFDGILGMAYPSISVDGVTPVFDNMIAQ GLVDEPVFSFYLNRDPSAEPGGELILGGTDPKYYKGDFTYVPVTRKAYWQFKMDGVSVG-ES----A---GL TLCKNGCQAIADTGTSLIAGPSDEIEKLQKAIGAKPLIGGEYMVDCSKIPSLPTISFVIGGKSFSLTGEDYV LKVSQM--GKTICLSGFMGLDIPPPAGPLWILGDVFIGKYYTVFDRGN----NRVGFAKA
С помощью данного сервиса было создано лого данного выравнивания - графическое отображение консенсусной последовательности (представлено на рисунке 7).
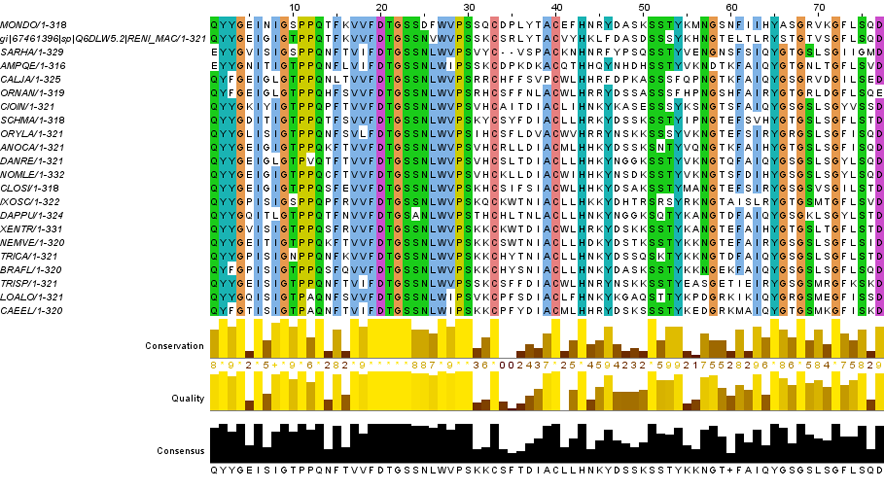
Рисунок 6. Выравнивание новой последовательности относительно первого вертикального блока.

Рисунок 7. Лого консенсусной последовательности.
Последним заданием было выравнивание негомологичных последовательностей. Для этого нужно было выбрать 5 последовательностей других студентов (мой выбор пал на Сашу Погорельскую, Ваню Кузнецова, Алину Софронову, Ирину Хвень и Ярослава Лозинского),извлечь их из базы данных с помощью JalView (File -> Fetch sequences) и выровнять с помощью сервиса MUSCLE. В итоге получилось выравнивание, которое можно скачать по этой ссылке.
Затем нужно было найти 2 вертикальных блока, как делали вначале. Они представлены на рисунках 8 и 9. Окрашивание было поставлено 50%, так как на 70% было лишь несколько отдельных столбиков. Видно, что последовательности негомологичны, так как получившиеся блоки слабо похожи на достоверные.
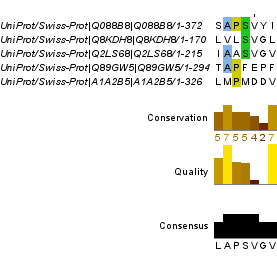
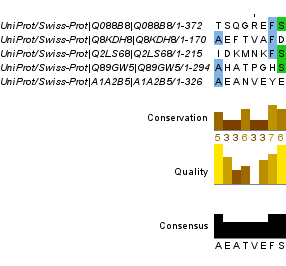
Рисунок 8. Первый вертикальный блок выравнивания (позиции №№ 38-43). Рисунок 9. Второй вертикальный блок выравнивания (позиции №№ 239-247).
Проект данного практикума можно скачать по этой ссылке.
Дата последнего изменения: 14.04.2014
