Предсказание генов у эукариот
Задача этого практикума - для своего фрагмента определить экзон-интронную структуру гена и описать его альтернативный сплайсинг, используя программы GENSCAN и UCSC Genome Browser.
GENSCAN
Предсказания интрон-экзонной структуры генов мы делали при помощи программы GENSKAN. На вход была подана нашу последовательность из генома человека, результат GENSCAN представлен в Таблице 1. GENSKAN выдал помимо целого гена, окончание другого, начиющегося в другом контиге. Его в таблицу я включать не стала.
Таблица 1. Результаты GENSKAN.
| Начало | Конец | Цепь | Тип |
| 11986 | 12025 | + | промотор |
| 12193 | 12265 | + | начальный |
| 12734 | 12773 | + | внутренний |
| 13701 | 13867 | + | внутренний |
| 18944 | 19095 | + | внутренний |
| 20275 | 20488 | + | внутренний |
| 21272 | 21406 | + | внутренний |
| 21767 | 21835 | + | внутренний |
| 22040 | 22155 | + | внутренний |
| 22198 | 22435 | + | внутренний |
| 23882 | 24001 | + | внутренний |
| 24276 | 24402 | + | внутренний |
| 27240 | 27313 | + | внутренний |
| 27400 | 27490 | + | внутренний |
| 27694 | 27893 | + | внутренний |
| 28255 | 28387 | + | внутренний |
| 28469 | 28559 | + | внутренний |
| 29274 | 29434 | + | внутренний |
| 31741 | 31788 | + | внутренний |
| 35273 | 35411 | + | внутренний |
| 35492 | 35614 | + | внутренний |
| 35866 | 35964 | + | внутренний |
| 36680 | 36865 | + | внутренний |
| 37003 | 37158 | + | внутренний |
| 37281 | 37356 | + | внутренний |
| 37661 | 37843 | + | внутренний |
| 38516 | 38613 | + | внутренний |
| 38769 | 38957 | + | внутренний |
| 39249 | 39501 | + | внутренний |
| 39643 | 39998 | + | конечный |
Genome Browser
Теперь предскажем структуру гена другим способом - с помощью Genome Browser. Он позволяет просмотреть разнообразную информацию, относящуюся к заданному фрагменту ДНК и использует программу BLAT, аналогичную blast. Для получения результатов нужно было поместить последовательность ДНК в текстовое поле формы, выбрать поиск в геноме человека, сборка hg38 (Dec. 2013). Среди всех находок выбрала одну с наибольшим весом и идентичностью 100%, скрыла все параметры, кроме Blat Sequence.
На рисунках 1, 2, 3, 4 и 5 можно заметить примеры альтернативного сплайсинга.
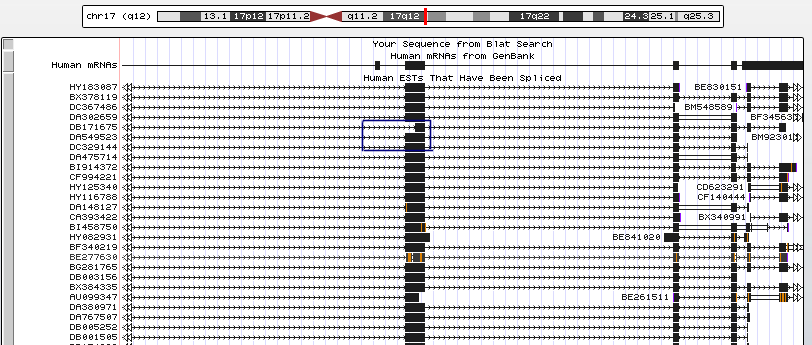
Рисунок 1. Альтернативные акцепторные сайты. Обведены синей рамкой. мРНК DB171675-DA549523.
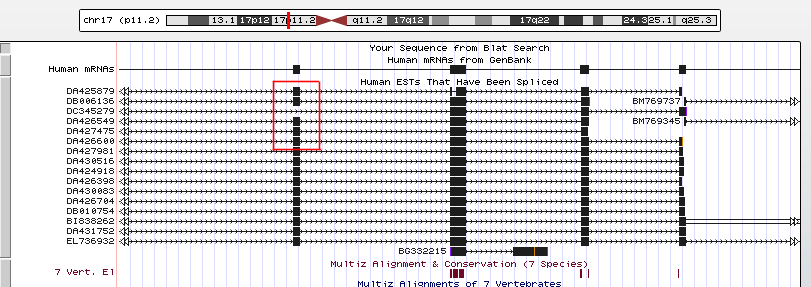
Рисунок 2. Касcетные экзоны. Обведены красной рамкой. мРНК DC345279-DA426549.
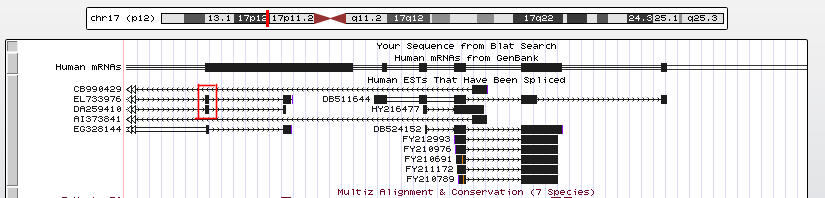
Рисунок 3. Касcетные экзоны. Обведены красной рамкой. мРНК CB990429-EL733976.
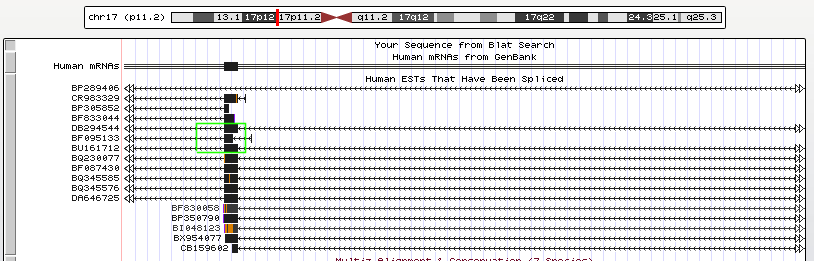
Рисунок 4. Альтернативные донорные сайты. Обведены зеленой рамкой. мРНК DB294544-BF095133.
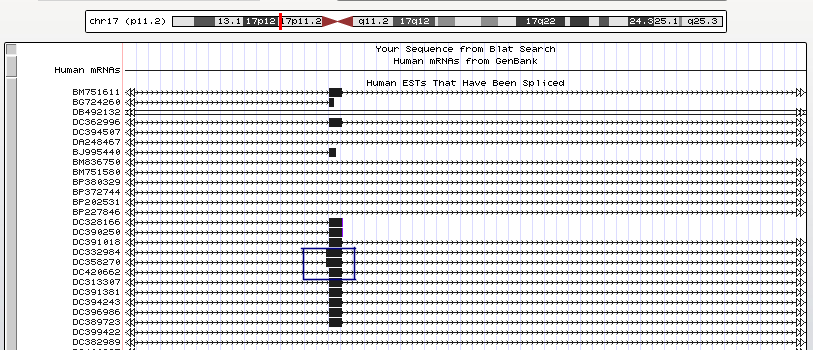
Рисунок 5. Альтернативные акцепторные сайты. Обведены синей рамкой. мРНК DC358270-DC420662.
BLASTX
В это задании проводилась работа с фрагментом ДНК из генома киви Actinidia chinensis. Для него проводился поиск гомологов с помощью blastx с установленными параметрами: банк SwissProt, стандартный генетический код, исключены Models (XM/XP) и Uncultured/environmental sample sequences, исключён геном винограда Vitis vinifera, поиск ограничен белками Viridiplantae. В итоге было найдено 6 генов, которые можно увидеть на рисунке 6.
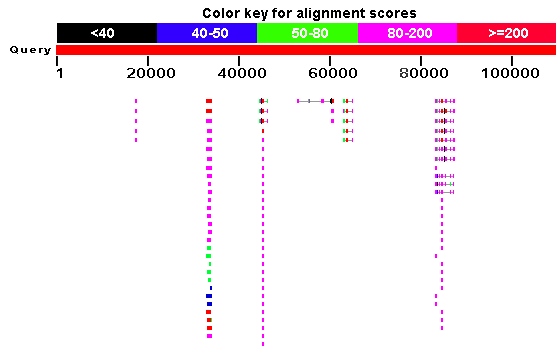
Рисунок 6. Результаты поиска blastX против базы SwissProt.
В таблице 2 приведена информация об этих генах.
Таблица 2. Информация о предсказанных генах.
| Начало экзона | Конец экзона | Цепь |
| Дерлин-2.1 | ||
| 15151 | 15237 | + |
| 15758 | 15862 | + |
| 17597 | 17788 | + |
| 17942 | 17998 | + |
| 18171 | 18171 | + |
| 20038 | 20217 | + |
| Белок-пентатрикопептид, содержащий повторы | ||
| 33102 | 34286 | + |
| Сери-треониновая протеин киназа | ||
| 45162 | 45198 | + |
| 45202 | 45668 | + |
| 44872 | 45057 | + |
| 44592 | 44768 | + |
| 46262 | 46444 | + |
| Гомолог 4 фактора полиаденилирования и отщепления | ||
| 52983 | 53339 | + |
| 55641 | 55859 | + |
| 58266 | 58781 | + |
| 55400 | 55543 | + |
| 60235 | 60300 | + |
| 60426 | 60932 | + |
| Триптофановая синтаца β-цепи 2 | ||
| 62947 | 63084 | - |
| 63200 | 63319 | - |
| 63696 | 64112 | - |
| 64854 | 65219 | - |
| Белок-лейциновая застёжка гомеобокса | ||
| 83154 | 83222 | - |
| 83353 | 83522 | - |
| 83615 | 83713 | - |
| 84022 | 84300 | - |
| 84387 | 84481 | - |
| 84691 | 84857 | - |
| 85023 | 85124 | - |
| 85222 | 85638 | - |
| 86330 | 86524 | - |
| 87291 | 87444 | - |
| 87016 | 87083 | - |
Дата последнего изменения: 15.09.2013
