Онлайн BLAST
Этот практикум был посвящён освоению BLAST при поиске гомологов нуклеотидных последовательностей.
Поиск организма по фрагменту нуклеотидной последовательности
Для выполнения этого задания была выдана нуклеотидная последовательность:
tgcgcgatgtgggcattacggcaattatccactctcccctctctaccactttggcggggc taatcctgcgcctgcccggaaaagccgccactattctatctctgtgcgccgcggcgacgg ccgccaaataggccttgacggcttgaaacgccttaccagcggcgtttcggatgaggccgt tttttaaaaactcctcggccagctcggcctcgtacaacgcctcttttaagcggtctcgcc ggtacttttctaaatccacccaaggcttggagagagcaatcacgataaatgccgcgggta
В этом задании нужно было по заданной последовательности найти, к какому организму она относится, используя megablast. В критериях поиска была выбрана база данных "refseq_genomic" и убрана галочка с Low complexity regions в разделе Filters and Masking. Также в задании нужно было ограничен поиск только бактериями, но так ничего не нашлось. Поэтому я убрала этот критерий, и тогда нашлось два организма, первый из которых и есть нужная бактерия.
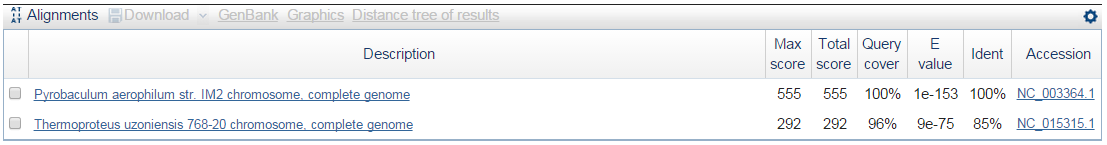
Рисунок 1. Результаты поиска организмов по заданной последовательности.
Нужной бактерией оказалась Pyrobaculum aerophilum str. IM2. AC записи Ref_seq этой бактерии: NC_003364. Координаты этого отрывка: 1145..1444, он не является кодирующим.
Поиск гомолога белка человека в слоне
Следующим заданием был поиск гомологов выбранного человеческого белка в геноме африканского слона. Выбор белка осуществлялся так: идентификатор Swiss-Prot и моя фамилия (Mukhaleva) должны максимально совпадать. Проверка на совпадение проводилась с помощью следующей команды: infoseq sw:x*_human -only -name -desc -out file_name.txt, где, в моём случае, х оказался "mu". Из списка белков я выбрала один - предшественник антигена рака желудка Ga34 (Mucen-14) (его fasta-файл), и стала искать его гомологов с помощью ENA Sequence Search. Среди организмов для поиска я выбрала Loxodonta africana, а параметр поиска - "spliced translated nucleotide search". В итоге был найден 1 гомолог, которы1 можно посмотреть на рисунке 1.

Рисунок 2. Результаты поиска гомологов предшествнника антигена рака груди в геноме африканского слона.
Параметры выравнивания: E-Value: 8E-17, длина выравнивания - 67, Identity(%): 71, координаты гена - Target Range: 7157354<-7147387. В этом гене содержится 1 интрон.
Поиск гомолога белка человека в слоне
Для выполнения этого пункта практикума мною был создан fasta-файл с поледовательностью серотониновой тРНК из бактерии Geobacter sulfurredescens PCA (ID:NC_002939), координаты в геноме которой следующие: 48680-48765. Далее был определён порядок моей бактерии ( с помощью NCBI-Taxonomy) - он оказался Desulfuromonadales. И теперь, когда все приготовления были завершены, был проведён поиск гомологов этой последовательности среди организмов того же порядка при помощи 3 разных способов. Результаты поиска представлены в таблице 1.
Таблица 1. Результы поиска гомологов серотониновой тРНК.
| Параметр сравнения | Megablast | Blastn с параметрами по умолчанию | Blastn с длиной "слова" 7, match/mismatch = 1/-1 |
| Количество результатов с e-value < 0,001 | 2 | 16 | 20 |
По результатам таблицы можно заметить, что Megablast ищет самые близкие гомологи, в то время как blastn расширяет список, но тоже ещё может использоваться. Но blastn с особыми параметрами выдаёт уже довольно далёкие гомологи, поэтому им пользоваться нежелательно.
Сравнение программ BLASTN и MegaBLAST
В этом пункте необходимо было провести более детальную работу со списками гомологов, полученными заранее. При использовании поиска megablast было найдено 2 гомолога к различным родам из различных семейст (Pelobacteraceae, Pelobacter и Geobacteraceae, Geobacter). В результатах поиска blastn - 16 гомологов, из которых 12 относятся к роду Geobacter семейства Geobacteraceae и 4 - к роду Pelobacter семейства Pelobacteraceae. Помимо этих двух семейств, в порядке Desulfuromonadales есть ещё одно - Desulfuromonadaceae. Так что, получается, нельзя утверждать, что blastn расширяет диапозон поиска внутри отряда, так как в обоих алгоритмах в результате получились одни и те же рода и семейства. По этому критерию точность поиска megablast и blastn может считаться примерно одинаковой.
Можно сравнить алгоритмы по другому критерию: длине выравниваний. В обоих случаях параметр Query cover равен 100%, что означает длину выравнивания значением в 86. Но при этом длина выравниваний у blastn колеблется от 86 до 91, что позволяет усомниться в точности метода. В итоге, по критерию длины выравнивания явно заметно, что megablast предпочтительнее blastn.
Дата последнего изменения: 22.09.2014
