Предсказание генов у прокариот
В этом практикуме был дан контиг микробиома кардиального отдела желудка валлаби. Сначала нужно было найти в нём открытые рамки считывания длиной 60 триплетов и более, используя бактериальный генетический код (11). Для этого использовалась уже изученная программа getorf. Команда: getorf -minsize 180 -table 11 -find 1 kontig.fasta. В результате был получен следующий файл, в котором оказалось 16 рамок считывания: 7 на прямой и 9 на обратной цепи. На прямой цепи: третья [1411 - 1590] рамка полностью содержится в четвёртой [375 - 1688], пятая [1893 - 2165] - полностью в шестой [1688 - 2572], что позволяет предположить, что третья и пятая рамки были определены ошибочно. На обратной цепи: десятая [1679 - 1362] и одиннадцатая [1393 - 1103] рамки перекрываются на 31 нуклеотид, двенадцатая [1028 - 648] и тринадцатая [753 - 571] - на 105 нуклеотидов, тринадцатая и четырнадцатая [638 - 435] - на 67 нуклеотидов, пятнадцатая [342 - 148] и шестнадцатая [226 - 2] - на 78 нуклеотидов.
Далее для каждой найденной рамки был проведён поиск гомологов. Для этого использовался локальный blast через опцию -remote: blastp -query kontig.orf -db swissprot -out blast.out -evalue 0.001 -outfmt 7 -remote. Полученный результат - blast.out. В итоге для первой рамки считывания было найдено 373 гомолога, для четвертой - 387, для шестой - 2, для остальных - 0. Наличие гомологов свидетельствует о достоверности найденных рамок считанные, среди же тех, которым гомологи не нашлись, я оставила только те, у которых большая длина. Информация о рамках считывания, которые я посчитала вероятными - а это 1, 4, 6, - представлена в таблице 1.
Таблица 1. Информация об открытых рамках считывания данного контига, найденных с помощью blast и getorf.
| Начало | Конец | Длина в а.о. | Цепь | Описание |
| 4 | 249 | 246 | прямая | предполагаемый мембранный белок |
| 375 | 1688 | 1314 | прямая | мембранный белок-инсертаза YidC 1 |
| 1688 | 2572 | 885 | прямая | белок jag |
Помимо поиска гомологов последовательностей рамок считывания, предсказать гены контига можно другим способом - используя программу GeneMark. В результате был получен pdf-файл с графиком кодирующего потенциала и fasta-файл с рамками. Их было найдено всего две, информация представлена в таблице 2.
Таблица 1. Информация об открытых рамках считывания данного контига, найденных с помощью GeneMark.
| Начало | Конец | Длина в а.о. | Цепь |
| 402 | 1691 | 129 | прямая |
| 1691 | 2575 | 885 | прямая |
Эти рамки нашёл и getorf (4 и 6). Однако координаты отличаются на 3 а.о. (стоп-кодон), и для четвёртой рамки считывания сдвинута первая координата на 27 нуклеотидов в сторону уменьшения гена (с 375 на 402 нуклеотид). Это связано с тем, что рамка началась с другого старт-кодона. Для обоих находок GeneMark были найдены гомологи с помощью blast. Рассмотрим графики кодирующего потенциала этих находок (рисунки 1 и 2).
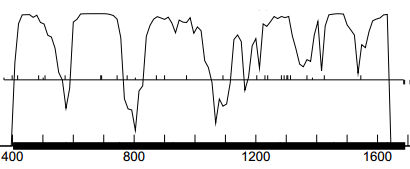
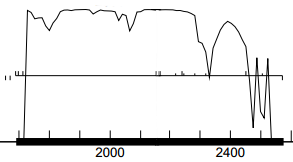
Рисунок 1. Участок, предположительно кодирующий первый белок [402-1691]. Рисунок 2. Участок, предположительно кодирующий второй белок [1691-2575].
На втором участке (1691-2575) график идёт без явных спадов, имя провалы лишь в конце, так что можно считать, что он кодирует один ген. Но в первом же графике есть два провала: если второй ещё можно посчитать незначительным, то первый почти доходит до нуля, что позволяет предположить, что этот участок кодирует два гена.
На рисунке 3 показана графическая интерпретация находок getorf и GeneMark. Фиолетовым выделены рамки считывания, найденные в blast и getorf и подтвердившиеся в GeneMark, синим - найденные только с помощью blast и getorf, зелёным - найденные GeneMark.
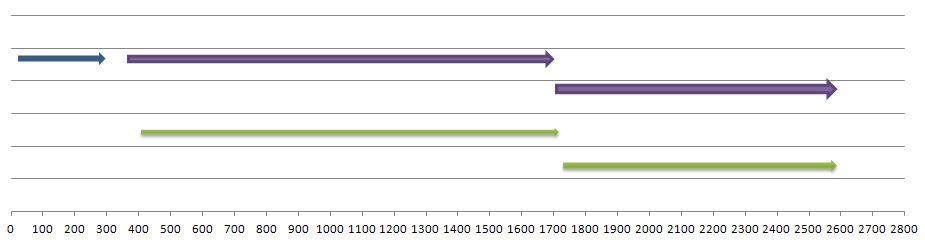
Рисунок 3. Наглядное представление сравнения предсказания генов с помощью getorf и blast с предсказанием через GeneMark.
Дата последнего изменения: 15.09.2013
