A- и B- формы ДНК. Структура РНК
Темой этого практикума стала структура нуклеиновых кислот, поэтому работали мы с уже известным Jmol. Первым заданием было получение модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA, установленном в Putty. А- и В-формы состоят из 5 раз повторённой последовательности "gatc", в то время как Z-форма встречается в областях, насыщеннных G-C парами, поэтому в этом файле будет содержаться десять таких последовательностей. Файлы, полученные мною, можно скачать по следующим ссылкам: gatc-a, gatc-b и gatc-z.
Во втором задании нужно было выделить в А-форме указанные объекты с помощью Jmol. Результат показан на рисунках ниже.
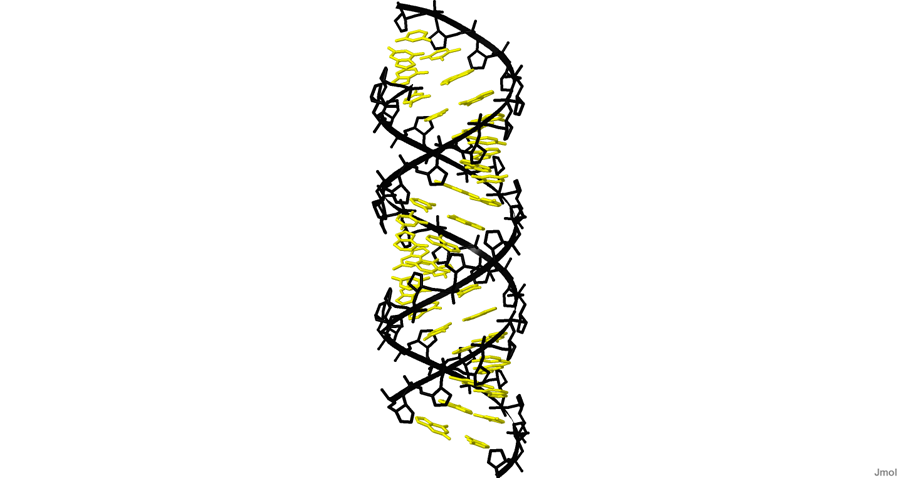
Рисунок 1. Cахарофосфатный остов ДНК выделен чёрным цветом. Изображение получено с помощью Jmol.
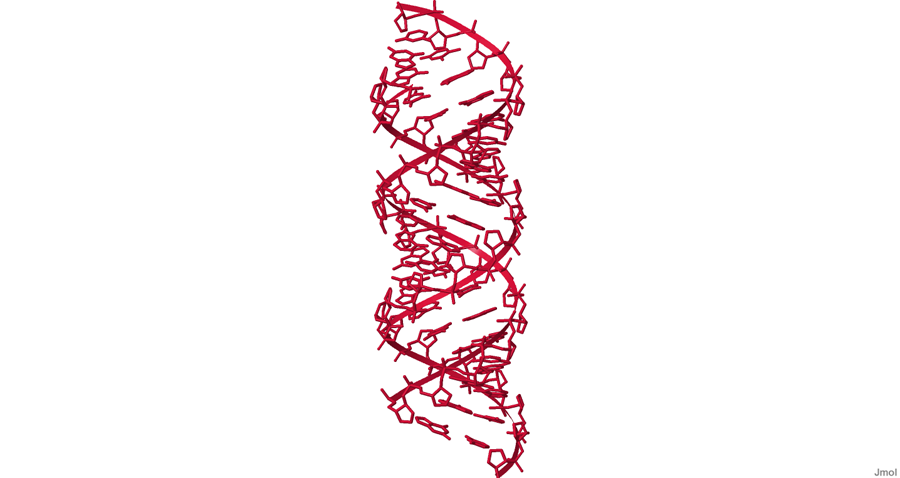
Рисунок 2. Нуклеотиды выделены малиновым цветом. Изображение получено с помощью Jmol.
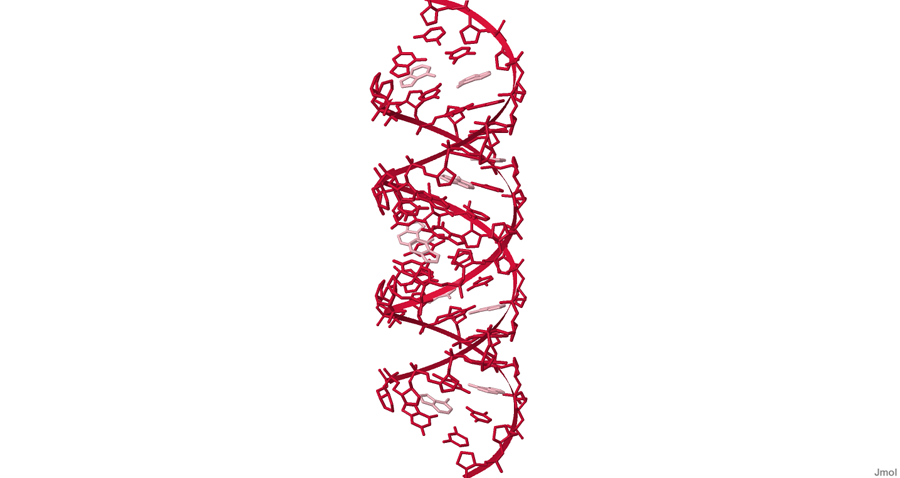
Рисунок 3. Аденины выделены розовым цветом. Изображение получено с помощью Jmol.
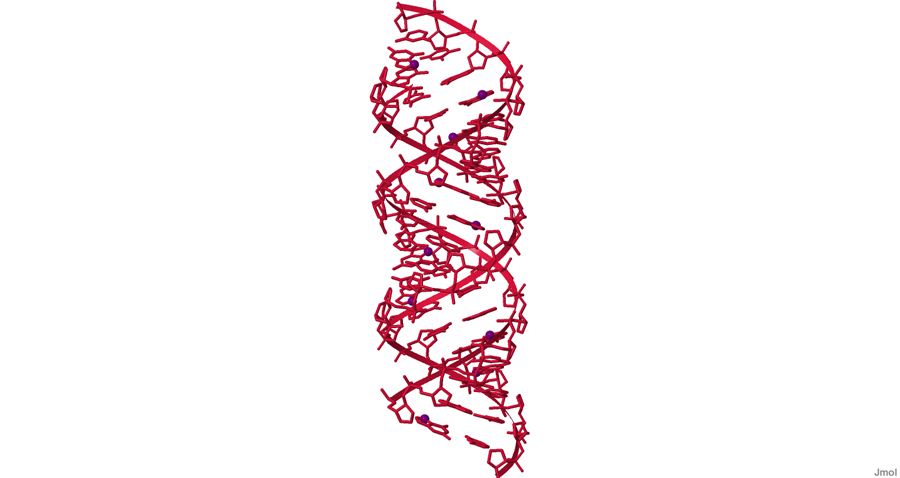
Рисунок 4. Атом N7 во всех гуанинах выделен фиолетовым цветом. Изображение получено с помощью Jmol.
Следующим заданием была работа с выданными ДНК и РНК. Мне достались белки с идентификаторами PDB 1U0B и 1BDT. Нужно было проверить нуклеиновую составляющую этих белков на разрывы. Исходя из рисунков 5 и 6 можно заметить, что в данных структурах разрывов не наблюдается.
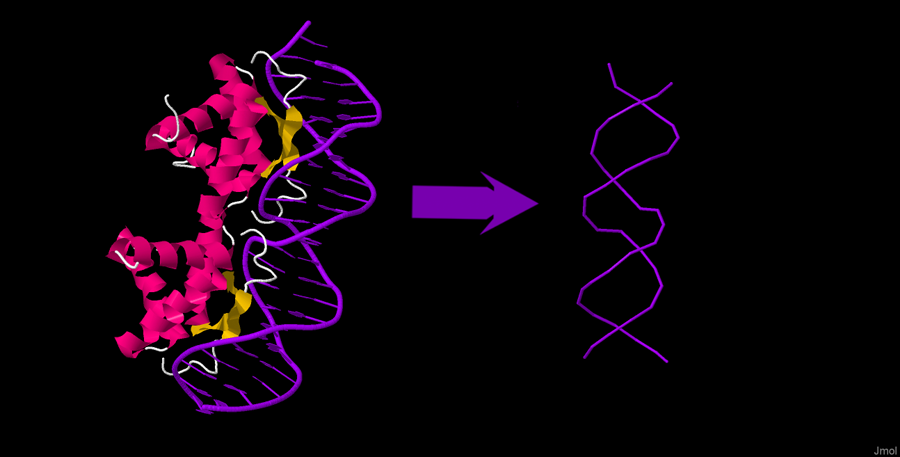
Рисунок 5. ДНК-беловый комплекс 1BDT. Справа изображена изолированная цепь ДНК. Изображение получено с помощью Jmol.
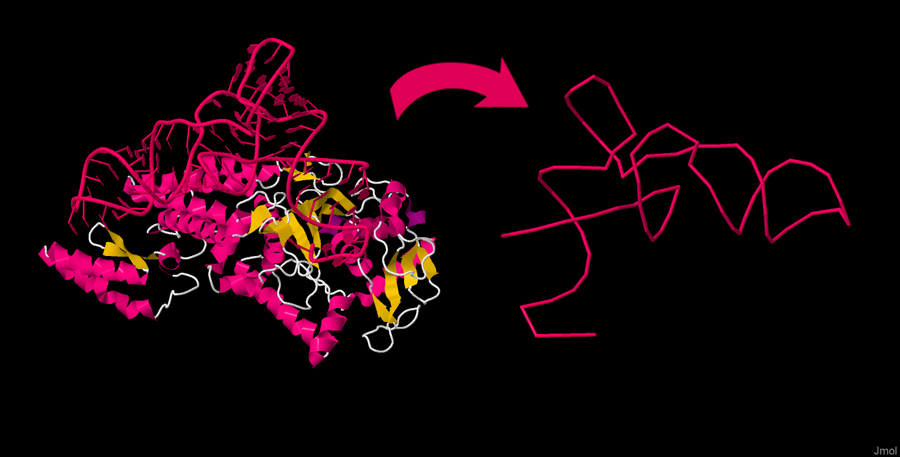
Рисунок 6. РНК-беловый комплекс 1U0B. Справа изображена изолированная цепь РНК. Изображение получено с помощью Jmol.
Далее нам нужно было научиться находить малые и большие бороздки ДНК и определить, какие атомы в выданных нам азотистых основаниях куда ориентированы. Мне достался аденин, поэтому я могу проверить его расположение только в А- и В-формах, так как в Z-форме его нет.
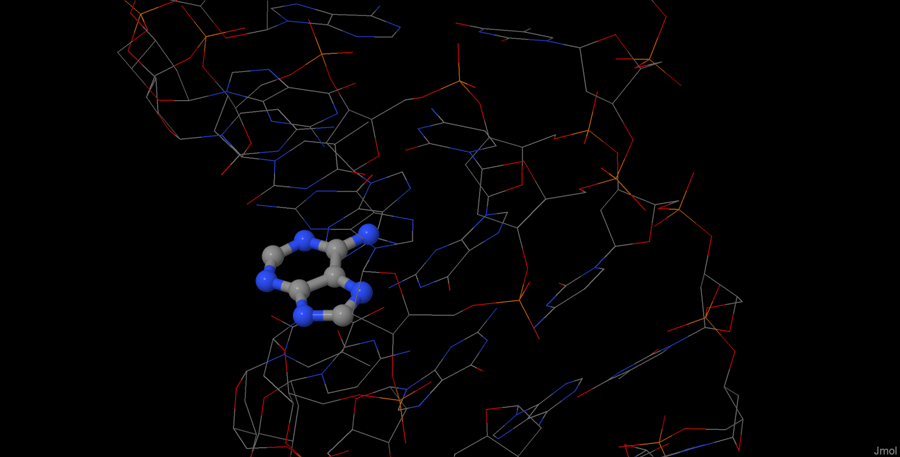
Рисунок 7. Аденин А10 (выделен окраской cpk) в А-форме ДНК. Изображение получено с помощью Jmol.
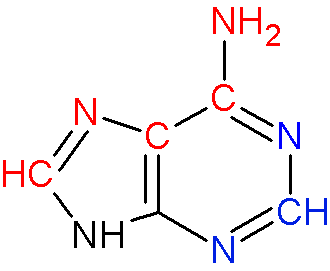
Рисунок 8. Структурная формула аденина. Красным выделены атомы, обращённые к большой бороздке, синим - к малой. Изображение получено с помощью ChemSketch.
Из рисунка 7 понятно, что N6, C6, C5, N7 и С8 повёрнуты в сторону большой бороздки, в то время как N1, C2, N3 в сторону малой. Остальные атомы расположены примерно между двумя бороздками. На рисунка 9 изображён тот же аденин 10, но в В-структуре. Можно заметить, что ориентировка атомов не поменялась.
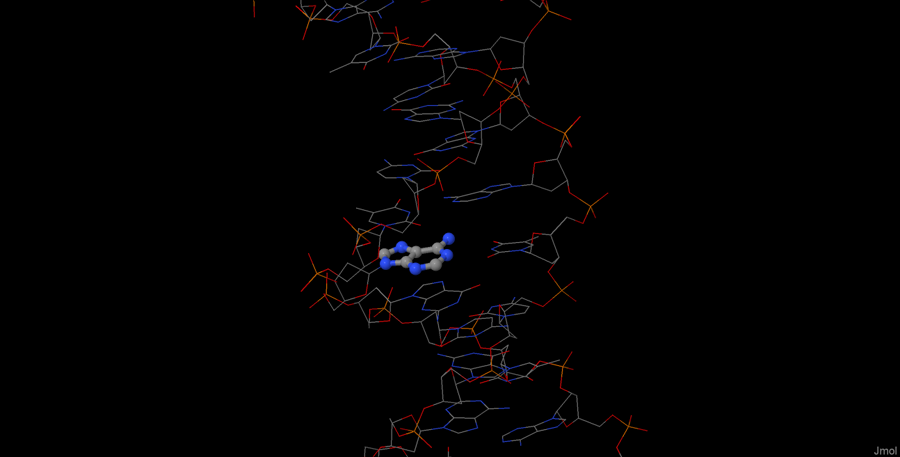
Рисунок 9. Аденин А10 (выделен окраской cpk) в А-форме ДНК. Изображение получено с помощью Jmol.
Затем мы проводили сравнительную характеристику трёх типов ДНК. Результаты представлены на рисунках 10-12 и в таблице 1.
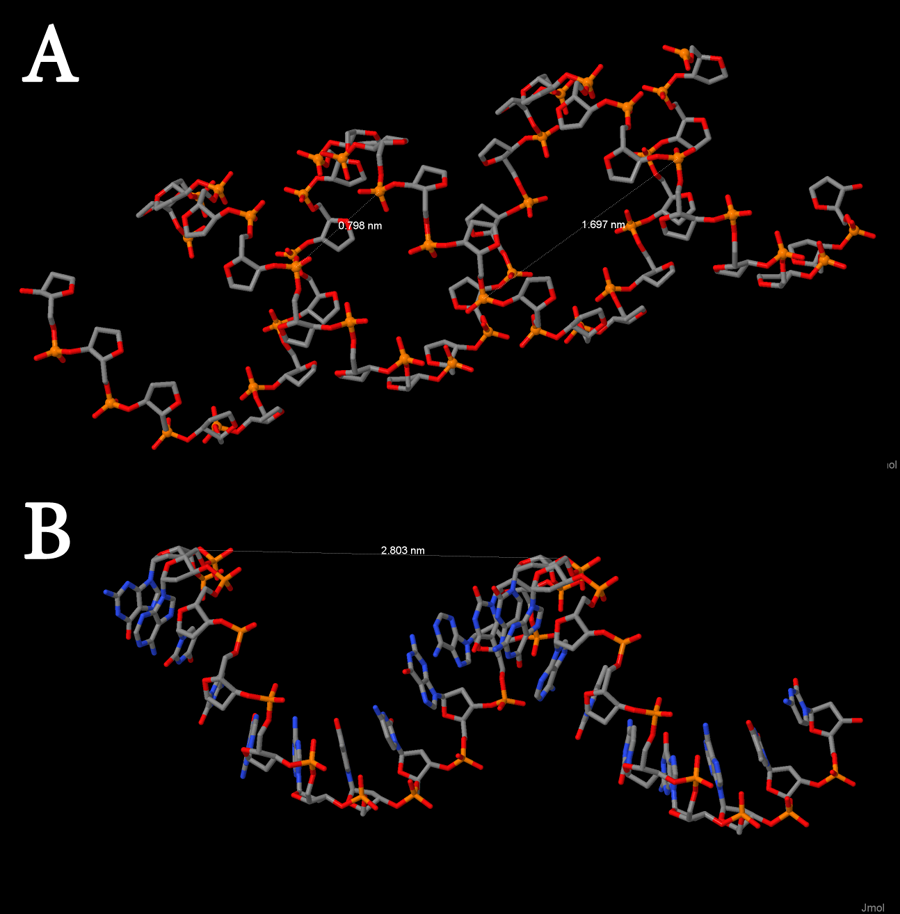
Рисунок 10. А-форма ДНК: (А) - ширина малой и большой бороздок, (В) - шаг спирали. Изображение получено с помощью Jmol.
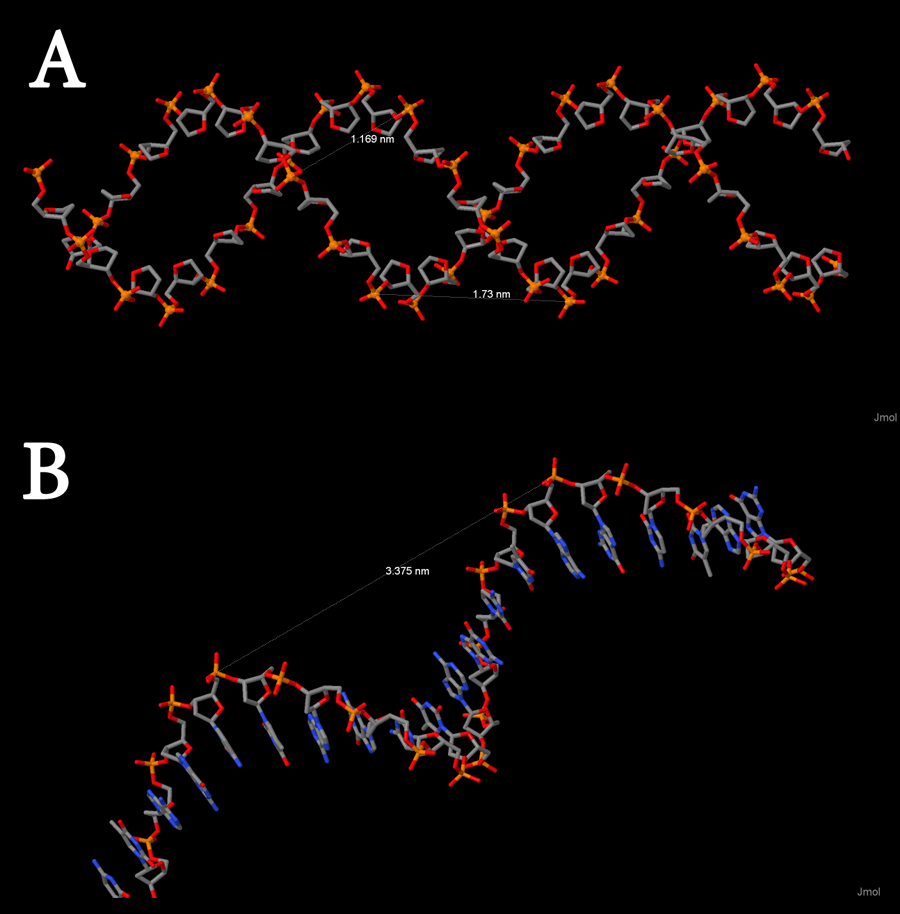
Рисунок 11. B-форма ДНК: (А) - ширина малой и большой бороздок, (В) - шаг спирали. Изображение получено с помощью Jmol.
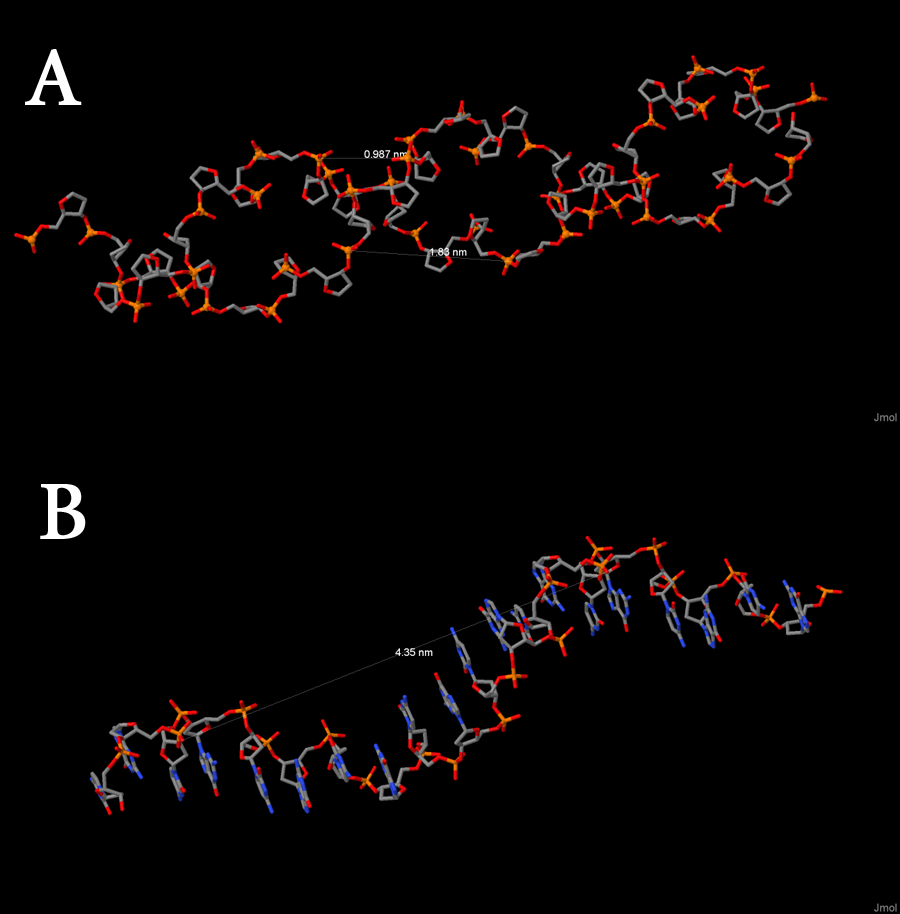
Рисунок 12. Z-форма ДНК: (А) - ширина малой и большой бороздок, (В) - шаг спирали. Изображение получено с помощью Jmol.
Таблица 1. Характеристики цепей ДНК различных форм.
| Спиральный параметр | А-форма | В-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (нм) | 2.803 | 3.375 | 4.35 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 1.697 | 1.73 | 1.83 |
| Ширина малой бороздки | 0.798 | 1.169 | 0.987 |
Следующим заданием было определение торсионных углов с помощью Jmol и сравнение их с данными в презентации. Результаты моего сравнения представлены в таблице 2.
Таблица 2. Значения торсионных углов.
| Форма | α | β | γ | δ | ε | ζ | χ |
| А | 64,1 | 174,8 | 41,7 | 79,1 | 100,4 | -75,1 | -157,2 |
| В | 85,9 | 136,3 | 31,2 | 23,1 | 105,8 | -44,7 | -98,3 |
С данными из презентации есть существенные различия. Возможно, это произошло из-за того, что данная структура лишь модель, построенная для идеальных условий. А углы из презентации - реально померенные данные.
Следующим пунктом было определение торсионных углов с помощью пакета 3DNA. Для работы с этим пакетом нужно было перевести все pdb-файлы в старый формат (с помощью команды remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb, с которым дальше я стала работать. При помощи команд find_pair и analyze я получила значения торсионных углов, которые представлены в таблице 3. Самым откланяющимся нуклеотидом был А5.
Таблица 2. Значения торсионных углов.
| Тип кислоты | α | β | γ | δ | ε | ζ | χ |
| ДНК | -32,28 | 156,88 | 35,82 | 137,32 | -97,44 | -107,14 | -114,32 |
| РНК | -75 | -170 | 50 | 80 | -150 | -70 | -170 |
тРНК характеризуется наличием "шпилек", которые образуют стебли. И в этом задаии нам нужно было их найти. Результаты моего поиска показаны на рисунке 13.
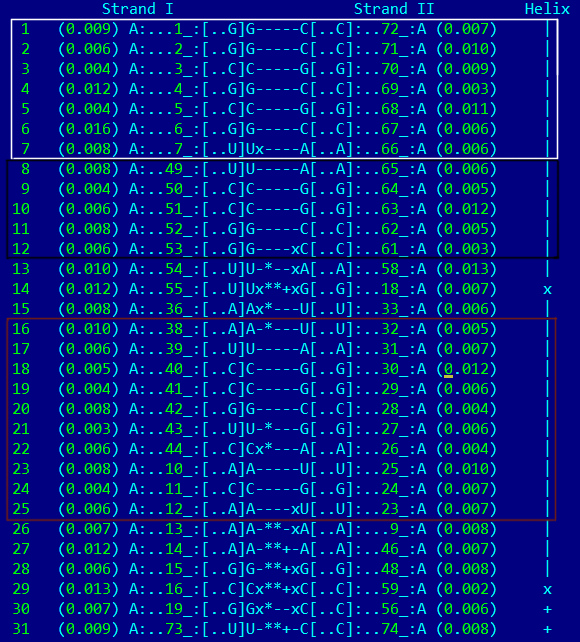
Рисунок 13. Стебли в тРНК (выделены цветными прямоугольниками).
Следующим заданием был поиск неканонических пар в тРНК. Все найденные пары представлены на рисунке 14.
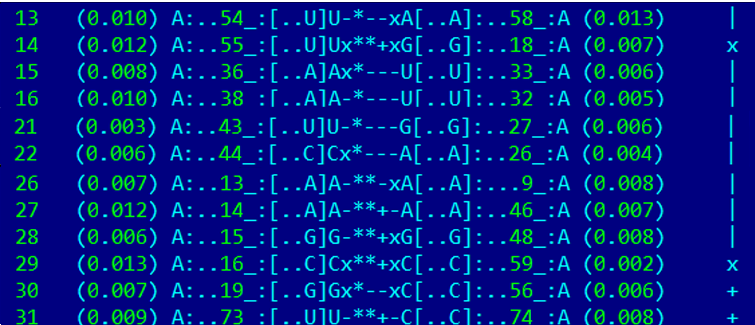
Рисунок 14. Неканонические пары в тРНК.
Также в структура тРНК есть так называемые дополнительные связи, стабилизирующие третичную структуру, - это пары нуклеотидов, соединяющие стебли. Эти пары нуклеотидов данной тРНК представлены на рисунке 15.
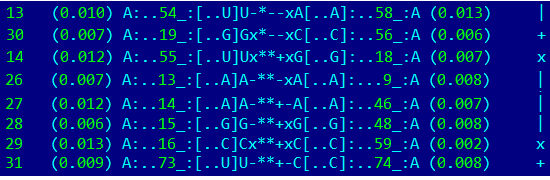
Рисунок 15. Пары нуклеотидов-дополнительные связи в тРНК.
Последним заданием был поиск стекинг-взаимодействий. Их сила определяется площадью перекрывания. На рисунке 16 приведён список возможных стекинг-взаимодействий и их площади.
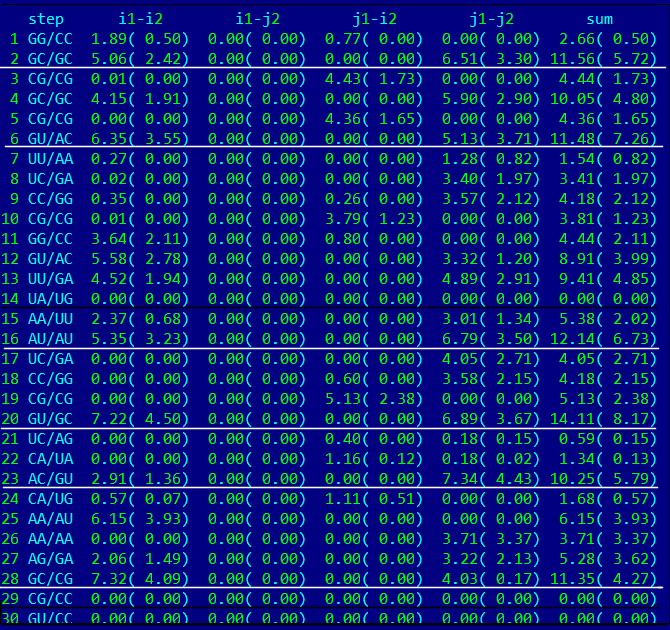
Рисунок 16. Возможные стекинг-взаимодействия. Белым подчёркнуты наиболее перекрывающиеся, чёрным - наименее.
Также были получины изображения самого максимального и минимального перекрытий (соответственно рисунки 17 и 18). Для этого использовались следующие команды: ex_str -X stacking.pdb stepX.pdb и stack2img -cdolt stepX.pdb stepX.ps.
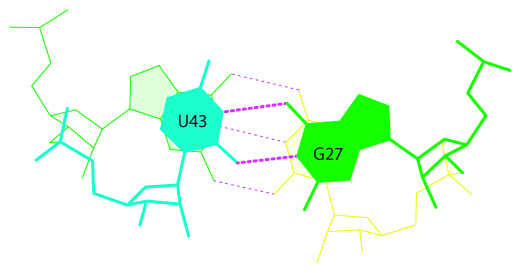
Рисунок 17. Изображение максимального перекрытия в данной тРНК (шаг 20).
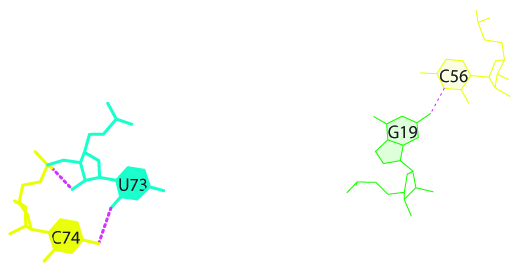
Рисунок 18. Изображение минимального перекрытия в данной тРНК (шаг 30).
Дата последнего изменения: 22.09.2014
