Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
Построение дерева по нуклеотидным последовательностям
В первом задании данного практикума нужно было построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA). Последовательности РНК были получены из базы NCBI (файл с расширением frn). Затем они были помещены в один общий файл, при этом полные названия записей были заменены на короткие обознечения. После этого было проведено выравнивание при помощи модуля Muscle в программе Jalview. Наконец, с помощью программы MEGA (метод Maximum likelihood) было построено филогенетическое дерево, которое представлено на рисунке 1.
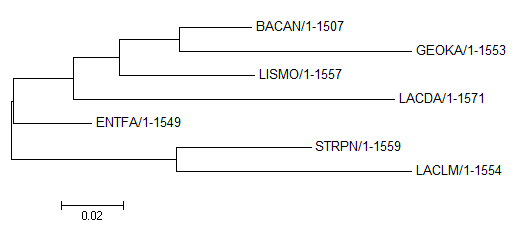
Рисунок 1. Филогенетическое дерево выбранных бактерий на основе различий в последовательностях 16S рРНК.
Если сравнить получившееся дерево с правильным, то можно заметить, что оно совпадает по топологии, но укоренение в среднюю точку произошло неверно. Однако деревья всё равно являются одинаковыми.
Построение и анализ дерева, содержащего паралоги
В этом задании практикума нужно было найти в выбранных бактериях достоверные гомологи белка CLPX_BACSU. Для этого я воспользовалась программой blastp (поиск с порогом на E-value 0,001) по протеомам отобранных вами бактерий. В итоге был найден 31 гомолог. После этого, предварительно выровняв последовательности с помощью Muscle, было построено филогенетическое дерево в программе MEGA, с использованием алгоритма Neighbor-joining (Рис.4). На нём отмечены ортологов (их разделение произошло в результате видообразования) и паралогов (синим выделены те, которые образовались в результате дупликации генов).
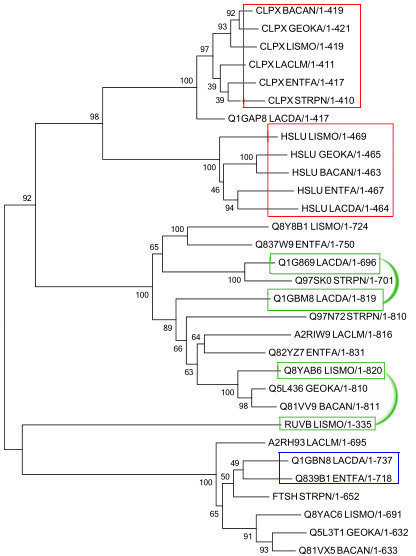
Рисунок 1. Филогенетическое дерево выбранных бактерий на основе гомологов белка CLPX_BACSU. Красные рамки - пример ортологов, синяя и зелёные - паралогов.
Дата последнего изменения: 22.05.2015
