Построение и визуализация электронной плотности (ЭП)
Для выполнения этого задания была выбрана структура белка предполагаемая эндо-1,4-бета-глюконаза - YSDC_BACSU (PDB ID - 1VHE, на сервере EDS - VHE). Разрешение структуры составляет 1.90 Å. Также для данного белка были найдены структурные гомологи со следующими параметрами (при помощи сервиса PDBeFold): с RMSD от 0.8 до 2.5 ангстрем и N_align от 50% числа а.о. входного белка до 90% (представлены на таблице 1).
Таблица 1. Отобранные структурные гомологи белка YSDC_BACSU.
| Название | PDB code | RMSD | N_align |
|---|---|---|---|
| CRYSTAL STRUCTURE OF AN ARCHAEAL AMINOPEPTIDASE | 1xfo:B | 1.01 | 327 |
| DE NOVO CRYSTAL STRUCTURE OF THE PYROCOCCUS FURIOSUS TET3 AMINOPEPTIDASE | 4x8i:A | 1.38 | 324 |
| CRYSTAL STRUCTURE OF PROTEIN OF UNKNOWN FUNCTION (POSSIBLE AMINOPEPTIDASE) S2589 FROM SHIGELLA FLEXNERI 2A STR. 2457T | 1ylo:B | 1.39 | 326 |
| CRYSTAL STRUCTURE OF THE M42 AMINOPEPTIDASE TMPEP1050 FROM THERMOTOGA MARITIMA | 4p6y:D | 1.19 | 320 |
Построение изображения ЭП вокруг полипептидной цепи
На рисунках ниже показаны изображения электронной плотности для данного белка с различными уровнями подрезки 0.5, 1, 1.5, 2 и различными значениями параметра carve: 1.5, 2 и 5.
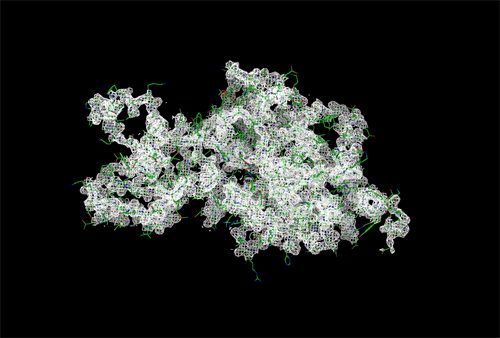
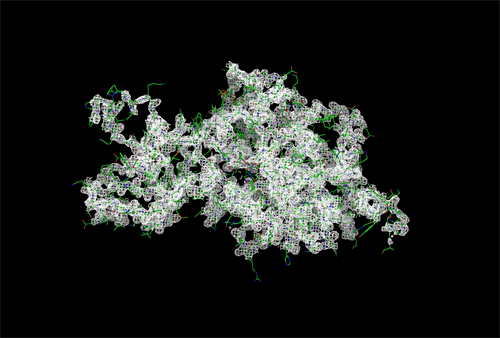
Рисунок 1. Изображение ЭП вокруг полипептидной цепи level = 1.0 σ, carve = 2 Å. Рисунок 2. Изображение ЭП вокруг полипептидной цепи level = 1.5 σ, carve = 2 Å.
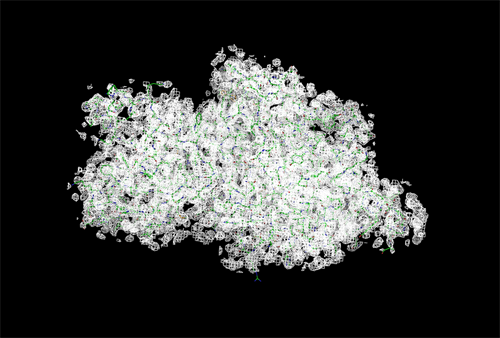
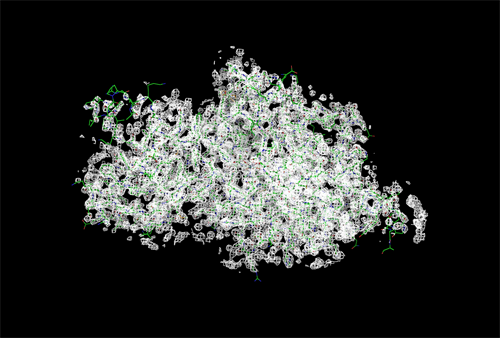
Рисунок 3. Изображение ЭП вокруг полипептидной цепи level = 1.0 σ, carve = 5 Å. Рисунок 4. Изображение ЭП вокруг полипептидной цепи level = 2 σ, carve = 5 Å.
Рисунок 5. Изображение ЭП вокруг полипептидной цепи level = 0.5 σ
Можно заметить, что при увеличении уровня подрезки количество атомов полипептидной цепи, покрытых электронной плотностью, немного уменьшается, но белок всё равно хорошо покрывается картой ЭП.
Построение изображение ЭП вокруг трех аминокислотных остатков
Для построения изображения электронной плотности вокруг трёх аминокислотных остатков были выбраны лизин 12, аспартат 13 и лейцин 14. Изображения построены для трёх уровней подрезки: 1.0, 2.0 и 3.0 и представлены на рисунках ниже.
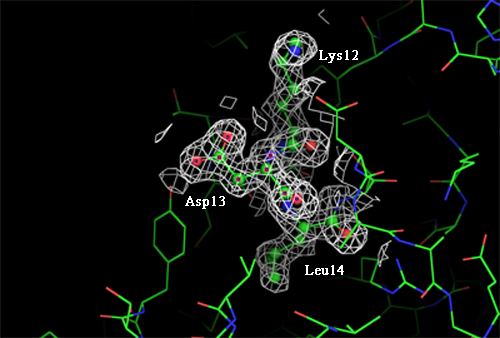
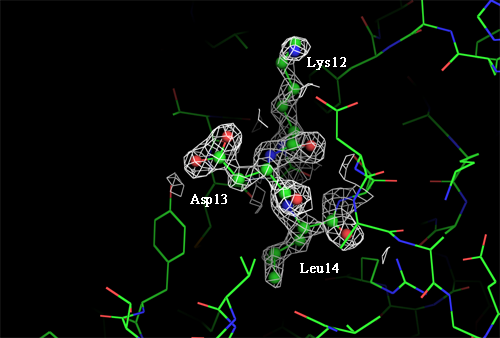
Рисунок 6. Изображение ЭП вокруг трёх а.о. level = 1.0 σ, carve = 2.5 Å. Рисунок 7. Изображение ЭП вокруг трёх а.о. level = 2.0 σ, carve = 2.5 Å.
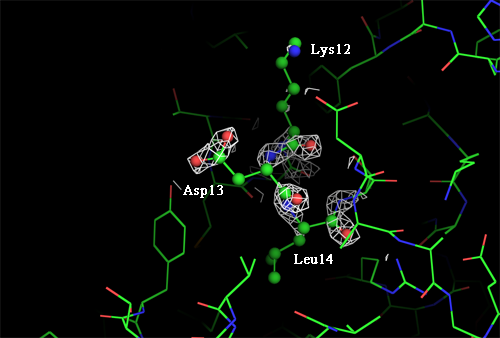
Рисунок 8. Изображение ЭП вокруг трёх а.о. level = 3.0 σ carve = 2.5
Видно, что на первых двух уровнях подрезки все аминокислоты вписываются в электронную плотность очень хорошо, но на третьем покрытие стало гораздо хуже и покрытыми остались буквально по 3-4 атома у каждой аминокислоты.
Дата последнего изменения: 25.12.2016
