Совмещение структур
Для выполнения задания был выбран белок 1VHE. С помощью PDBeFold было найдено 4 гомолога: 1xfo:A, 1ylo:A, 3isx:A, 3cpx:A (критерии: rmsd между 0.8 и 2.5, и длинной выравнивания более 50%). Для этих белков было построено множественное выравнивание структур (изображено на рисунке 1).
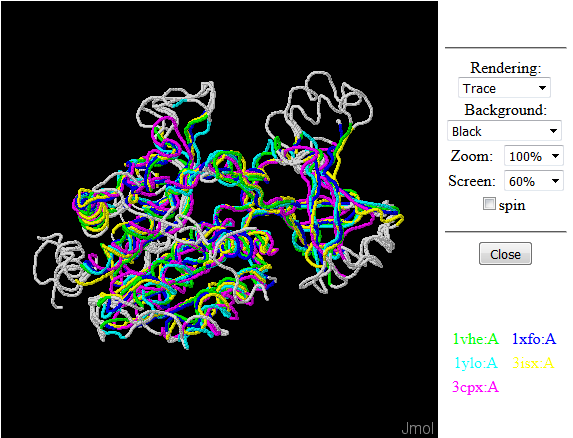
Рисунок 1. Множественное выравнивание структурных гомологов 1VHE.
Также было получено выравнивание, построенное по алгоритму Tcoffee. Оба выравнивния полностью приведены на рисунках 2 и 3, причем выровненными считаются только большие буквы.

Рисунок 2. Выравнивание по совмещению структур, окраска Clustalx, above identity threshold 70%.

Рисунок 3. Выравнивание, построенное Tcoffee, окраска Clustalx, above identity threshold 70%.
В целом оба выравнивания похожи, и небольшое различие заключается в промежутке с 40 по 60 позиции. Последовательность 3CPX что в первом, что во втором случае имеет целую аминокислотную вставку, только выравние tcoffee собрала все гэпы в одном месте, а структурное выравнивание их раскидало немного. Но так как по рисунку 1 кажется, что последовательности совместились хорошо, то, возможно, структурное выравние чуточку лучше.
Для поиска белков, для которых гибкое структурное выравнивание включает существенно больше остатков, чем жесткое, был использован сервер POSA. В разделе примеров был выбран следующий: 5 белков с 2-RRM доменом в комплексе с РНК. На рисунках 4 и 5 представлены гибкое и жёсткое выравнивания соотвественно и из них видно, что гибкое выравнивание получилось лучше. В нем 144 общие аминокислоты, в жёстком - 69.
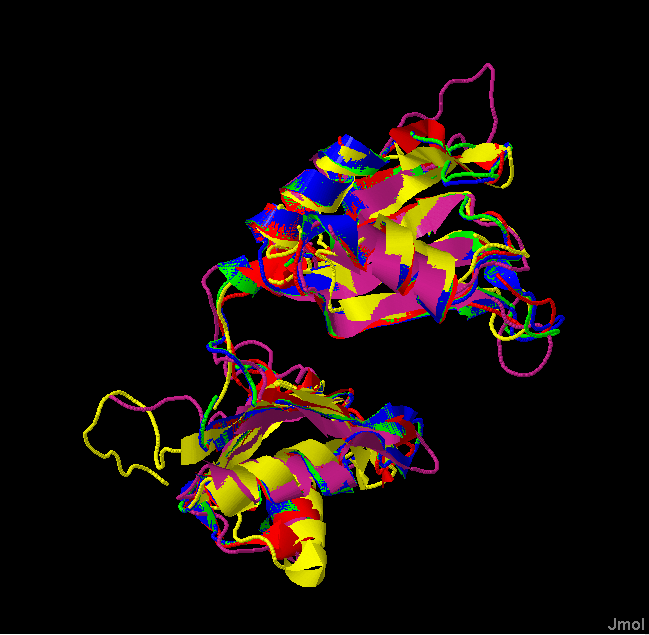
Рисунок 4. Гибкое структурное выравнивание.
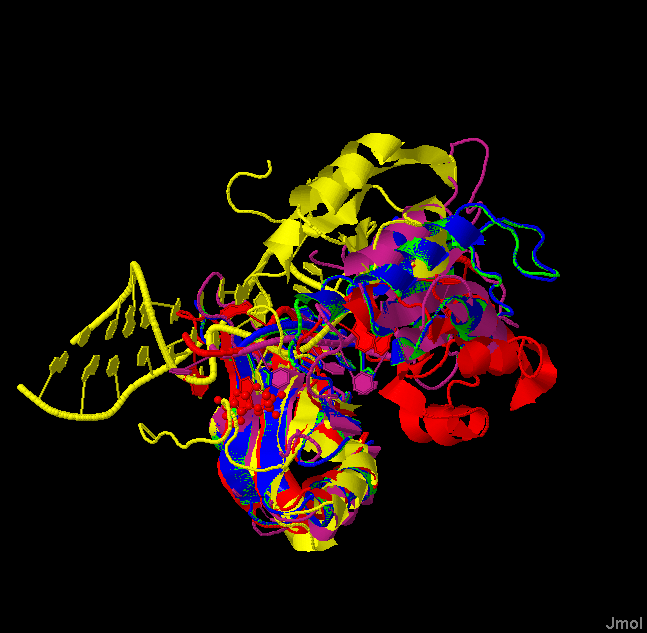
Рисунок 5. Жёсткое структурное выравнивание.
Дата последнего изменения: 25.12.2016
