Для работы я выбрала домен с идентификатором PF03066 (из прошлого практикума). Далее я задала следующий поисковый запрос в Uniprot:
database:(type:pfam id:PF03066) taxonomy:"Vertebrata [7742]" AND reviewed:yes
Нашлось 15 белков, из которых я построила выравнивание. Проект с выравниванием, а на рисунке 1 представлен фрагмент этого выравнивания.
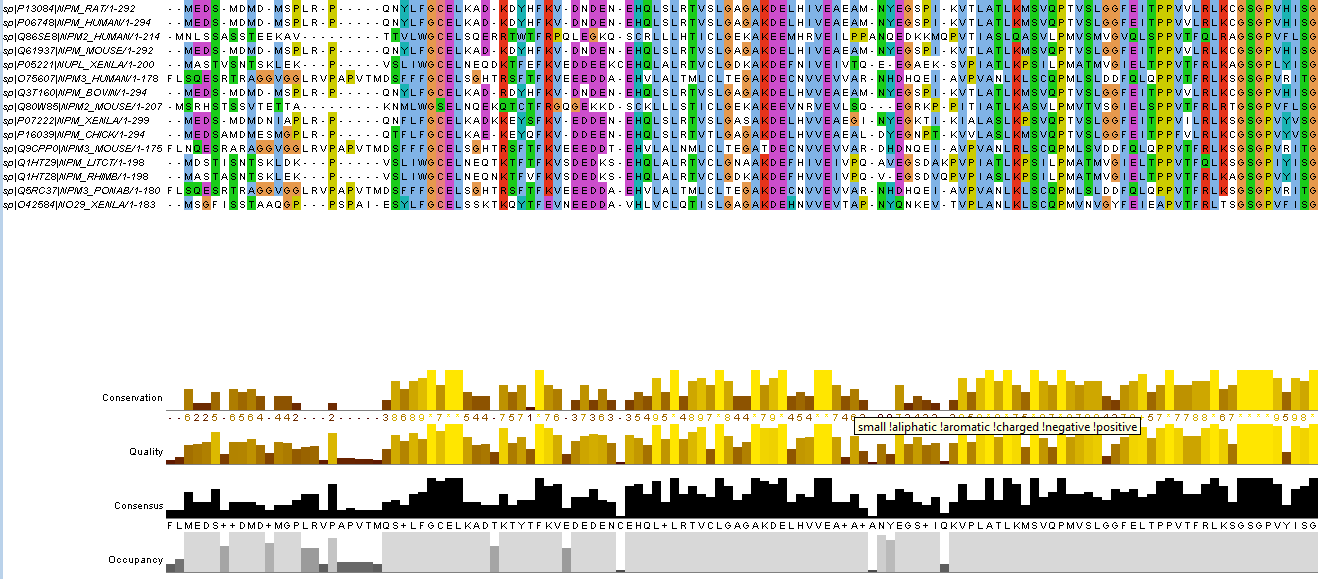
Рисунок 1. Фрагмент полученного выравнивания
Далее я создала HMM-профиль по полученному выравниванию при помощи команды:
hmm2build -g profile.out npmal.mfa
Полученный профиль был откалиброван:
hmm2calibrate profile.out
Поиск по профилю осуществлялся с помощью следующей команды:
hmm2search --domE 1000 --domT -50 profile.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > find.out
Для осуществления более расширенного поиска порог E-value убирался, а порог веса снижался до 50.
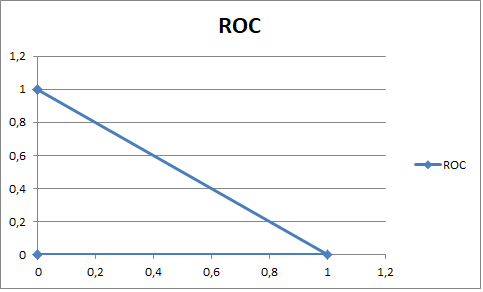
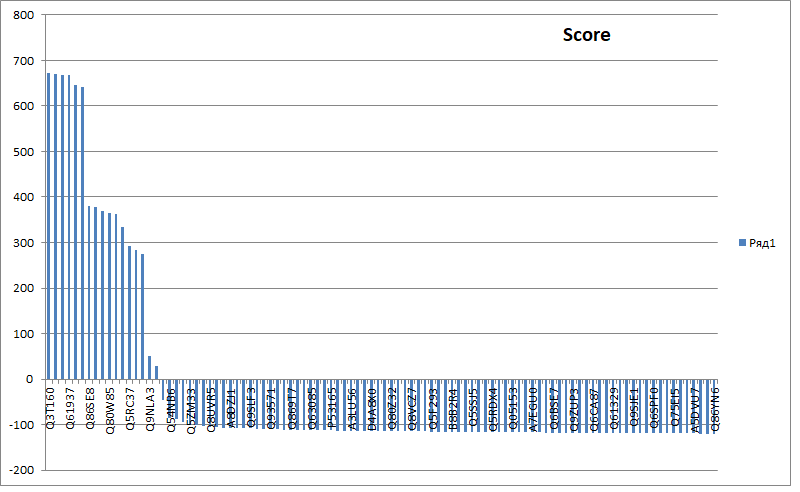
Была постороена гистограмма находок и их оценок (score).

Рис. 2. Гистограмма оценок.
В качестве порогового значения было выбрано 380,7. Excel-файл с вычислениями, файл с результатами поиска
Как можно видеть, поиск был произведён очень качественно: все предсказанные члены семейства действительно в него входят.