Миниобзор по Leptospira tipperaryensis
Зюбина Елизавета, ФББ, 1 курс
Ключевые слова: Leptospira tipperaryensis, геном, обзор.
1. Введение:
Leptospira tipperaryensis – патогенная бактерия рода Leptospira, вызывающее лептоспироз. Лептоспироз – опасное зоонозное заболевание, распространенное во всех частях мира [1].
Род Leptospira очень гетерогенен и генетически отличается от других бактерий. Представители этого рода имели форму спирали длиной от 9 до 14 мкм, диаметром ~ 0,2 мкм и длиной волны от 0,6 до 0,9 мкм [2]. Сейчас в нем выделяют 22 вида и более 300 сероваров. Для этого рода характерен открытый пангеном, а также высокая скорость горизонтального переноса генов [3]. Филогенетический анализ, основанный на 16s рРНК и полногеномных последовательностях, показал, что в роде можно выделить 3 монофилетические группы: «сапрофиты», «патогены» и «промежуточные продукты». «Сапрофиты» - виды не являющиеся патогенными для человека и животных, «Патогены» - опасные для жизни виды. «Промежуточные продукты» - виды, способные заражать людей и животных, но не приводящих к смерти [4]. Патогены имеют больший размер генома, по сравнению с 2 другими группами. Однако процентное содержание гуанина и цитозина меньше, нежели в группе «промежуточные звенья» и приблизительно равно содержанию группы «сапрофитов» [5].
Leptospira tipperaryensis относится к группе патогенных бактерий. Раннее этот штамм ошибочно относили к L. Altonii [6].
2. Материалы и методы:
Для этого обзора использовалась таблица особенностей генома бактерии, а также геном бактерии [7]. Для их обработки были использованы Microsoft Excel 2010 [8] и Visual Studio Code. Для статистической обработки использовался тест хи-квадрат с доверительным уровнем - 0,05.
3. Результаты:
3.1. Общие сведения
Генома бактерии Leptospira tipperaryensis представлен двумя хромосомами: первая состоит из 4105414 п.н, вторая - из 486474 п.н.
В геноме преобладает AT - пары. Процентное содержание GC - пар в первой хромосоме - 43,42%, во второй - 42,37%, что свидетельствует о низкой устойчивости молекулы к денатурации.
3.2. Белки протеома
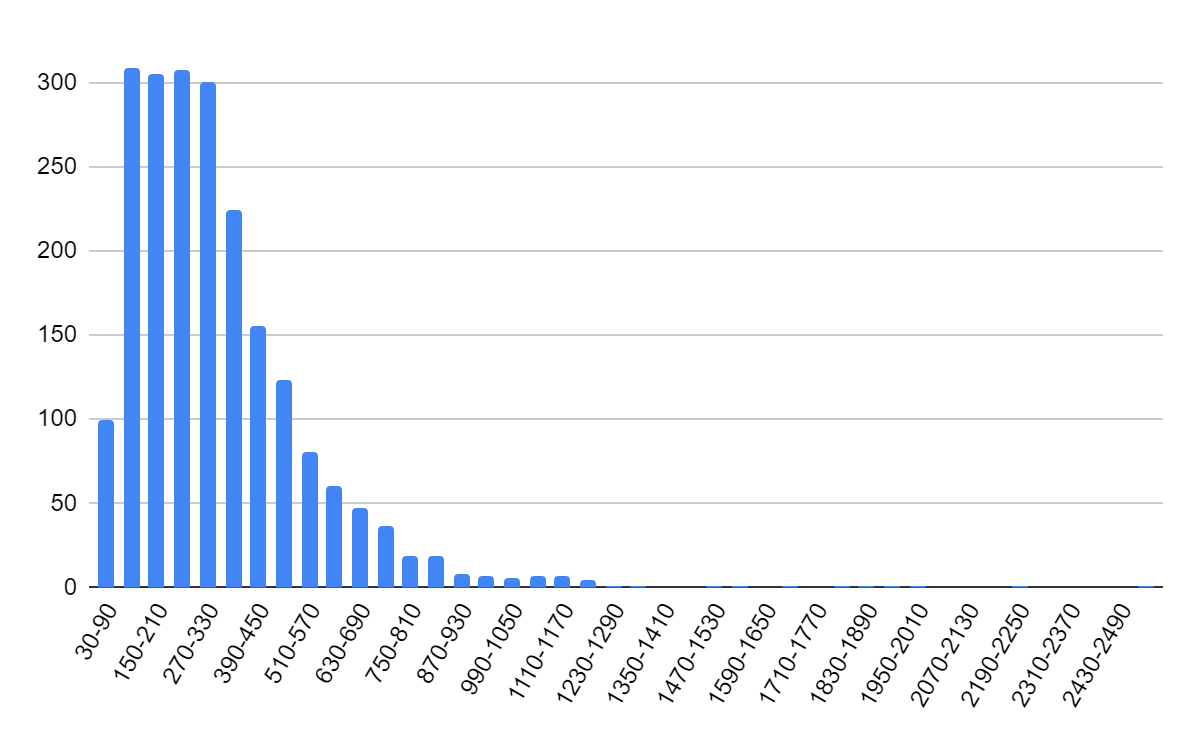
В геноме Leptospira tipperaryensis закодировано 4055 белков. Медиана длин белков равна 281. Максимальная длина белка - 2588 аминокислота, минимальная - 37. Распределение длин белков представлено на диаграмме (рис.1)
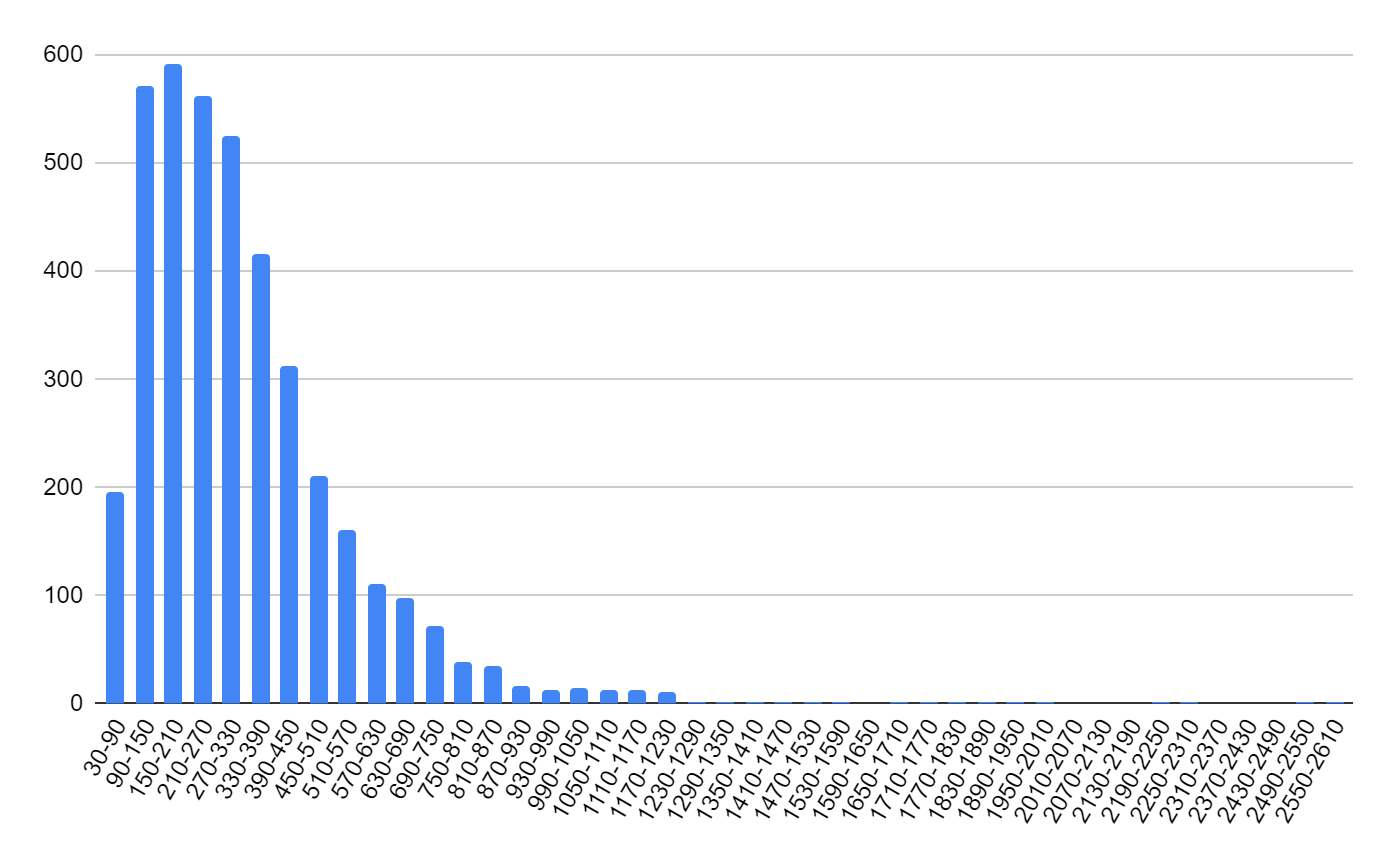
На первой хромосоме закодированы 3439 белков, максимальная длина которых - 2588 аминокислот, минимальная - 37. Распределение длин белков на первой хромосоме представлено на диаграмме (рис.2).
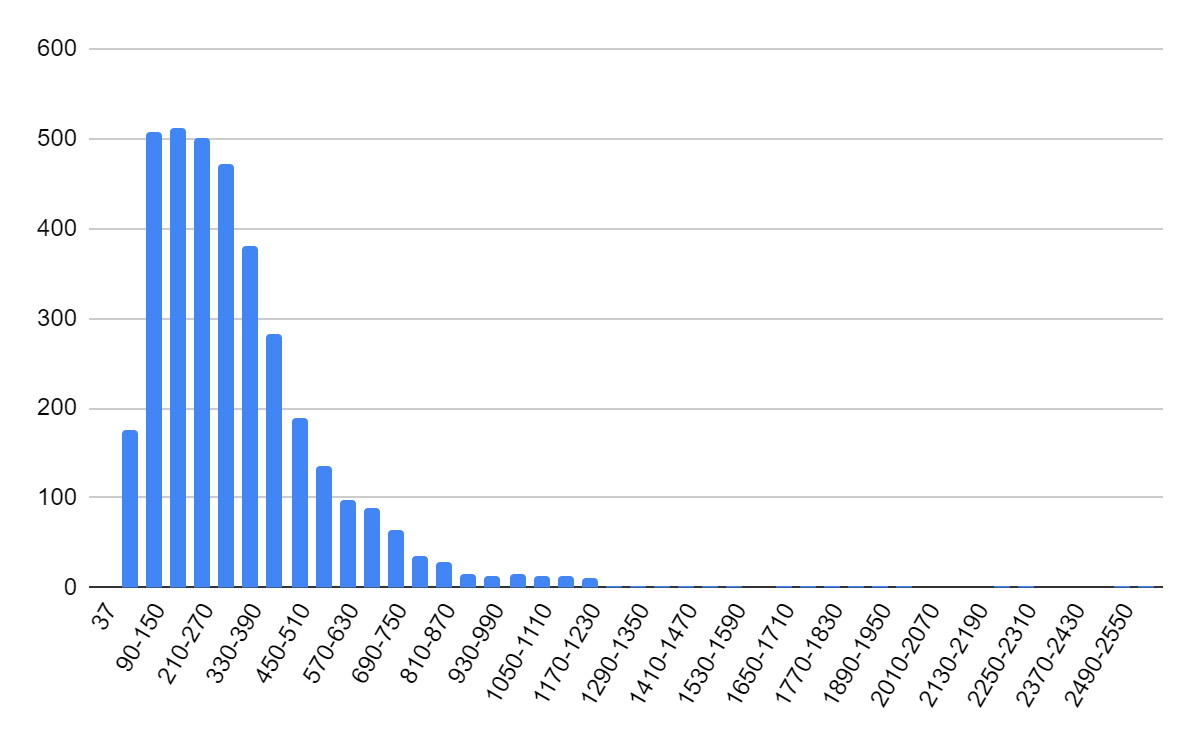
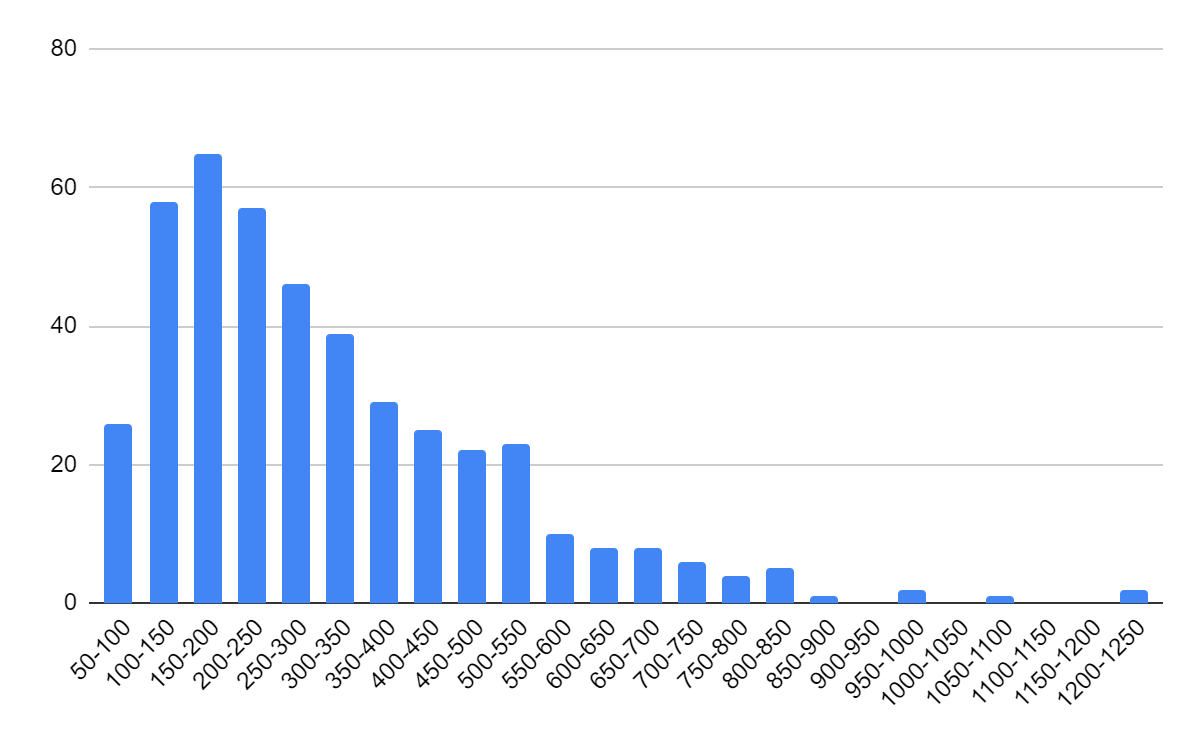
На второй хромосоме закодировано 446 белков, максимальная длина которых - 1246 аминокислот, минимальная - 54. Распределение длин белков представлено на диаграмме (рис.3).
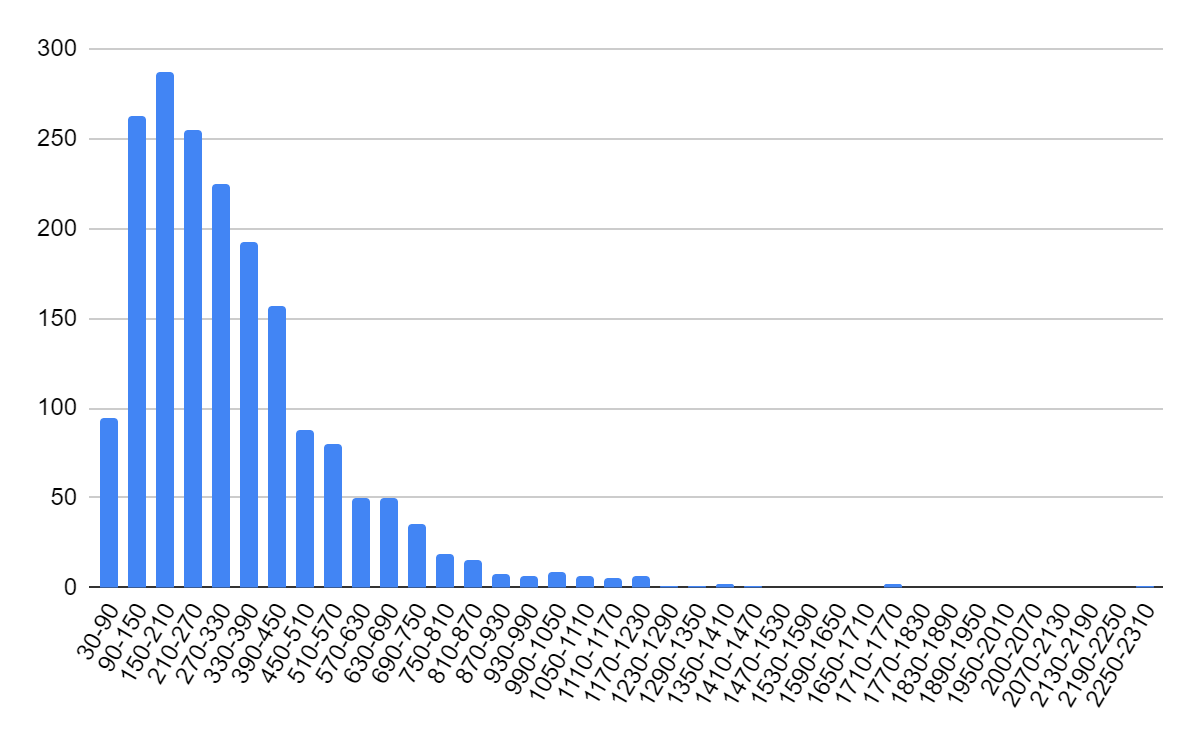
На прямой цепи ДНК было закодировано 2109 белков, на обратной - 1839. Белки распределены по двум цепям не равновероятно (Предположим обратное, тогда доверительный уровень - 0,05, критическое значение - 3,8, значение хи-квадрата - 18,46. гипотеза отвергнута.). Распределение длин белков на прямой и обратной цепи показаны на диаграммах (Рис. 4,5).
Среди закодированных белков есть рибосомальные, транспортные и гипотетические. Их количество и доля представлены в таблице 1.
В результате работы в течение 1 семестра, я сделала миниобзор по бактерии Leptospira tipperaryensis . Миниобзор и все сопутсвующие материалы можно найти здесь .
| Белки | Рибосомальные | Транспортные | Гипотетические |
|---|---|---|---|
| Число | 57 | 157 | 915 |
| Процентное содержание | 1,41% | 3,87% | 22,56% |
3.3. РНК
Всего в геноме бактерии 46 генов кодирующих РНК. У Leptospira tipperaryensis 4 типа РНК: транспортная, рибосомальная, некодирующая и транспортно-матричная. Их количество представлено в таблице 2.
| РНК | Рибосомальная | Транспортная | Некодирующая | Траснпортно-матричная |
|---|---|---|---|---|
| Число | 6 | 38 | 1 | 1 |
| Процентное содержание | 13,04% | 82,61% | 2,17% | 2,17% |
Нуклеотидный состав хромосом
Нуклеотидный состав хромосом представлен в таблице 3. Предположим, что нуклеотиды А и Т на одной цепи распределяются равномерно (доверительный уровень - 0,05, критическое значение - 3,8) Значение хи-квадрата для первой хромосомы - 10,171, для второй - 81,974. Значения хи-квадрата превышают заданный уровень, значит гипотеза неверна и распределение А и Т происходит неравномерно. Проверим эту гипотезу для G и C нуклеотидов. Значения хи-квадрата для первой хромосомы - 9,27, для второй - 37,712. Значения превышают заданный уровень, значит распределение G и C неравномерно.
| Нуклеотид | Аденин | Тимин | Гуанин | Цитозин |
|---|---|---|---|---|
| Хромосома 1 | 1184230 н. | 1179327 н. | 872938 н. | 868919 н. |
| Процентное содержание | 28,85% | 28,73% | 21,26% | 21,17% |
| Хромосома 2 | 142578 н. | 137784 н. | 104450 н. | 101662 н. |
| Процентное содержание | 29,31% | 28,32% | 21,47% | 20,90% |
4. Список литературы:
- Costa F, Hagan JE, Calcagno J, Kane M, Torgerson P, et al. Global morbidity and mortality of leptospirosis: a systematic review. PLoS Negl Trop Dis. 2015;9:e0003898. doi: 10.1371/journal.pntd.0003898.
- Vincent, A.T.; Schiettekatte, O.; Goarant, C.; Neela, V.K.; Bernet, E.; Thibeaux, R.; Ismail, N.; Mohd Khalid, M.K.N.; Amran, F.; Masuzawa , T.; et al. Revisiting the taxonomy and evolution of pathogenicity of the genus Leptospira through the prism of genomics. PLoS Negl. Trop. Dis. 2019, 13, e0007270.
- Fouts DE, Matthias MA, Adhikarla H, Adler B, Berg DE, Bulach D, et al. What Makes a Bacterial Species Pathogenic?: Comparative Genomic Analysis of the Genus Leptospira. PLoS Negl Trop Dis. 2016;10:e0004403. pmid:26890609.
- Slack AT, Khairani-Bejo S, Symonds ML, Dohnt MF, Galloway RL, et al. Leptospira kmetyi sp. nov., isolated from an environmental source in Malaysia. Int J Syst Evol Microbiol. 2009;59:705–708. doi: 10.1099/ijs.0.002766-0
- Picardeau M. Genomics, Proteomics, and Genetics of Leptospira. In: Adler B, editor. Leptospira and Leptospirosis: Springer-Verlag Berlin Heidelberg; 2015.
- Vincent, A.T.; Schiettekatte, O.; Goarant, C.; Neela, V.K.; Bernet, E.; Thibeaux, R.; Ismail, N.; Mohd Khalid, M.K.N.; Amran, F.; Masuzawa, T.; et al. Revisiting the taxonomy and evolution of pathogenicity of the genus Leptospira through the prism of genomics. PLoS Negl. Trop. Dis. 2019, 13, e0007270.
- Полный геном Leptospira tipperaryensis
- Таблица особенностей Leptospira tipperaryensis