Обзор сета генов
Начало
Мне достался набор из 35 генов. "Посмотрев его руками", могу выдать следующие соображения:
- TYR, TYRP1 - что-то связанное с тирозином
- MAOA, MAOB - моноаминоксидазы А и B, которые в основном связаны с обратным захватом нейромедиатров (дофамин, серотонин...)
- бросается в глаза набор генов ADH1A, ADH1B, ADH1C, ADH4, ADH6, ADH7 и возможно ассоциированные с ними ALDH3A1, ALDH3B1, ALDH3B2. Но о них я ничего не знаю
Небольшой вывод ожидаемого: возможно, будет какой-то метаболический путь, связанны с нейромедиаторами, поэтому по тканям мб мозг.
Gene Ontology & PANTHER
Первый взгляд
Gene Ontology - "это база" для начального обзора генов. Закинула туда общий список, сравнение со всеми генами человека (так как ваще непонятно, к какому процессу они примерно относятся итд). Для p-value критерий Фишера. Выдача в PANTHERе.
Так как генов было много (35), то вставлять сюда скрины из выдачи смысла не вижу)) Поэтому:
тут ссылка на выдачу в виде таблицы
До начала разбора результатов можно еще добавить, что вообще-то в выдаче было 38 ID. Разберемся, почему:
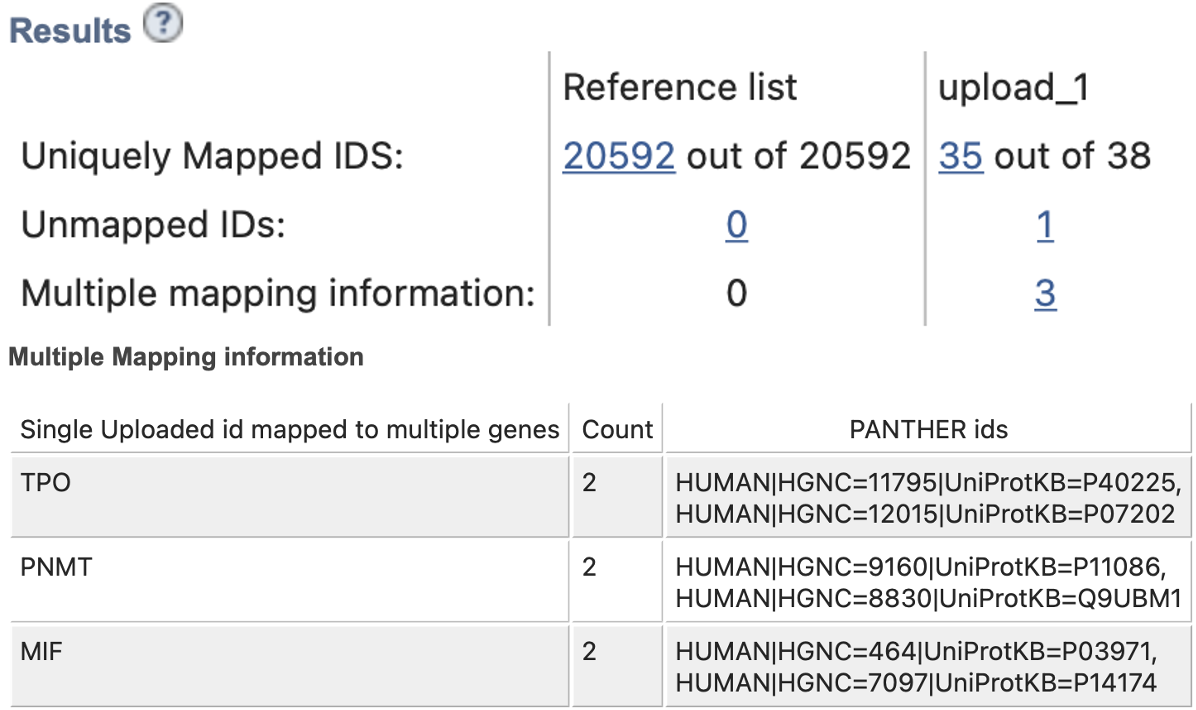
Задавшись вопросом "а почему..." я посмотрела в NCBI Genes эти гены. Но к сожалению "а почему..." меня не оставило.
- TPO: ссылкаTPO и ссылкаTHPO. Если почитать аннотации, то можно зависнуть над
полем аннотация:
Annotation information Note: TPO (Gene ID: 7173) and THPO (Gene ID: 7066) share the TPO symbol/alias in common. TPO is a widely used alternative name for thrombopoietin (THPO), which can be confused with the official symbol for TPO, thyroid peroxidase.
Насколько я понимаю, это разные реально существующие гены, которые почему-то иногда называют одним символом TPO, хотя у тромбопротеин-кодирующего гена правильное название THPO. Если это правда, то можно ожидать, что если TPO и THPO гены, вовлеченные в разные процессы, то один из ID, определенных PANTHERом будет избыточным и собьет с толку. Увы))
- PNMT: сслыкаPNMT. Для гена известно несколько альтернативных транскрипта, поэтому скорее всего оба они попали в список
- MIF: сслыкаMILF. Тоже вроде разница транскриптов, так как ген ОЧЕНЬ широко экспрессируется

Анализ
Ну, сначала можно быстренько проверить на то, а не рандомный ли набор мне дали (было бы смешно). Видно, что для каждого GO термина гены в выборке перепредставлены, что наталкивает на мысль, что все-таки взяты из какого-то конркетного процесса. P-value везде хорошее
Прочитав все GO термины, я скажу, что все они относятся к метаболическим процессам. Встречаются как и термины катаболизма, так и биосинтеза. Красиво оформлю мысли в список:
- Процесс: метаболизм
- "Подпроцессы": катаболизм и анаболизм
- Субстраты: гормоны - дофамин, норэпинифрин, эпинифрин; АК - тирозин, глутамат, аспартат, фенилаланин; жирные кислоты; этанол и формальдегид
- Самый крупный частный случай: большинство из субстратов относятся к ароматическим (бензил-содержащим) соединениям (гормоны и торизин с фенилаланином)
- Проверка моих первых слов: обратный захват гормонов и их метаболические пути, а также метаболизм тирозина
Наверное, глубже тут я копнуть не смогу. Посмотрю в других базах
STRING
Первый взгляд
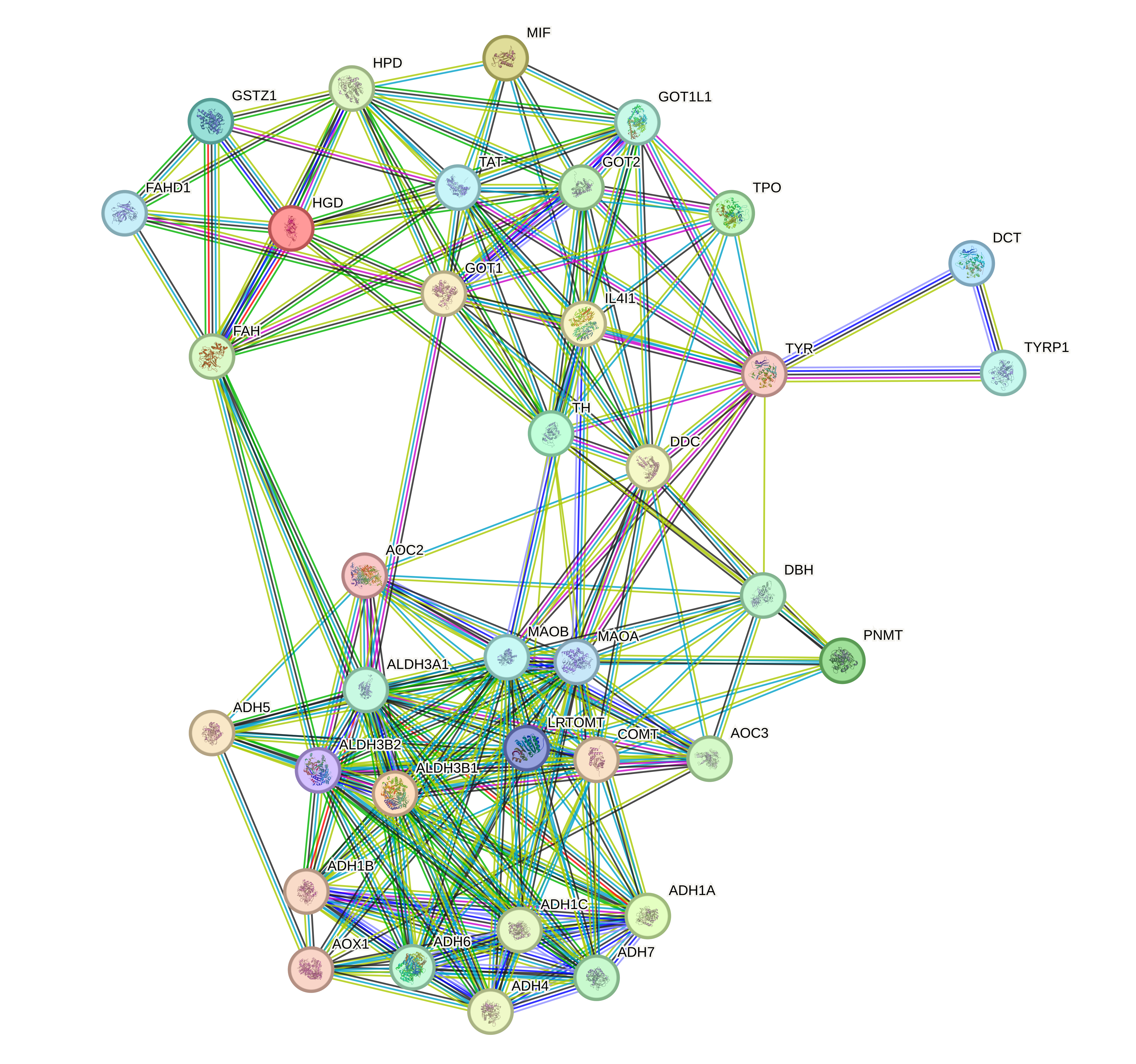
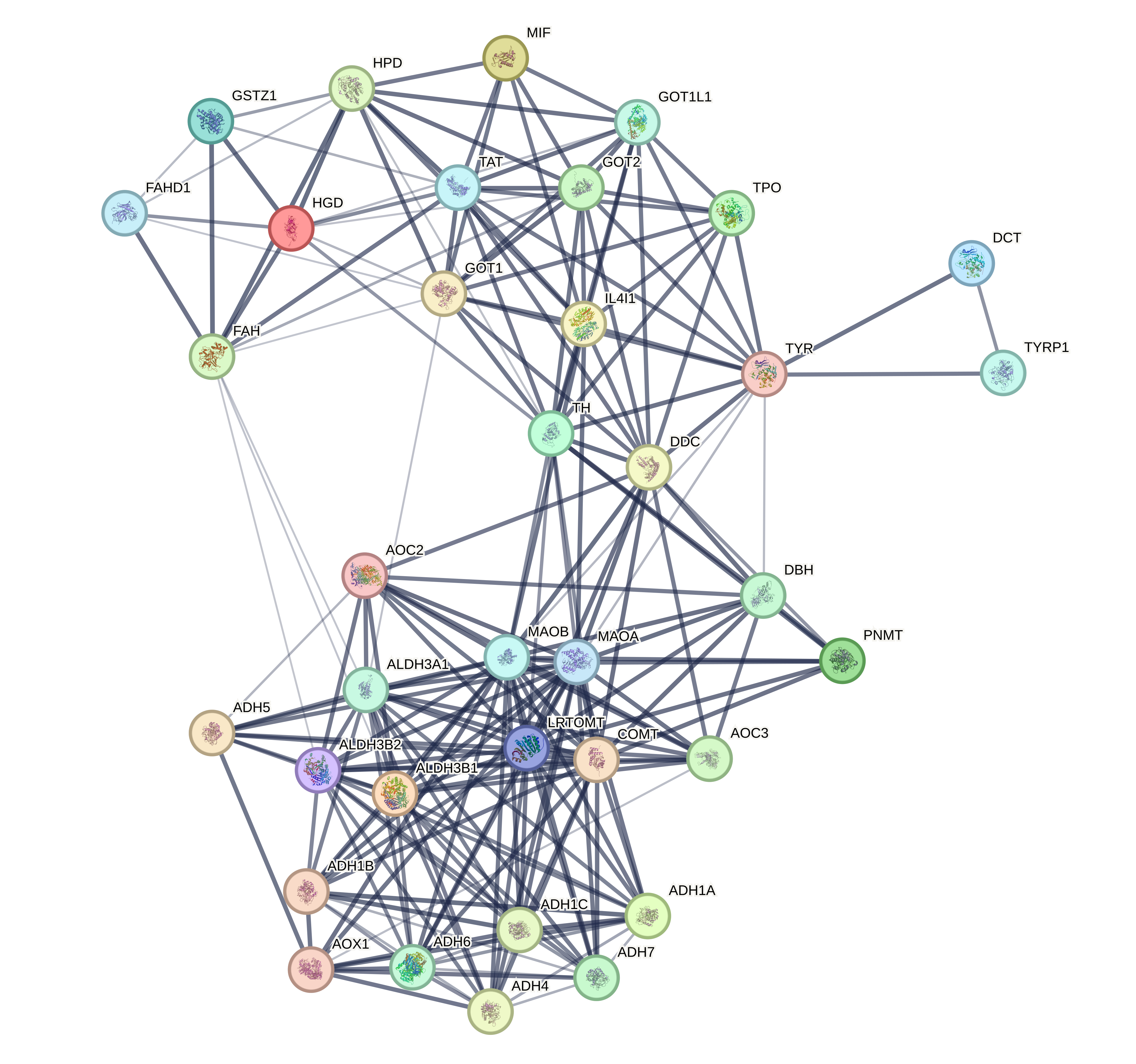

Анализ
В принципе, граф хороший, потому что изолированных вершин нет, и связь между генами добротная. Ну то, что для всех известна 3D структура продукта, было в принципе понятно из предыдущего полотна текста...
Мне еще показалось, что interactome graph предполагает именно взаимодействие между белками и комплексами белков, то есть если для работы белку нужно собраться в комплекс с какими-то другими, то лучше об этом знать. Поэтмоу я их кластеризовала по 'physical complex'. Получилось следующее:
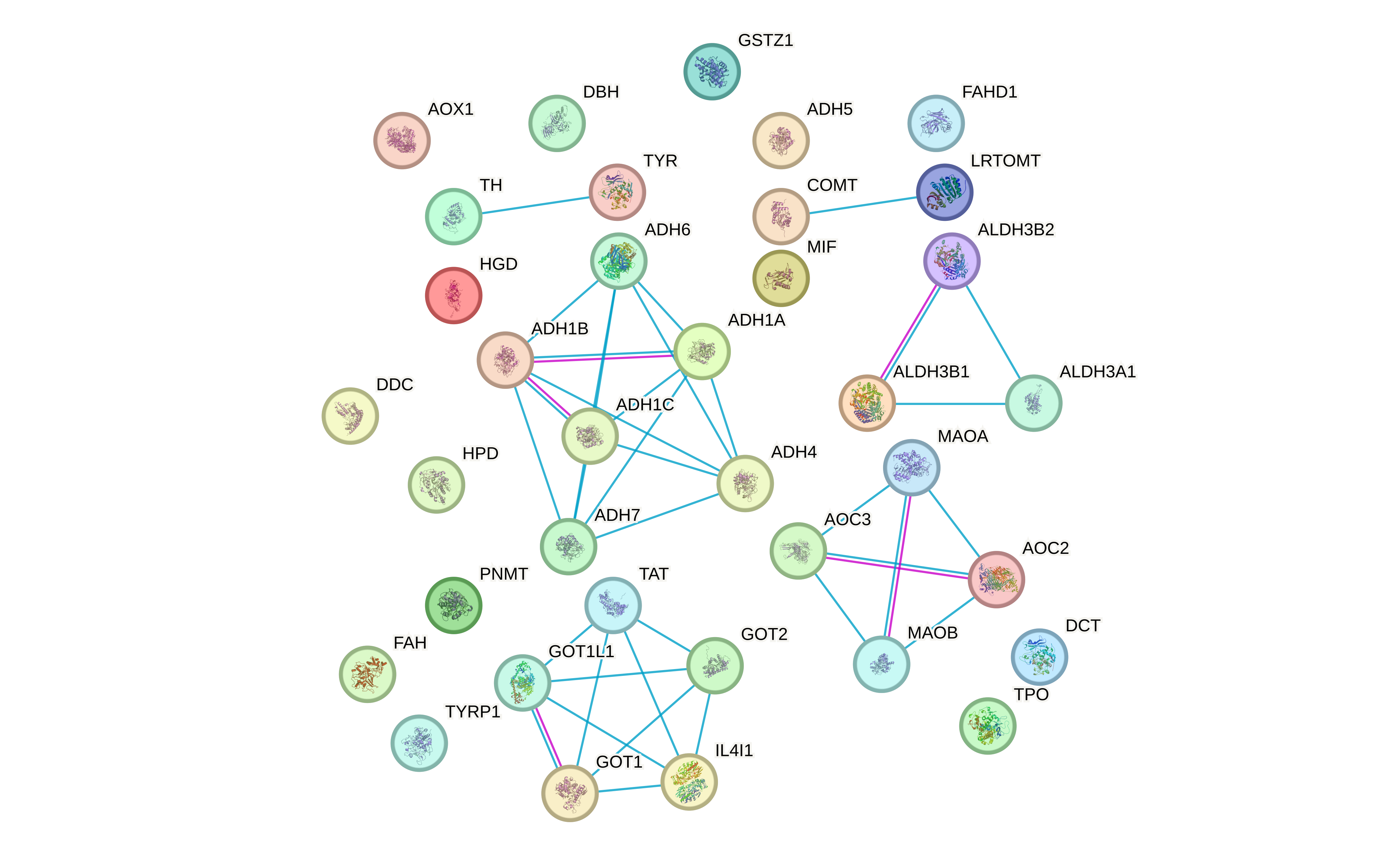
Так, хорошо, можно еще поиграться с кластеризацией, потому что на рисунках 3-4 кажется, что точно заметно два больших кластера, а мб даже и три.
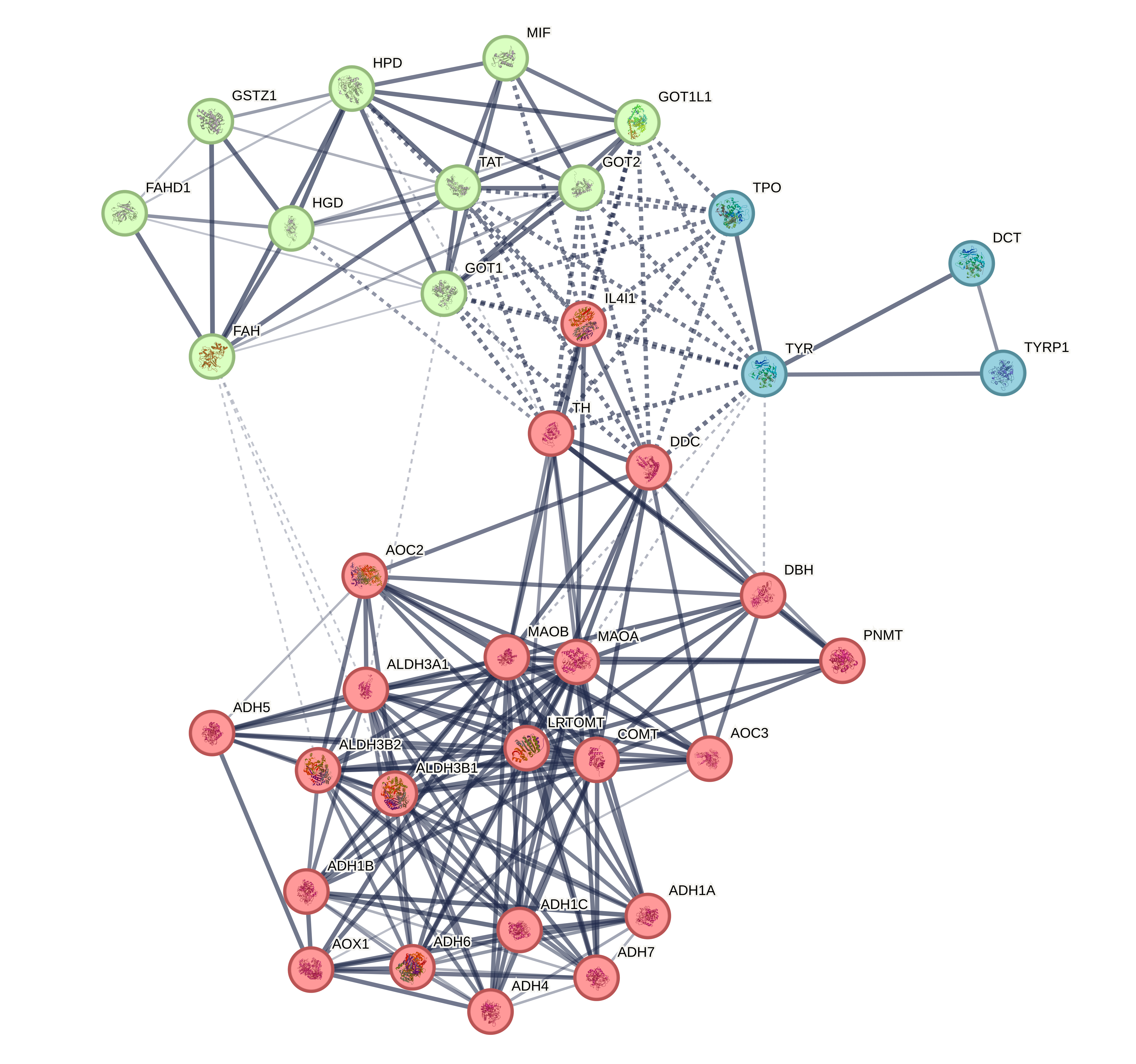
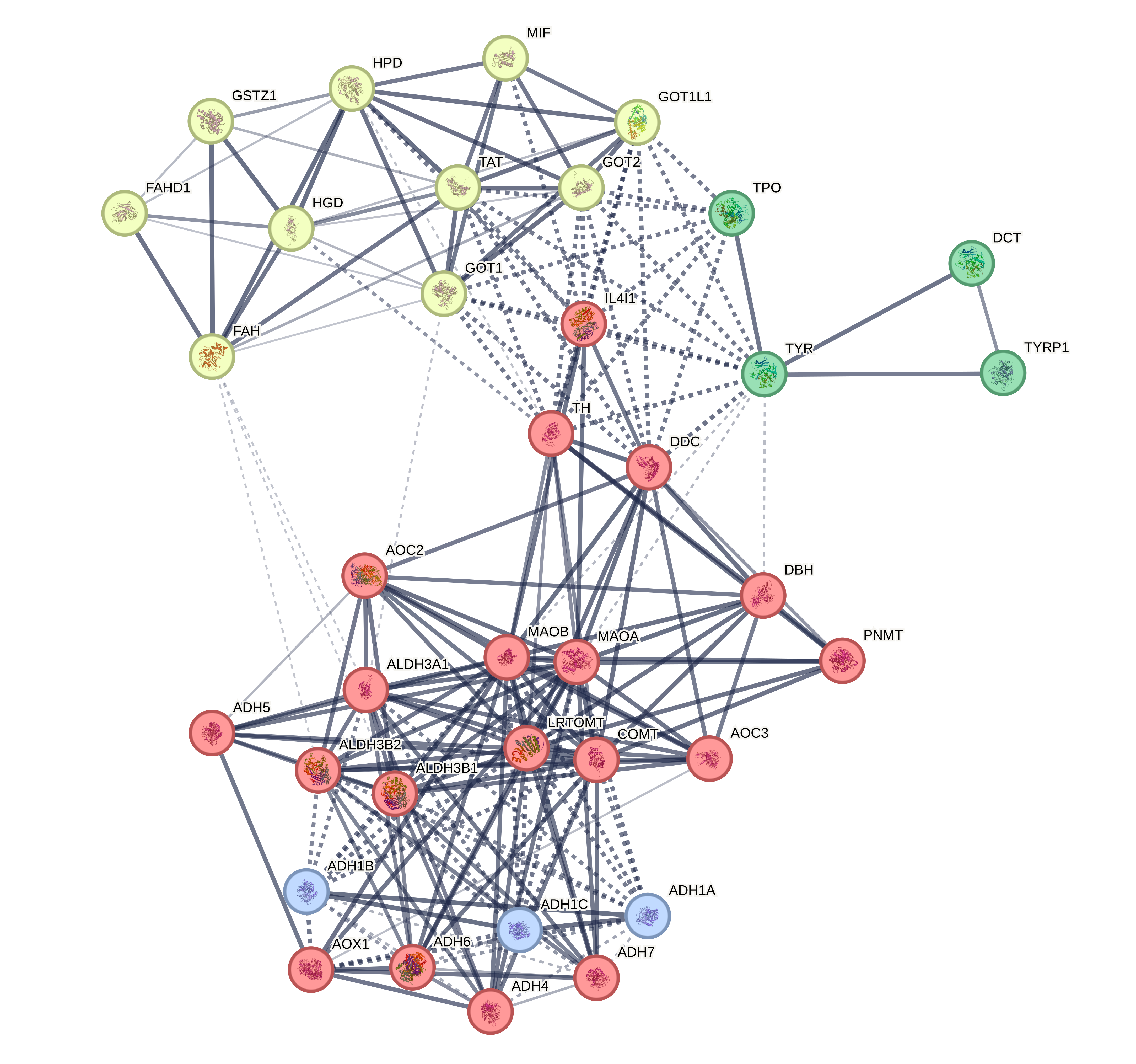
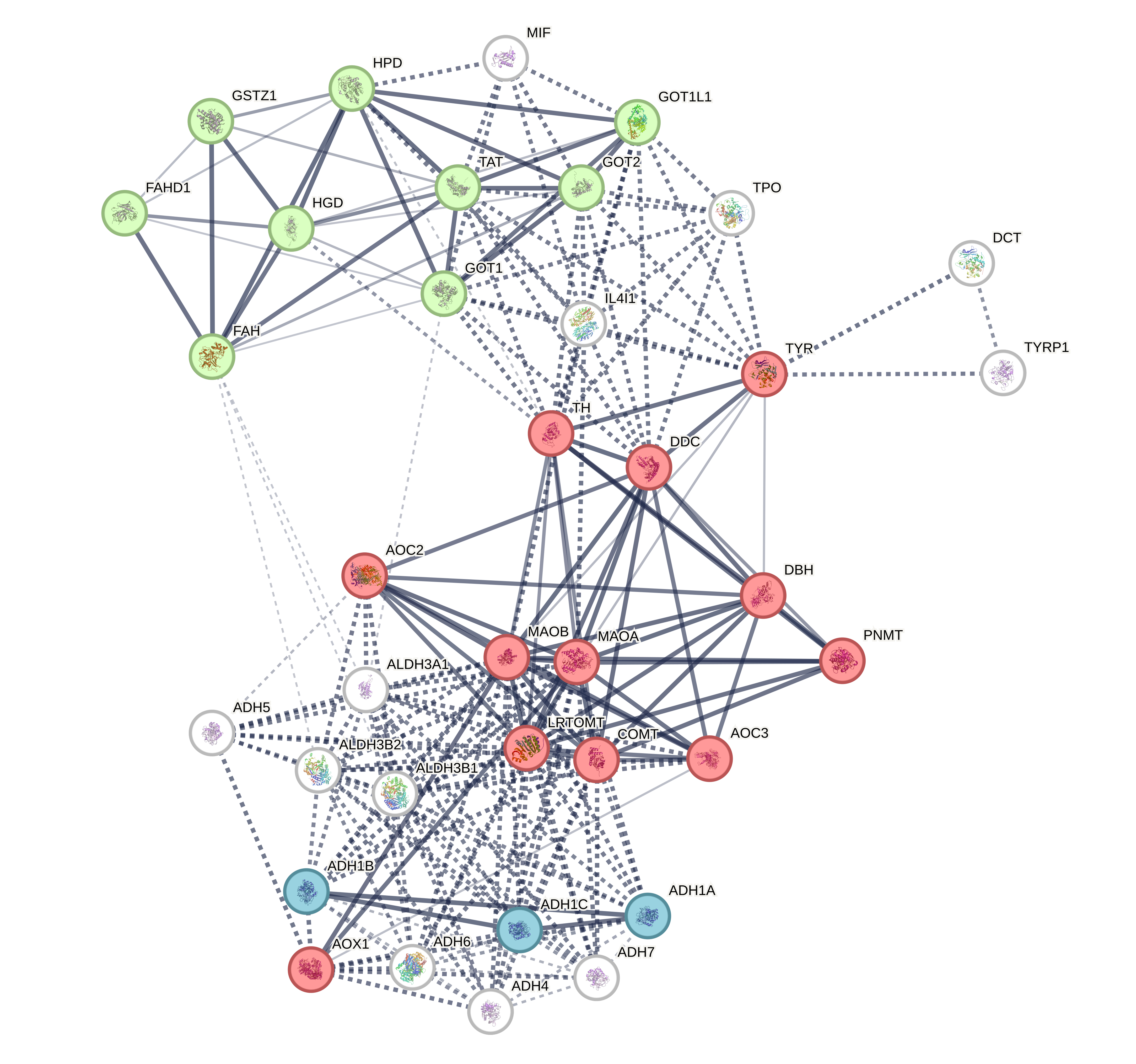
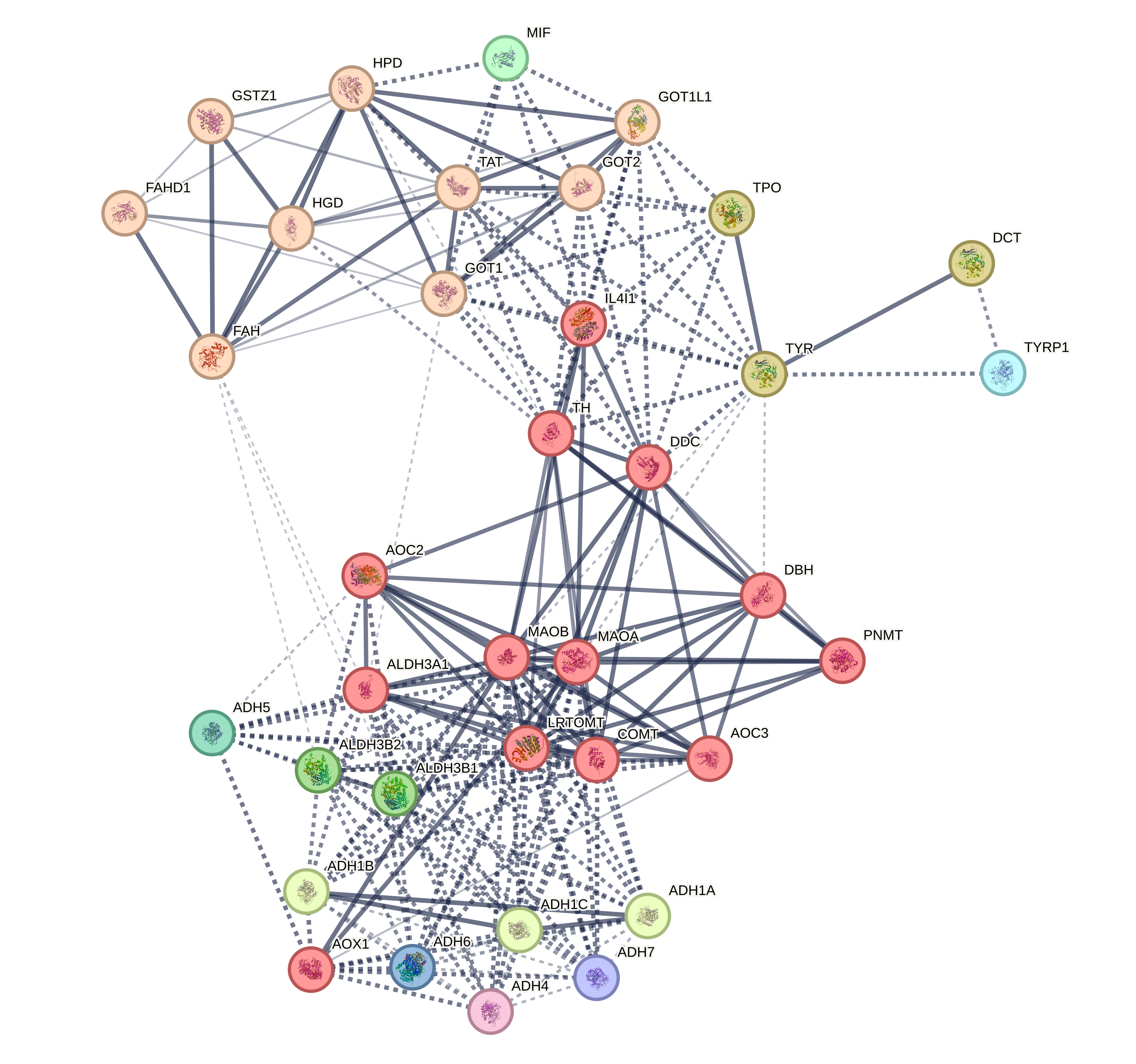
Видно, что большие кластеры одинаковые (ну, то есть 3 кластера из алгоритма с плотностью узлов). Причем один из них это часть комплекса белков ADHxx. Интересно, что только 3 белка из комплекса ВЕЗДЕ выделяются в отдельны кластер. Но, кстати, в алгоритме по плотности узлов остальные белки комплекса ADHxx не относятся ни к какому кластеру. супергуд!!!
Если посмотреть раздел Analyses, то можно взглянуть на данные с низким FDR (ну хотя бы меньше 0.001)
Биологический процесс GO: катаболизм тирозина и биосинтез аспартата
Локальные кластеры STRING
- катаболизм дофамина: MAOA, MAOB, PNMT, DBH, COMT, LRTOMT (6/6)
- альдегид- и этанол-дегидрогиназы, NADP-зависимые
- катаболизм тирозина: HPD, HGD, FAH, GSTZ1< (4/5)
- большой кластер, связанный с нейромедиаторами (8/11)
Прикола ради посмотрю коэкпрессию и cooccurrence (хз как красиво на русский перевести), но в принципе, и так понятно, что там все будет ок
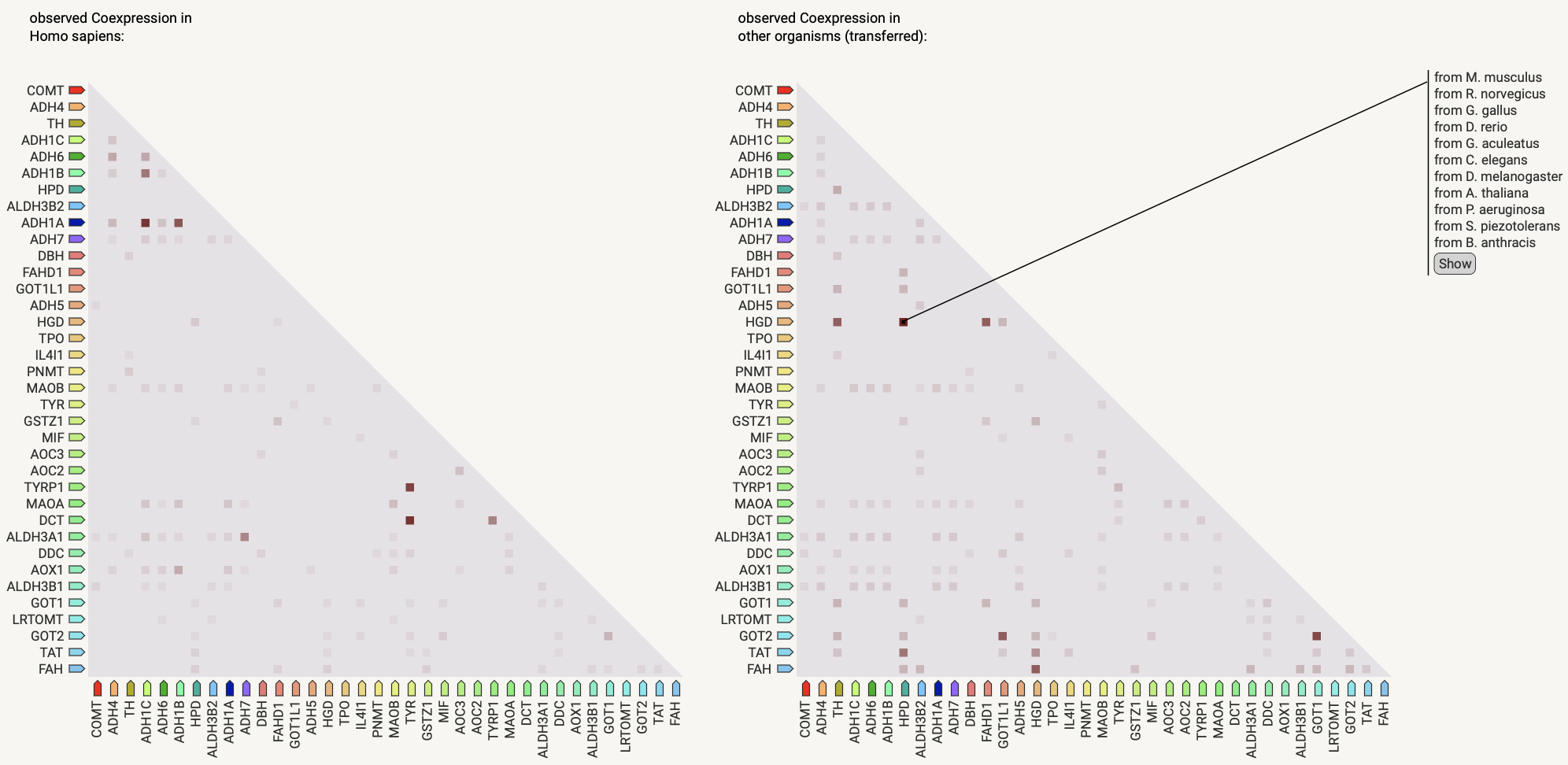
Human Protein Atlas
Оказалось, что тут можно смотреть только отдельные гены... кринж... смотреть 35 генов я убьюсь, а по 3 генам делать вывод по остальным 32 неохота. Поэтому я просто потом их закину в reactome, а ради красивых картиночек выберу неизвестный мне ген GOT1 (в честь GOT7, благослови господь джексона вана)
Посмотрим на экспрессию по тканям
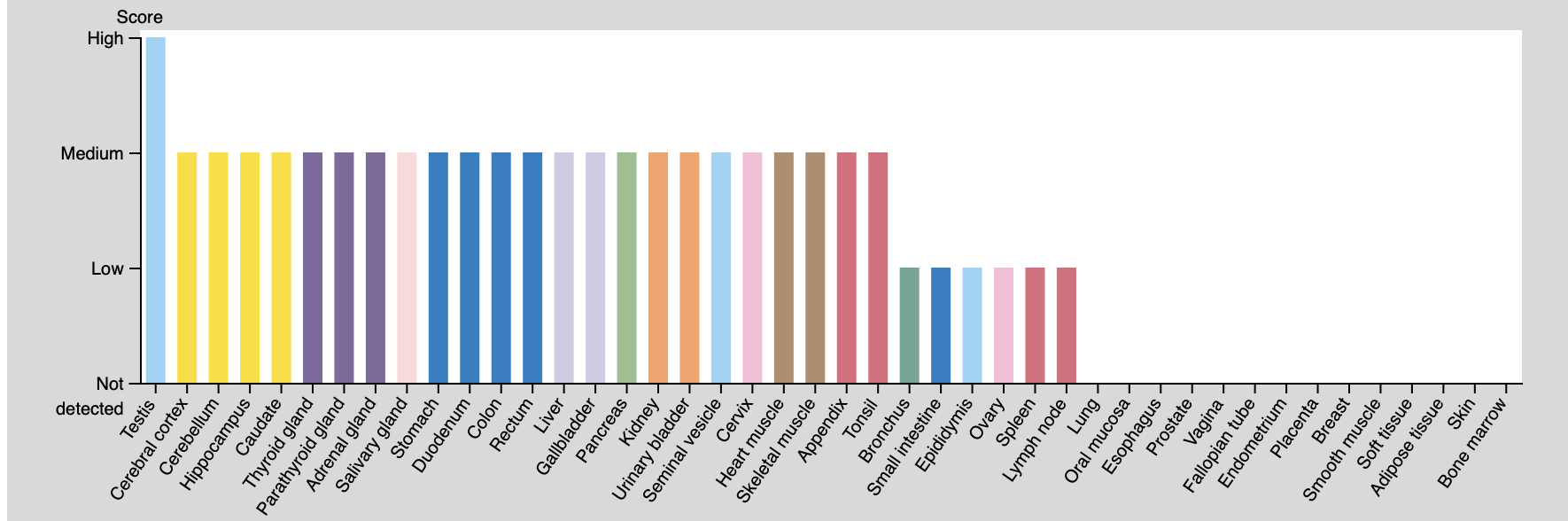
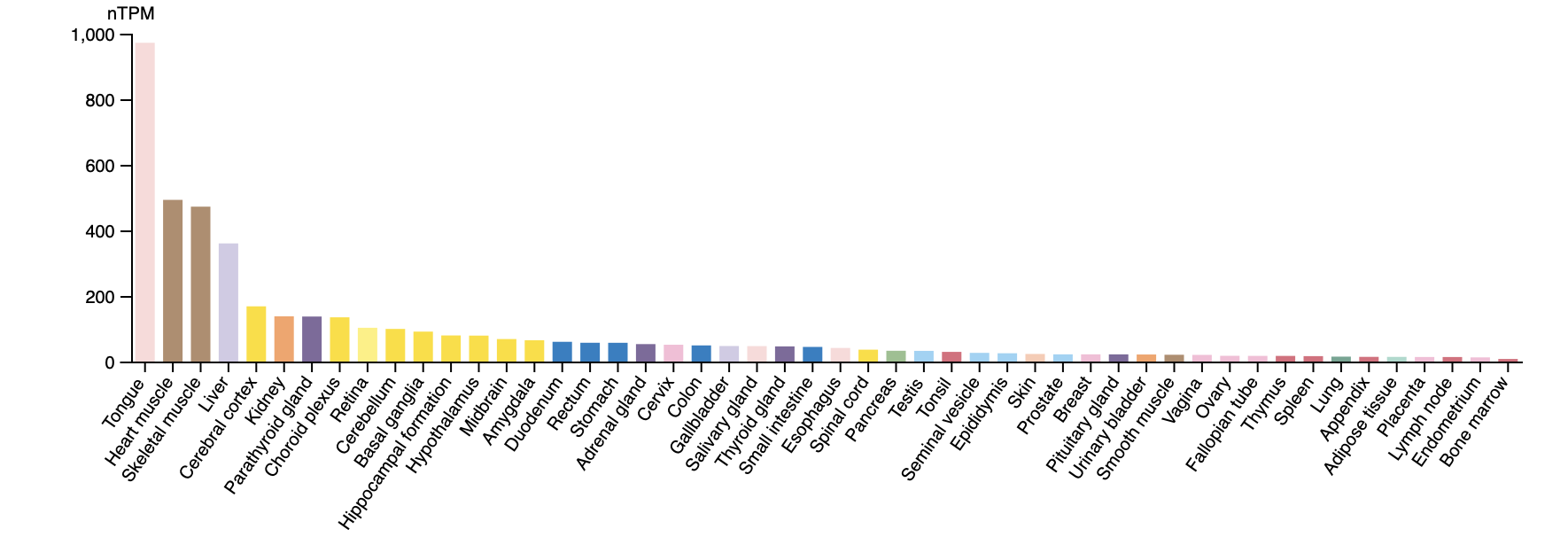
аэаааъоъ... Довольно неожиданно увидеть на первом месте экспрессии рнк язык, а белка - тестикулы...

но в целом, можно сказать, что белок экспрессируется в большом количестве тканей и очень сильно, при этом разные зоны мозга занимают топ-места. Кстати про мозг:
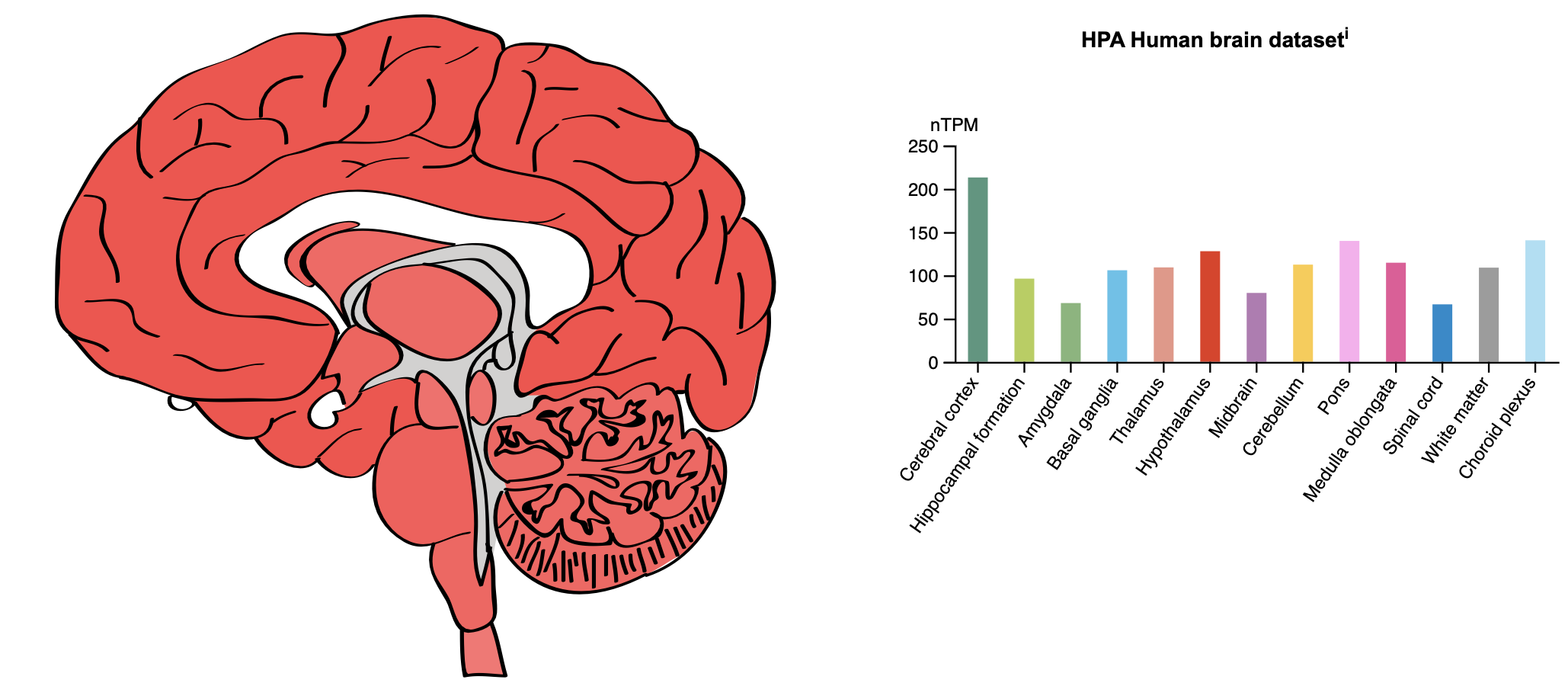
Reactome
Какой убогий интерфейс... мне не понравилось((
Но в общем, путем долгих тыканий, дошла до следующих картинок и процессов:
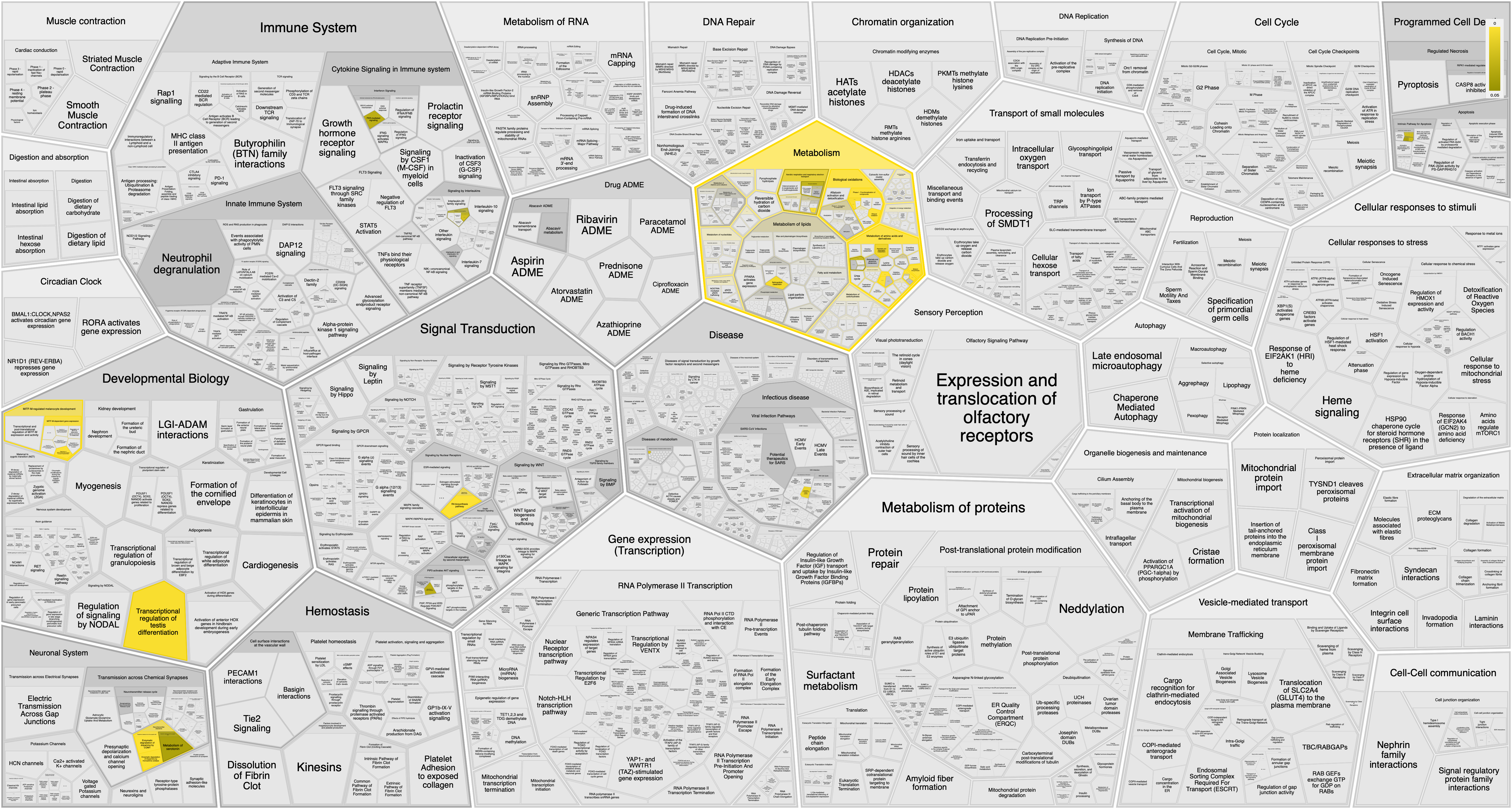
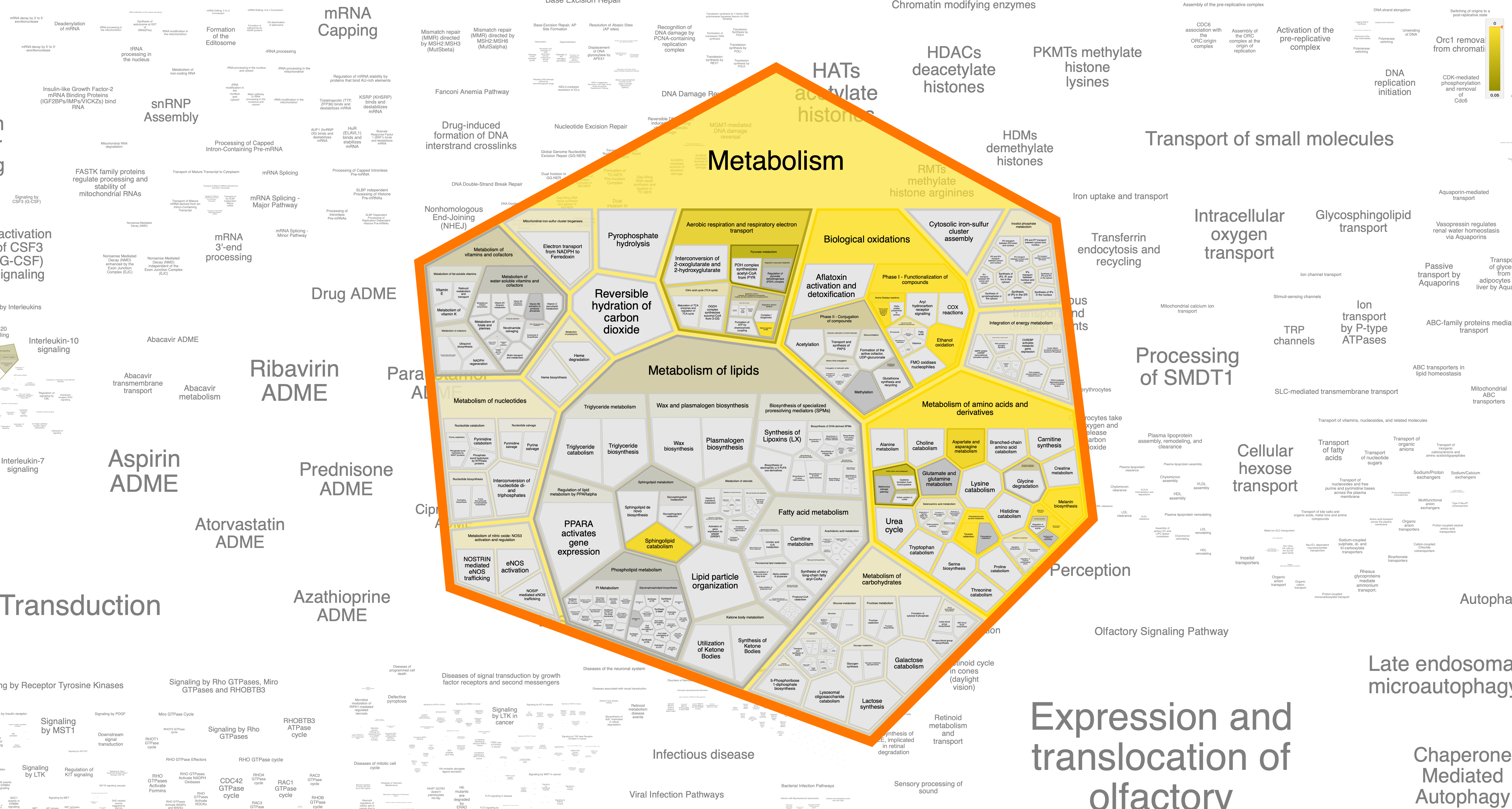
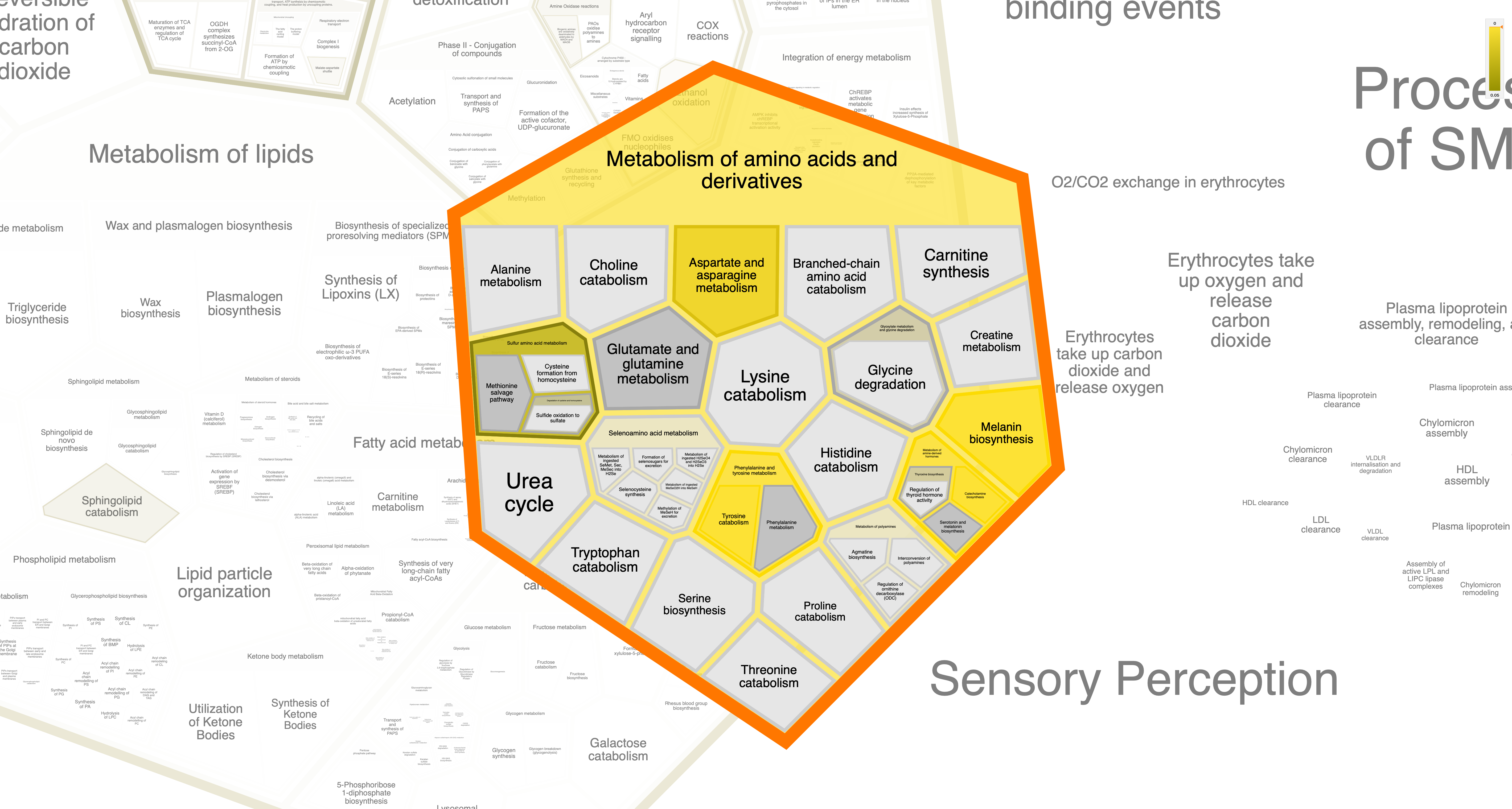
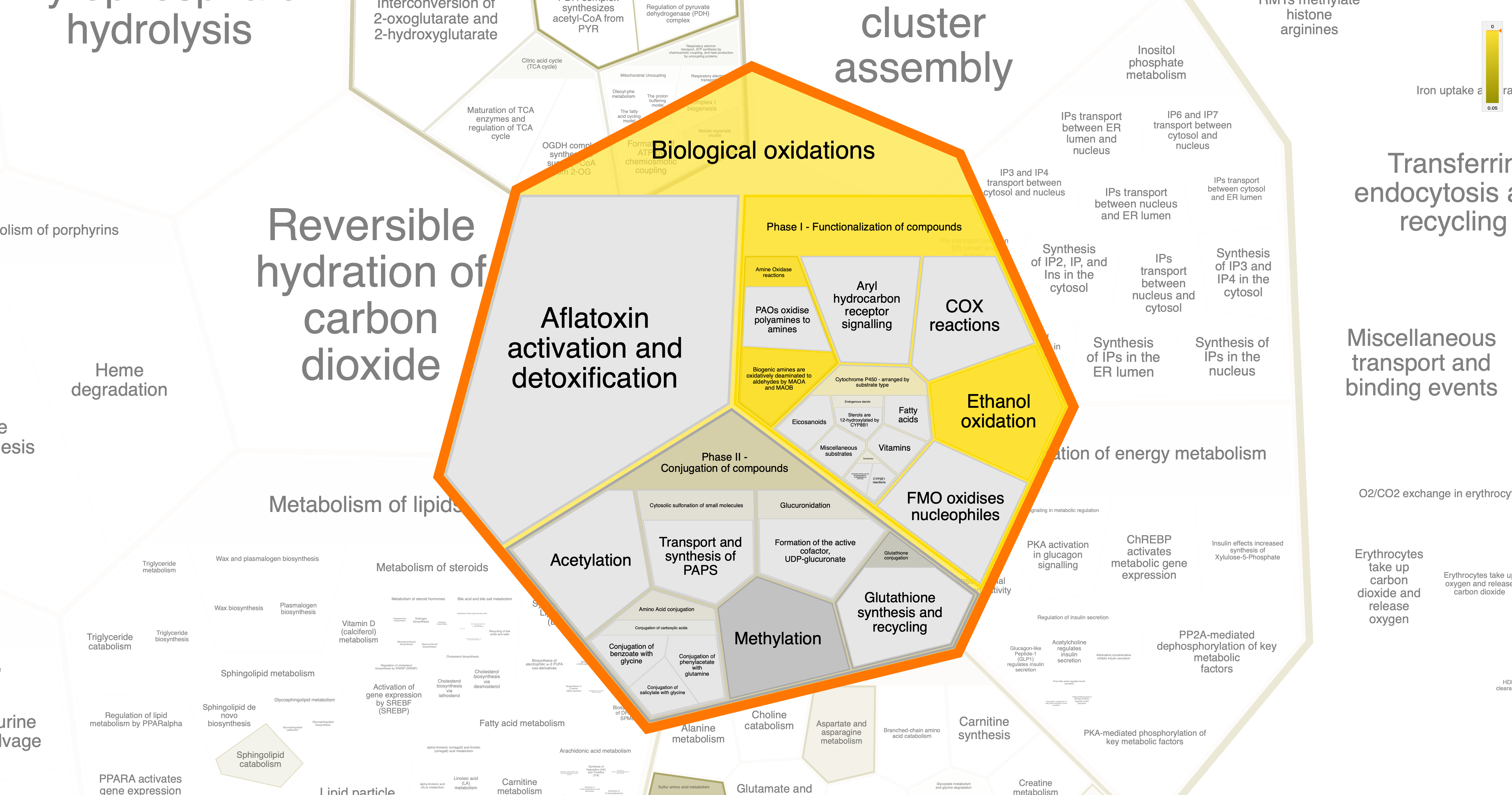
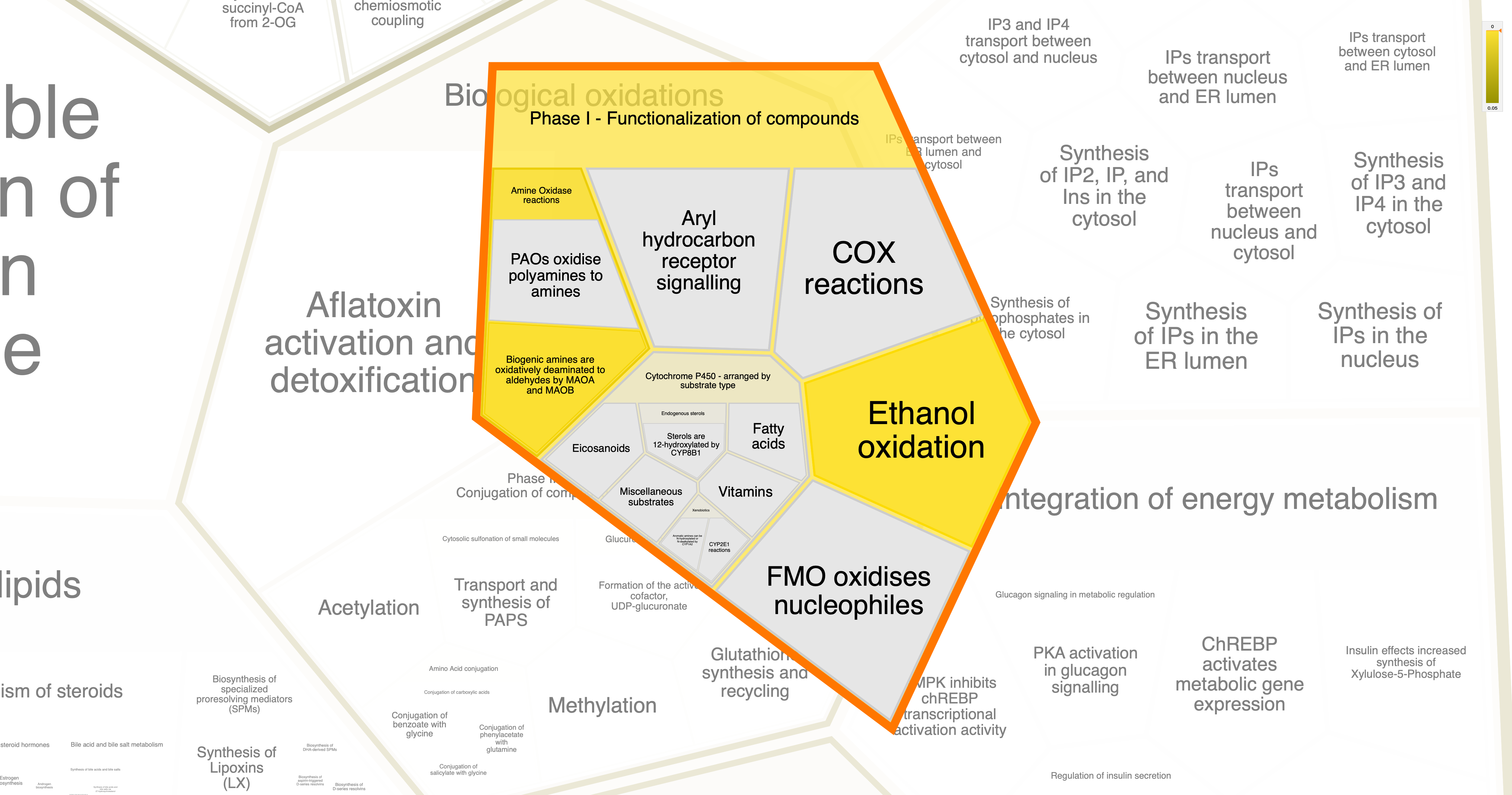
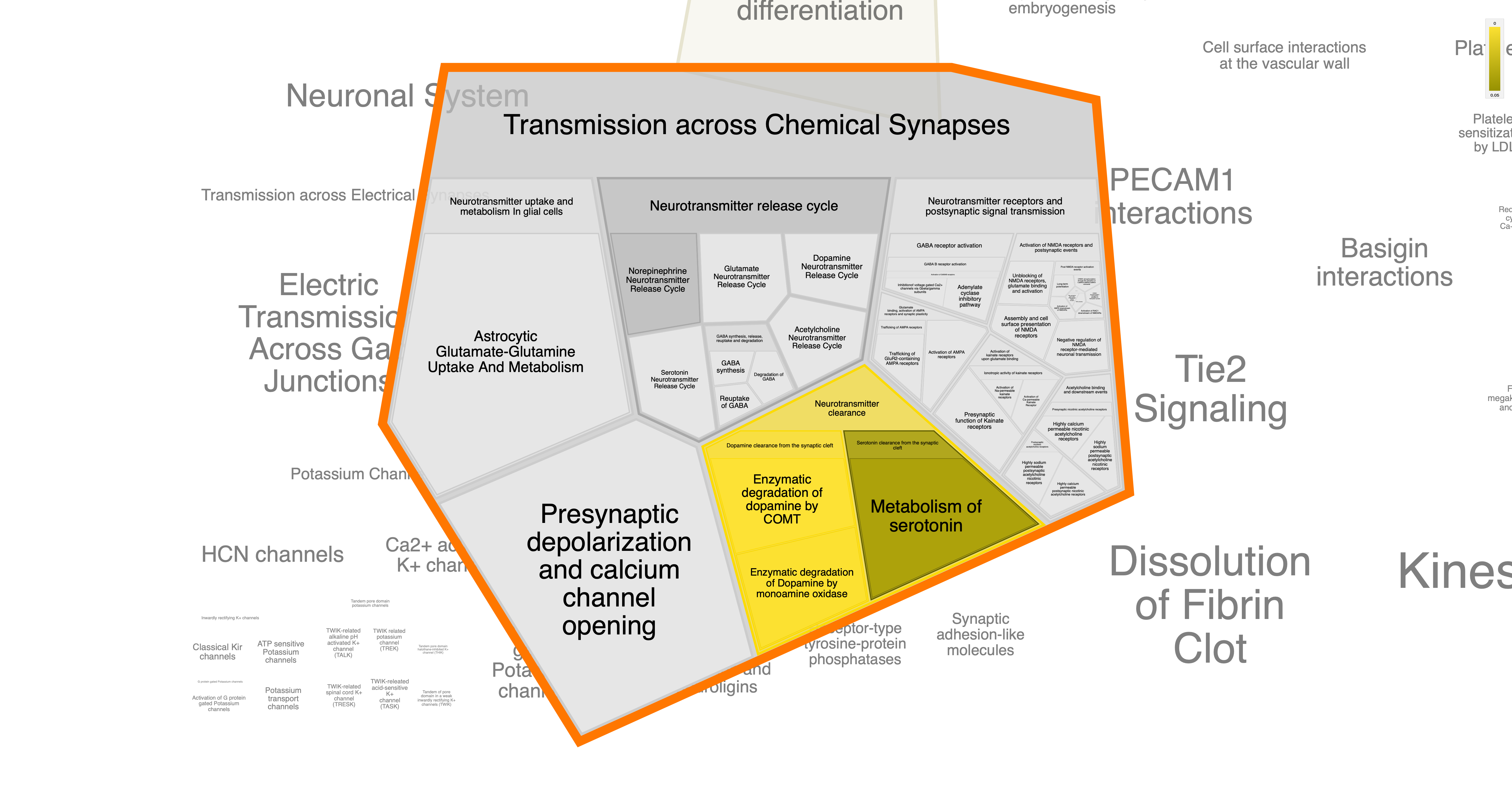


KEGG
Tyrosine pathway
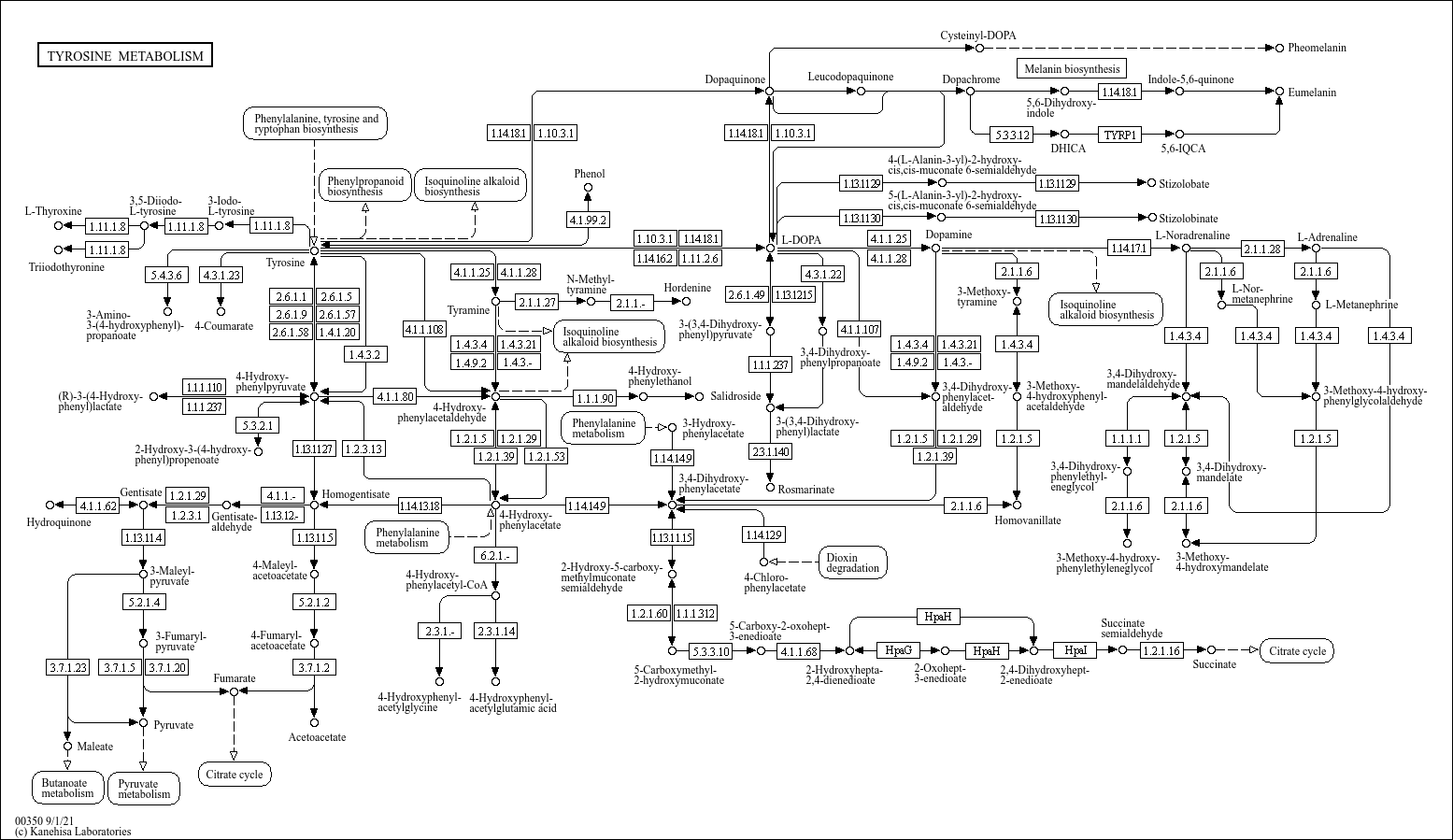
ВЫВОДЫ
Во-первых, про бд: GO самая информативная в плане объема информации, хотя там нет никаких картинок. Использовать ее не очень удобно, но для какого-то анализа выборки достаточно. STRING мне понравилась больше всего, есть сводка из GO, графы красивые и кастомизируемые на любой вкус. Protein Atlas - больше похоже на игрушку и склад красивых картинок (ну собственно он и есть атлас), а Reactome просто не оч удобно использовать, хотя ответ дал проще всех.
Итого, я вроде как сразу вдалась в правильное направление мыслей про нейромедиаторы и тирозин. Мой сет генов принимает участие преимущественно в 4 процессах:
- Метаболизм АК и их производных, преимущественно ароматических (и еще более преимущественно тирозина)
- Регуляиця нейромедиаторов, их ингибирование
- Окисление этанола и биогенных аминов
- дИфФеРеНцИаЦиЯ тЕсТиКуЛ (увы)

"Не стесняйтесь проявлять свою фантазию и испытывать мое чувство юмора."
извините