Практикум 4
Составление списка гомологичных белков, включающих паралоги
Отобранные организмы
| Название | Мнемоника |
|---|---|
| Yersinia pestis | YERPE |
| Polynucleobacter asymbioticus | POLAQ |
| Serratia proteamaculans | SERP5 |
| Brucella suis | BRUSU |
| Bordetella pertussis | BORPE |
| Agrobacterium fabrum | AGRFC |
| Rhizobium meliloti | RHIME |
| Burkholderia mallei | BURMA |
Из директории /P/y22/term4/Proteomes я скопировала к себе в папку нужные мне протеомы бактерий. Провела с помощью программы blastp поиск достоверных гомологов белка CLPX_ECOLI. Для этого выполнила следующие команды:
1) cat AGRFC.fasta BORPE.fasta BRUSU.fasta BURMA.fasta POLAQ.fasta RHIME.fasta SERP5.fasta YERPE.fasta > proteomes.fasta
2) makeblastdb -dbtype prot -in proteomes.fasta -out clpx_db
3) blastp -query clpx_ecoli.fasta -db clpx_db -evalue 0.001 -out results.txt
Выдача
sp|A8GAR0|CLPX_SERP5 ATP-dependent Clp protease ATP-binding subun... 806 0.0
sp|Q8ZC66|CLPX_YERPE ATP-dependent Clp protease ATP-binding subun... 805 0.0
sp|Q62JK8|CLPX_BURMA ATP-dependent Clp protease ATP-binding subun... 617 0.0
sp|A4SXD7|CLPX_POLAQ ATP-dependent Clp protease ATP-binding subun... 613 0.0
sp|Q7VXI6|CLPX_BORPE ATP-dependent Clp protease ATP-binding subun... 613 0.0
sp|Q92QQ2|CLPX_RHIME ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|Q8UFY5|CLPX_AGRFC ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|Q8G0I5|CLPX_BRUSU ATP-dependent Clp protease ATP-binding subun... 586 0.0
sp|A8GL96|HSLU_SERP5 ATP-dependent protease ATPase subunit HslU O... 96.7 9e-22
sp|Q8ZJJ5|HSLU_YERPE ATP-dependent protease ATPase subunit HslU O... 95.1 2e-21
sp|Q7VUJ9|HSLU_BORPE ATP-dependent protease ATPase subunit HslU O... 93.6 8e-21
sp|Q92TA7|HSLU_RHIME ATP-dependent protease ATPase subunit HslU O... 92.8 1e-20
sp|Q8UJ87|HSLU_AGRFC ATP-dependent protease ATPase subunit HslU O... 92.0 3e-20
sp|Q8FY12|HSLU_BRUSU ATP-dependent protease ATPase subunit HslU O... 90.9 6e-20
sp|Q62F00|HSLU_BURMA ATP-dependent protease ATPase subunit HslU O... 82.4 5e-17
tr|A8GCD8|A8GCD8_SERP5 ATP-dependent Clp protease, ATP-binding su... 51.6 6e-07
tr|A0A5P8YGZ0|A0A5P8YGZ0_YERPE ATP-dependent Clp protease ATP-bin... 51.2 6e-07
tr|A8G901|A8G901_SERP5 ATP-dependent zinc metalloprotease FtsH OS... 46.2 2e-05
tr|A0A5P8YB42|A0A5P8YB42_YERPE ATP-dependent protease OS=Yersinia... 46.2 2e-05
tr|A0A5P8YCE6|A0A5P8YCE6_YERPE ATP-dependent zinc metalloprotease... 45.8 3e-05
tr|A0A0H3GCZ6|A0A0H3GCZ6_BRUSU ATP-dependent zinc metalloprotease... 45.4 4e-05
tr|Q92M98|Q92M98_RHIME ATP-dependent zinc metalloprotease FtsH OS... 45.4 4e-05
tr|Q7CT50|Q7CT50_AGRFC ATP-dependent zinc metalloprotease FtsH OS... 45.4 5e-05
tr|A0A0H2WJ72|A0A0H2WJ72_BURMA ATP-dependent zinc metalloprotease... 43.9 1e-04
tr|A4SXL5|A4SXL5_POLAQ ATP-dependent zinc metalloprotease FtsH OS... 43.5 2e-04
Полный файл с выдачей BLAST можно скачать здесь. Как мы видим, получилось 25 гомологов
Реконструкция и визуализация
Файл с полученными последовательностями можно посмотреть здесь. Загрузила файл на сайт NGPhylogeny.fr с параметрами:
Multiple Alignment: MAFFT
Tree Inference: FastME
Gamma distributed rates across sites: No
Starting tree: BIONJ
Number of bootstrap replicates: 100

Дерево в Newick формате здесь
Далее укореняю дерево в midpoint и отображаю bootstrap анализ

Примеры ортологов и паралогов:
Ортологи: СLPX_BURMA и CLPX_BORPE, HSLU_SERP5 и HSLU_YERPE, A4SXL5_POLAQ и A0A0H2WJ72_BURMA
Паралоги: CLPX_RHIME, HSLU_RHIME, Q92M98_RHIME
Выделение ортологических групп
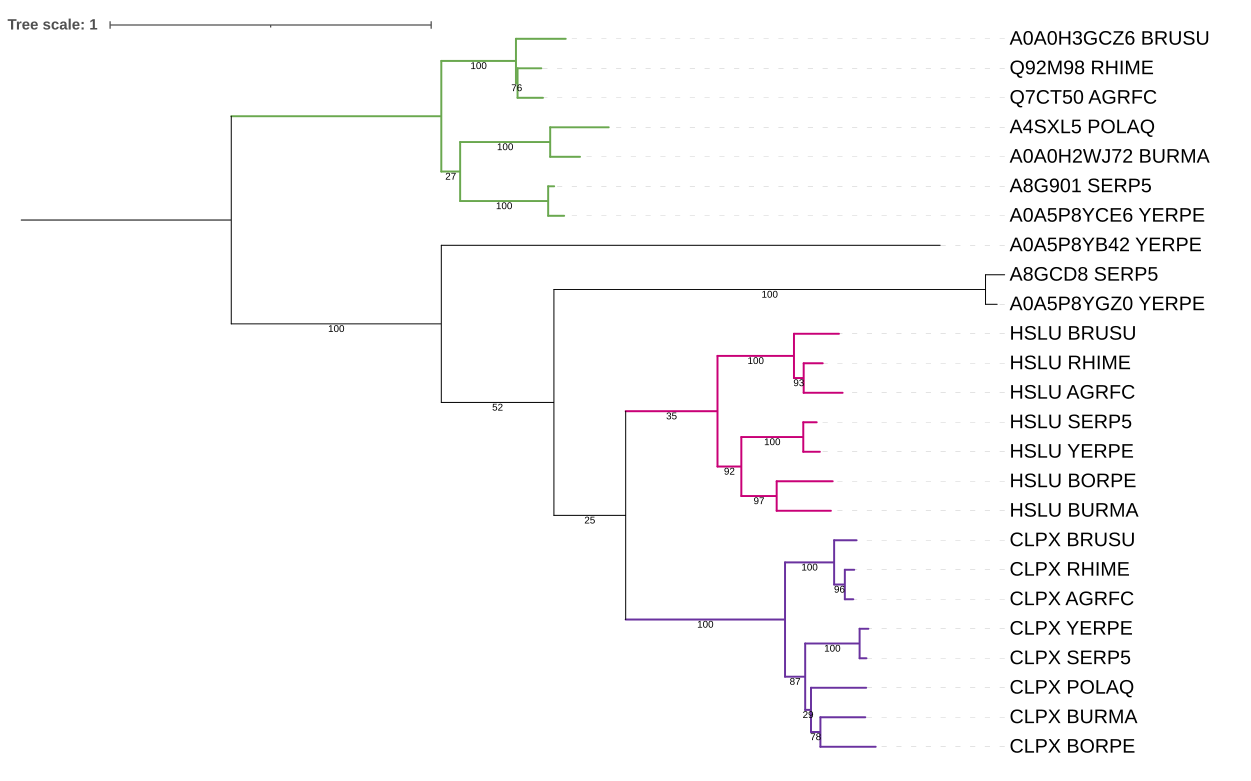
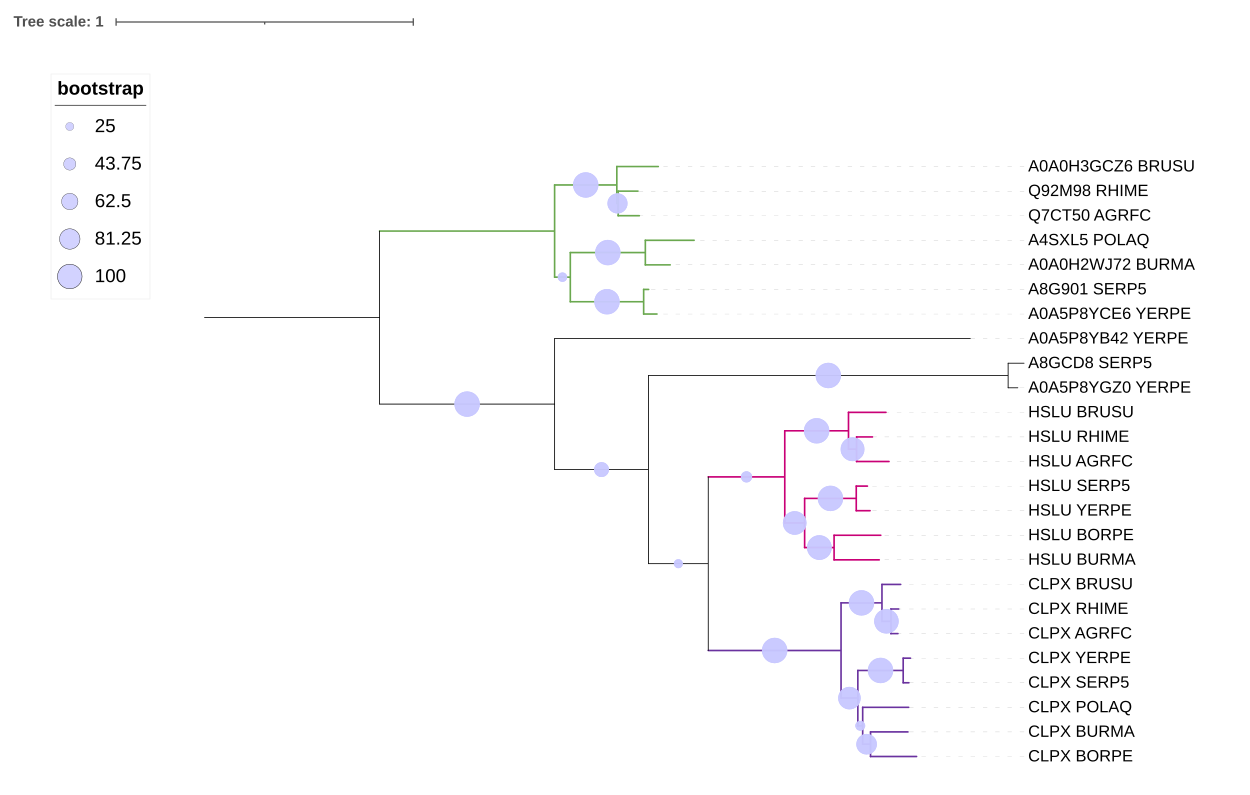

По полученным деревьям можно выделить 3 основные ортологические группы:
FTSH - АТФ-зависимые цинковые металлопротеазы FtsH (ATP-dependent zinc metalloprotease FtsH). В эту группу входят 7 белков из выбранных бактерий (все, кроме BORPE). Топология совпадает с исходным деревом
HSLU - АТФ-зависимая субъединица АТФ-зависимой протеазы HslU (ATP-dependent protease ATPase subunit HslU). В эту группу входят 7 белков из выбранных бактерий (все, кроме POLAQ). Топология совпадает с исходным деревом
CLPX - АТФ-связывающий сабъюнит АТФ-зависимой протеазы Clp (ATP-dependent Clp protease ATP-binding subunit СlpX). В эту группу входят все 8 белков из выбранных бактерий. Топология совпадает с исходным деревом