| A- и В- формы ДНК. Структура РНК | |||||||||||||||||||||||||||||||||||||||||||||||||
| Главная |
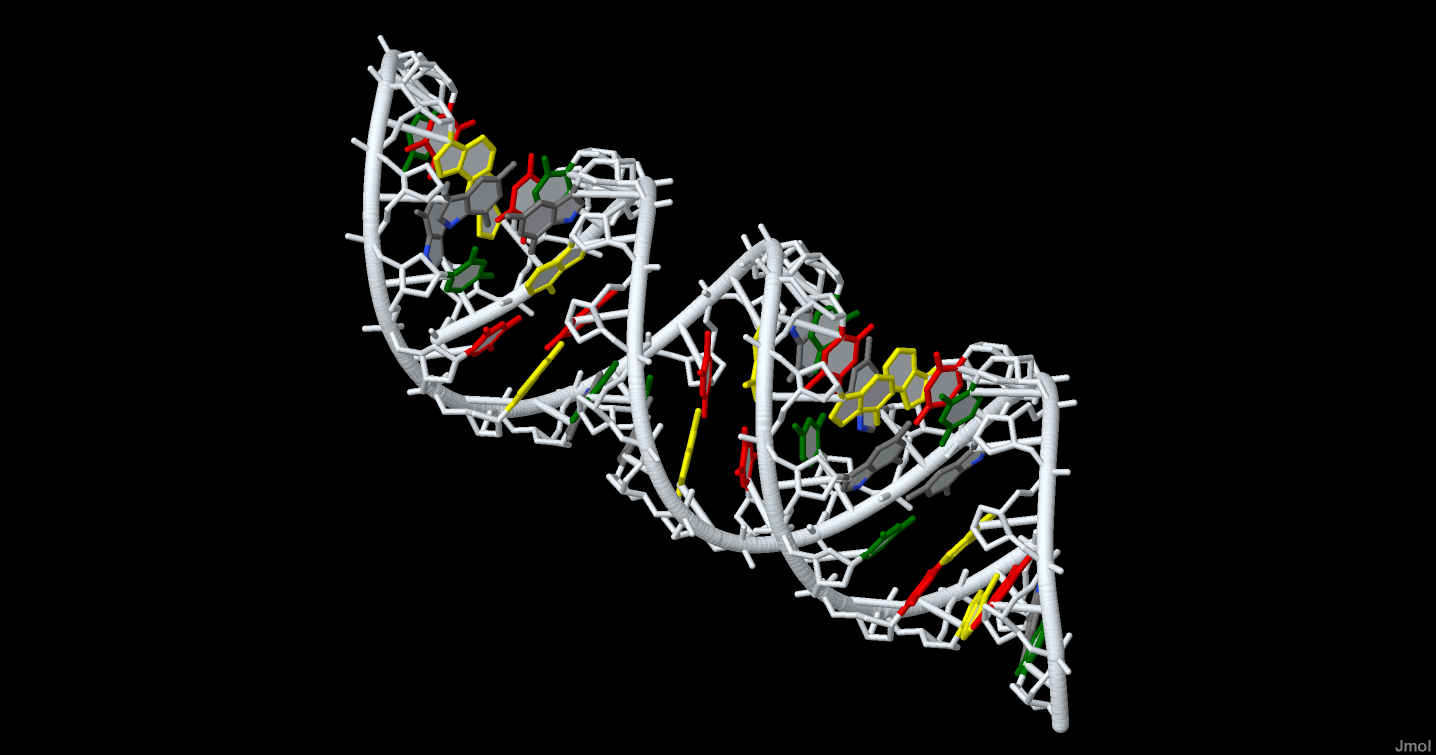
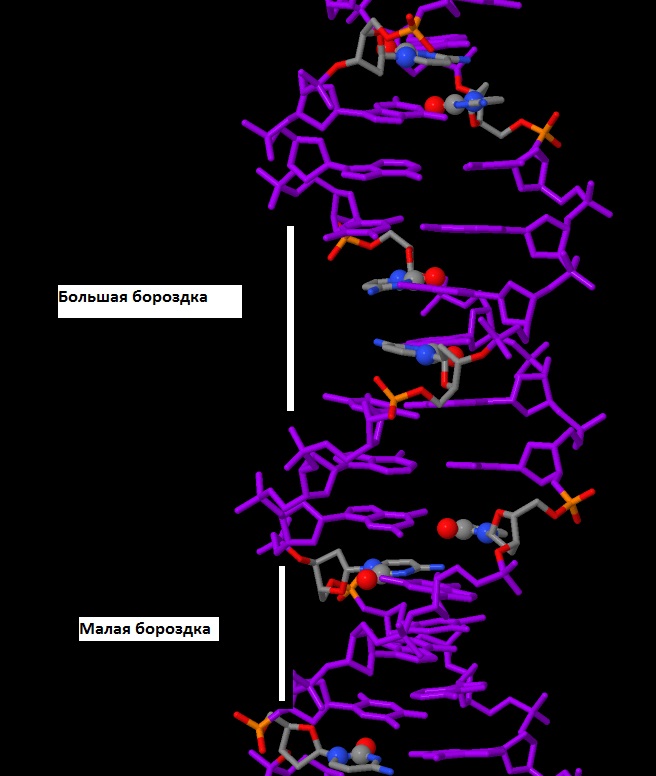
Задание 1 С помощью программы fiber пакета 3DNA были получены PDB-файлы A-, B- и Z-форм дуплекса ДНК. Команда для получения А-формы: fiber -a gatc_a.pdb Задание 2 На рис. 1 показана А-форма ДНК. В задании нужно было выделить: - сахарофосфатный остов ДНК; - все нуклеотиды; - все аденины; - атом N7 во всех гуанинах и/или только в первом по последовательности. Далее мне было предложено исследовать стрктуры тРНК и ДНК, с идентификаторами 1f7u и 1tro соответственно, на наличие разрывов. В структуре РНК (1f7u) с помощью визуализации Jmol разрывов не было обнаружено (рис. 2.1). В структуре ДНК (1tro) был обнаружен разрыв, это объясняется тем, что в файле PDB представлен димер, то есть 2 молекулы белка, связанных с ДНК. Задание 3 В A-, B- и Z-формах ДНК одни и те же атомы направлены к разным бороздкам. На рис. 3 изображена B-форма ДНК. Атомы цитозина O2, N1 и C2, смотрящие в сторону малой бороздки, показаны в шариковой модели. На рис. 4 представлено изображение цитозина, которое было получено в программе ChemSketch. Сравнение основных параметров A-, B- и Z-форм ДНК представлено в таблице 1.
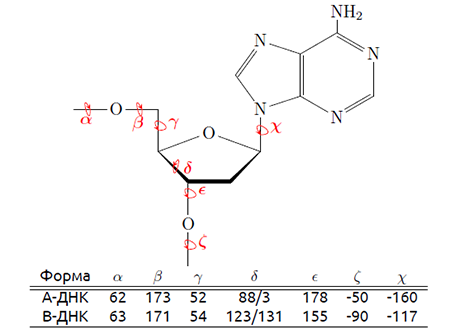
Для того, чтобы определить шаг спирали, я измерял расстояние между расположенными друг под другом атомами фосфора (рис. 5). Далее я сравнил торсионные углы в нуклетиде с цитозином, A- и B-формах ДНК. Результат представлен в таблице 2. Торсионные углы нуклеотида изображены на рис. 6.
Задание 4 Сравнение значений торсионных углов в стркутре ДНК с идентификатором PDB 1tro и РНК 1f7u представлено в таблице exel . Желтым выделены нуклеотиды, значения торсионных углов которых сильно отклоняются от средних. Для изучения структуры водородных связей в молекуле тРНК я использовал данные из файла с расширением .out, предсталенные ниже.
Strand I Strand II Helix
1 (0.044) B:.901_:[PSU]P-*---A[..A]:.972_:B (0.003) |
2 (0.003) B:.902_:[..U]U-----A[..A]:.971_:B (0.003) |
3 (0.002) B:.903_:[..C]C-----G[..G]:.970_:B (0.003) |
4 (0.002) B:.904_:[..C]C-----G[..G]:.969_:B (0.004) |
5 (0.002) B:.905_:[..U]U-*---G[..G]:.968_:B (0.007) |
6 (0.006) B:.906_:[..C]C-----G[..G]:.967_:B (0.007) |
7 (0.004) B:.907_:[..G]Gx----C[..C]:.966_:B (0.002) |
8 (0.016) B:.949_:[5MC]c-----G[..G]:.965_:B (0.004) |
9 (0.004) B:.950_:[..C]C-----G[..G]:.964_:B (0.008) |
10 (0.002) B:.951_:[..A]A-----U[..U]:.963_:B (0.004) |
11 (0.010) B:.952_:[..G]G-----C[..C]:.962_:B (0.006) |
12 (0.005) B:.953_:[..G]G----xC[..C]:.961_:B (0.004) |
13 (0.010) B:.954_:[5MU]u-**-xa[1MA]:.958_:B (0.005) |
14 (0.044) B:.955_:[PSU]Px**+xG[..G]:.917_:B (0.006) x
15 (0.005) B:.939_:[..C]C-----G[..G]:.931_:B (0.005) |
16 (0.005) B:.940_:[..C]C-----G[..G]:.930_:B (0.005) |
17 (0.002) B:.941_:[..A]A-----U[..U]:.929_:B (0.002) |
18 (0.006) B:.942_:[..G]G-----C[..C]:.928_:B (0.003) |
19 (0.003) B:.943_:[..A]A-*---P[PSU]:.927_:B (0.045) |
20 (0.004) B:.944_:[..A]Ax*---g[M2G]:.926_:B (0.005) |
21 (0.007) B:.910_:[2MG]g-----C[..C]:.925_:B (0.003) |
22 (0.005) B:.911_:[..C]C-----G[..G]:.924_:B (0.008) |
23 (0.004) B:.912_:[..C]C-----G[..G]:.923_:B (0.012) |
24 (0.005) B:.913_:[..C]C-*--xC[..C]:.922_:B (0.002) |
25 (0.009) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.004) |
26 (0.012) B:.915_:[..A]Ax**+xU[..U]:.948_:B (0.004) x
27 (0.015) B:.918_:[..G]G-----C[..C]:.956_:B (0.001) +
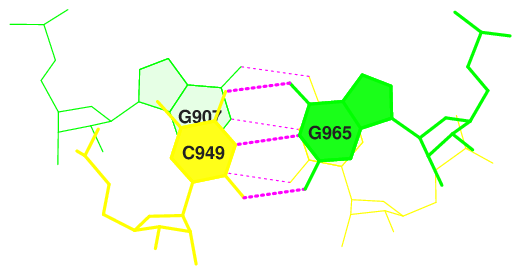
Шпильки тРНК выделены разными цветами. Пары с неканоническими взаимодействиями: 1, 5, 14, 19, 20, 24. Водородные связи между остатками 954 и 958, 955 и 917, 914 и 908, 915 и 948, 918 и 956 стабилизируют третичную структуру тРНК. В файле с расширением .out также можно найти данные о стекинговых взаимодействиях. Пара с самым сильным стекинговым взаимодествием показана на рис. 7. |
||||||||||||||||||||||||||||||||||||||||||||||||
| Обо мне | |||||||||||||||||||||||||||||||||||||||||||||||||
| Ссылки | |||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||
© Denis Moshensky, 2013
Дата последнего изменения: 16.02.2015
Задавайте вопросы по электронной почте