| Реконструкция филогении по нуклеотидным последовательностям. Паралоги | |
| Главная |
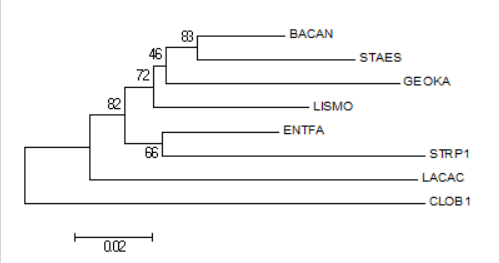
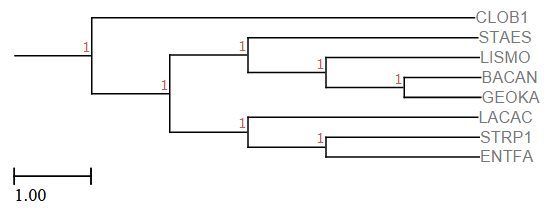
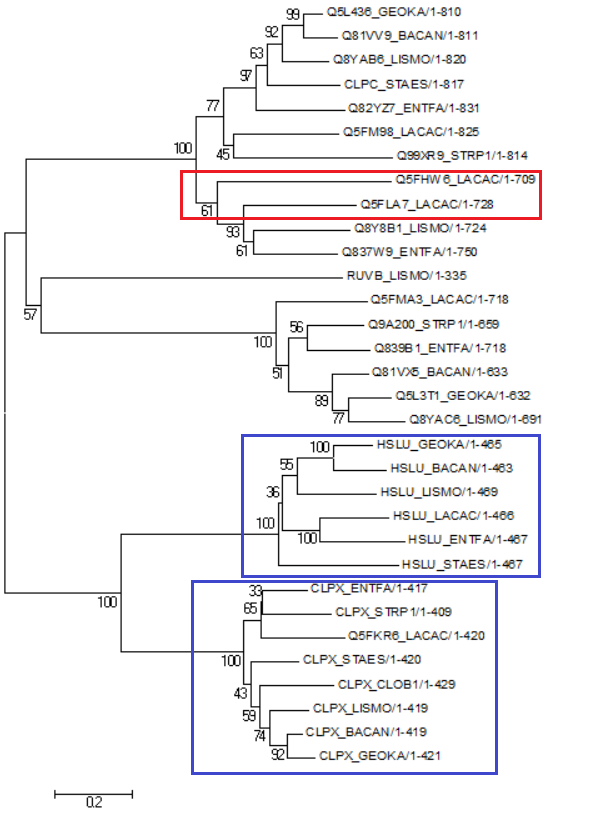
Построение дерева по нуклеотидным последовательностям Из геномов ранее выбранных 8-ми бактерий были взяты последовательности 16S рРНК. Эти последовательности были выровнены с помощью программы Muscle. Далее методов Neighbour-joining с количеством бутстрэп-реплик 100 по полученному выравниванию было построено филогенетическое дерево (рис. 1). При сравнении с исходным (правильным) филогенетическим деревом отобранных бактерий (рис. 2) видно, что появились новые неправильные нетривиальные ветви: 1) {CLOB1,LACAC} против {BACAN,STAES,GEOKA,LISMO,ENTFA,STRP1} 2) {COLOB1,LISMO,ENTFA,STRP1,LACAC} против {GEOKA,BACAN,STAES} 3) {CLOB1,ENTFA,LISMO,STRP1,GEOKA,LACAC} против {BACAN,STAES} Построение и анализ дерева, содержащего паралоги Методом Neighbour-joining с количеством бутстрэп-реплик 100 было построено филогенетическое дерево по выравниванию белков из отобранных бактерий, гомологичных белку CLPX_BACSU (рис. 3). Появление паралогов соответствует дупликации гена, а появление ортологов - разделению путей эволюции. |
| Обо мне | |
| Ссылки | |
|
|
|
© Denis Moshensky, 2013-2015
Дата последнего изменения: 28.05.2015
Задавайте вопросы по электронной почте