noreturn -infile pat_2.msf -system unix -outfile pat_unix.msf
Таким образом получен UNIX-овый конец строки.
pfw pat_unix.msf
pat_unix.msf
Значения весов изменились (в исходном файле они были равны 1.00)
pfmake pat_pfw.msf /usr/share/pftools23/blosum62.cmp
результат
seqret sw-org:bacteria bacteria.fasta
pfsearch -C4.0 -f pfmake.txt bacteria.fasta > my4.pfsearch
Первые раза находок было слишком много, поэтому порог выбран 4.0.
Найдена 765 последовательность
Вес >4: 765
Вес >5: 749
Вес >6: 651
Вес >7: 407
Вес >8: 166
Вес >9: 61 (max 9,3)
Excel
Число верных находок ("True positive hits", TP) 208
Число ложных находок ("False positive hits", FP) 553
Число ненайденных белков подсемейства ("False negatives", FN) 6
Чувствительность TP/(TP+FN) 0.9719 (лучше по сравнению с паттерном)
Селективность TP/(TP+FP) 0.2733
ROC-кривая:
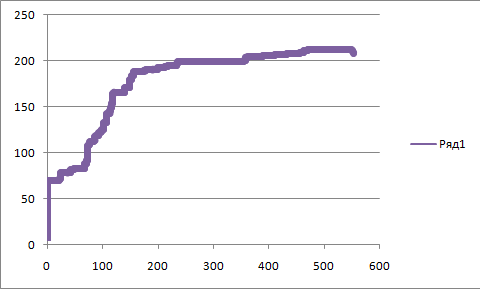
В прошлом задании чувствительность была равна 0,8174 (при пороге 50), селективность 0,3456.
В этот раз селективность (при той же чувствительности 0,8714) стала выше - 0.6967. Получается, что данный профиль лучше.
© Anastasia Maslova, 2011