Для выполнения работы использовалась программа aligncomp.
Для сравнения были построены выравнивания белков исправления мисмэтчей (белки семейства MutS) с доменной архитектурой, репрезентом которой является белок Salpingoeca rosetta (UniProtKB AC: F2UL61). Сравнивались программы множественного выравнивания Muscle, MSAprobs, Mafft.
Проект Jalview со сравниваемыми выравниваниями.
| Сравниваемые программы | Идентичные блоки | Идентичные колонки, не входящие в состав блоков |
|---|---|---|
| MSAprobes - MAFFT | 889,897 = 777,785 928,933 = 811,816 1023,1035 = 880,892 1079,1083 = 936,940 1134,1150 = 976,992 1162,1175 = 1003,1016 1405,1417 = 1173,1185 1505,1519 = 1261,1275 1571,1572 = 1312,1313 1580,1603 = 1320,1343 1607,1616 = 1347,1356 1704,1714 = 1432,1442 1742,1746 = 1474,1478 1900,1906 = 1602,1608 1939,1940 = 1638,1639 1943,1961 = 1642,1660 1982,1985 = 1682,1685 2004,2006 = 1704,1706 2012,2021 = 1712,1721 2033,2063 = 1733,1763 2178,2189 = 1876,1887 2213,2221 = 1904,1912 2366,2378 = 2022,2034 |
3,53 = 3,53 57,149 = 57,149 162,165 = 175,178 182,186 = 190,194 231,237 = 241,247 262,264 = 276,278 288,288 = 302,302 849,849 = 739,739 851,854 = 741,744 861,870 = 749,758 873,873 = 761,761 877,887 = 765,775 906,910 = 793,797 994,998 = 849,853 1060,1060 = 927,927 1116,1121 = 958,963 1393,1395 = 1163,1165 1428,1428 = 1196,1196 1480,1480 = 1232,1232 1494,1495 = 1250,1251 1605,1605 = 1345,1345 1617,1647 = 1357,1387 1658,1671 = 1392,1405 1719,1721 = 1447,1449 1730,1737 = 1462,1469 1781,1782 = 1524,1525 1814,1814 = 1498,1498 1882,1882 = 1587,1587 1907,1907 = 1609,1609 1918,1922 = 1621,1625 1927,1930 = 1630,1633 1976,1976 = 1675,1675 1986,1987 = 1686,1687 2008,2008 = 1708,1708 2010,2010 = 1710,1710 2022,2032 = 1722,1732 2066,2070 = 1766,1770 2072,2072 = 1772,1772 2095,2101 = 1795,1801 2110,2112 = 1808,1810 2121,2128 = 1819,1826 2136,2170 = 1834,1868 2207,2207 = 1898,1898 2229,2249 = 1921,1941 2267,2272 = 1959,1964 2312,2314 = 1966,1968 2335,2363 = 1991,2019 2384,2384 = 2040,2040 2388,2404 = 2058,2074 2443,2443 = 2093,2093 2450,2451 = 2100,2101 2463,2465 = 2113,2115 2475,2475 = 2125,2125 2495,2497 = 2170,2172 2540,2544 = 2234,2238 2717,2734 = 2378,2395 2770,2770 = 2431,2431 2781,2795 = 2442,2456 2809,2813 = 2470,2474 2836,2838 = 2497,2499 2871,2874 = 2532,2535 2877,2890 = 2538,2551 2953,2957 = 2559,2563 3076,3076 = 2656,2656 3081,3086 = 2661,2666 3090,3098 = 2670,2678 3147,3151 = 2636,2640 3157,3162 = 2646,2651 3255,3266 = 2751,2762 3273,3274 = 2769,2770 3297,3301 = 2800,2804 3312,3320 = 2815,2823 3332,3333 = 2226,2227 3382,3391 = 2867,2876 3487,3487 = 2950,2950 3490,3508 = 2953,2971 3510,3513 = 2973,2976 3532,3538 = 2990,2996 3543,3566 = 2298,2321 3724,3726 = 2911,2913 3829,3840 = 3096,3107 3848,3848 = 3115,3115 3856,3876 = 3123,3143 3885,3887 = 3152,3154 3900,3904 = 3167,3171 3906,3906 = 3173,3173 |
| MSAprobes - Muscle | 889,899 = 651,661 925,933 = 680,688 1021,1039 = 744,762 1132,1148 = 820,836 1160,1178 = 849,867 1404,1416 = 1019,1031 1467,1468 = 1066,1067 1473,1475 = 1072,1074 1507,1519 = 1105,1117 1566,1569 = 1144,1147 1580,1603 = 1156,1179 1607,1616 = 1183,1192 1705,1715 = 1251,1261 1742,1759 = 1288,1305 1944,1972 = 1430,1458 1990,1997 = 1474,1481 2005,2006 = 1489,1490 2012,2021 = 1496,1505 2033,2063 = 1517,1547 2177,2191 = 1658,1672 2212,2221 = 1686,1695 2330,2334 = 1786,1790 2367,2378 = 1823,1834 |
3,151 = 3,151 878,887 = 640,649 906,910 = 667,671 1005,1009 = 731,735 1125,1130 = 813,818 1307,1308 = 944,945 1470,1471 = 1069,1070 1605,1605 = 1181,1181 1617,1629 = 1193,1205 1722,1738 = 1268,1284 1764,1781 = 1309,1326 1924,1930 = 1414,1420 1999,2001 = 1483,1485 2003,2003 = 1487,1487 2022,2063 = 1506,1547 2066,2070 = 1550,1554 2080,2090 = 1563,1573 2095,2101 = 1578,1584 2108,2116 = 1589,1597 2122,2169 = 1603,1650 2203,2204 = 1677,1678 2225,2276 = 1699,1750 2336,2365 = 1792,1821 2409,2427 = 1845,1863 2449,2452 = 1885,1888 2466,2474 = 1902,1910 2489,2564 = 1925,2000 2647,2649 = 2044,2046 2696,2732 = 2057,2093 2836,2838 = 2138,2140 2848,2861 = 2150,2163 3009,3014 = 2232,2237 3018,3019 = 2241,2242 3255,3276 = 2453,2474 3317,3323 = 2515,2521 3382,3388 = 2562,2568 3454,3472 = 2591,2609 3541,3613 = 2325,2397 3832,3838 = 2678,2684 3894,3906 = 2740,2752 |
Суммарная длина идентичных блоков между выравниваниями MSAprobes и MAFFT составила 254 колонки (6,50% от длины выравнивания MSAprobes и 8,00% от длины выравнивания MAFFT), а суммарная длина идентично выровненных колонок (с учётом колонок с гэпами) - 941.
Суммарная длина идентичных блоков между выравниваниями MSAprobes и Muscle составила 295 коллонок (7,55% от выравнивания MSAprobes и 10,72% от выравнивания Muscle). Суммарная длина идентично выровненных колонок (с учётом колонок с гэпами) - 1030.
На основании полученных данных можно сделать вывод, что результат работы программы Muscle в большей степени приближен к результату работы MSAprobes, чем результат программы MAFFT. Основной признак, указывающий на это - суммарная длина идентичных в сравниваемых выравниваниях блоков. Также можно заметить, что по уменьшению времени работы сравниваемые программы располагаются так: MSAprobes, Muscle, MAFFT.
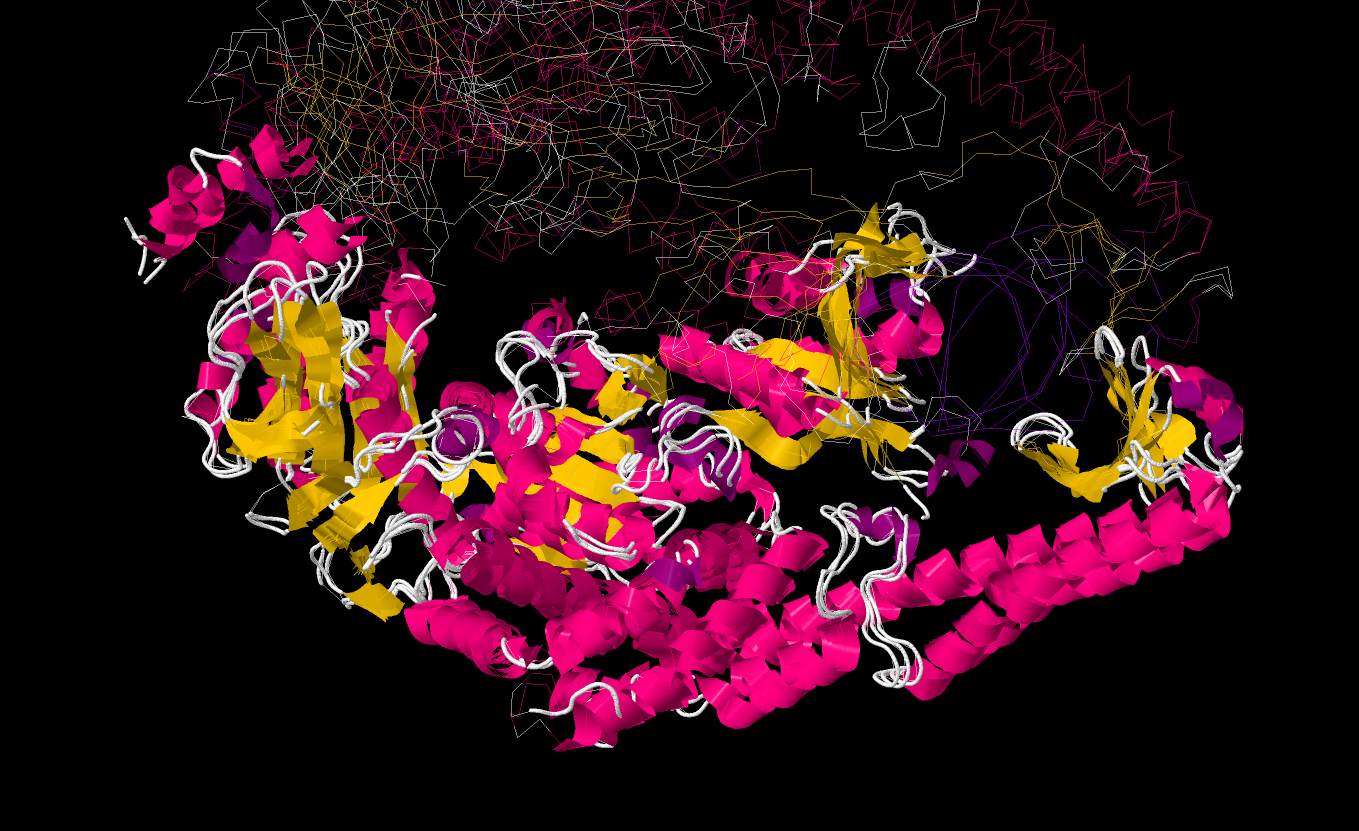
С помощью инструмента выравнивания структур PDB были совмещены структуры трёх белков семейства MutS (PDB ID: 1E3M (белок E. coli), 1EWQ (белок T. aquaticus), 5X9W (белок N. gonorrhoeae)).

Последовательности также были выравнены программой Muscle.
Проект Jalview с обсуждаемыми выравниваниями MutS.
Сравнение данных выравниваний показало, что общая протяженность идентичных блоков - 629 столбцов (77,65% от длины выравнивания по совмещению структур и 78,43% от длины выравнивания программой Muscle).
Таким образом, мы можем убедиться в том, что структура белка в эволюции действительно хорошо сохраняется, ведь эти 3 белка, судя по наложению, имеют весьма схожие структуры, хотя и синтезируются разными организмами и обладают отличными друг от друга последовательностями.
Muscle - эвристический алгоритм прогрессивного множественного выравнивания белковых последовательностей, разработанный Робертом Эдгаром. Полезную информацию о данной программе, а также ссылки для её скачивания, можно найти по ссылке. Последняя версия программы - Muscle5, но здесь речь пойдёт о Muscle3, т.к. она используется в Jalview.
Алгоритм работы программы:
1 этап - быстрое построение направляющего дерева. На данном этапе скорость важнее точности:
a). В последовательностях выделяют подпоследовательности длины k - k-меры. На основе сравнения k-меров разных последовательностей (родственные последовательности обладают большим числом общих k-меров, чем можно было бы наблюдать в результате случайных совпадений) составляется матрица расстояний для 'чернового' дерева (T1) (построение дерева по алгоритму UPGMA). Данная процедура не трбует проведения выравнивания, за счет чего достигается высокая скорость работы;
b). Прогрессивное выравнивание последовательностей по T1: сначала попарно выравниваются последовательности соседних свободных ветвей. Каждая колонка полученного выравнивания становится выравниваемым символом. Теперь производится выравнивание пар выравниваний, соответствующих соседним узлам и т.д. В результате получаем множественное выравнивание A1;
2 этап - A1 - неоптимальное выравнивание, т.к. дерево T1 не оптимально: расстояние, рассчитанное по k-мерам недостаточно точное. Так что теперь по полученному выравниванию A1 следует перерасчитать направляющее дерево для блоьшей точности:
a). По выравниванию A1 для каждой пары последовательностей перерасчитывается расстояние. Получаем новую матрицу расстояний;
b). По алгоритму UPGMA на основании новой матрицы строится новое направляющее дерево T2;
c). По дереву T2 производится прогрессивное выравнивание. Процесс занимает меньше времени за счёт того, что перерасчитываются лишь выравнивания для поддеревьев, у которых ветвление изменилось относительно T1;
3 этап - рафинирование:
a). Дерево T2 разделяется на два в результате удаления какого-либо выбранного ребра. Вычисляется профиль множественного выравнивания в каждом поддереве;
b). Новое множественное выравнивание создается путем повторного выравнивания полученных профилей;
c). Если вес нового выравнивания выше, чем вес A2, то новое выравнивание сохраняется. В противном случае новое выравнивание не сохраняется.
Шаги третьего этапа повторяются до тех пор, пока не будет получено выравнивание с оптимальным весом.
Таким образом, за счёт построения 'чернового' дерева на основании сравнения последовательностей с k-мерами и первичного выравнивания на основе этого дерева достигается достаточно высокая скорость работы алгоритма. Рафинирование кроме того сильно повышает точность выдаваемого результата.