A- и В-формы ДНК. Структура РНК
Для работы был использованы инструменты пакета 3DNA, который является одним из самых популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот. Используя программу fiber этого пакета были созданы файлы с А-, В- и Z-формой ДНК (gatc-a.pdb, gatc-b.pdb, gatc-z.pdb, соответственно).
Для визуализации структуры ДНК можно использовать программу JMol.
 |
 |
 |
 |
 |
 |
JMol позволяет выделить отдельные атомы, группировки. Используем для работы файл gatc-a.pdb, следовательно, на Рис. 7, Рис. 8, Рис. 9 представлена А-форма ДНК.
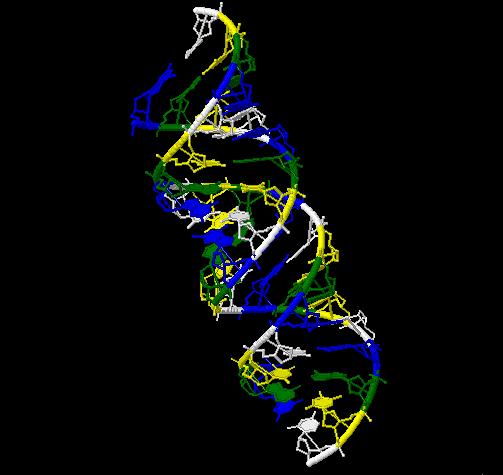
На Рис.7 разными цветами отмечены составляющие элементы ДНК (нуклеотиды):
- белым – дезоксицитидин-5-фосфат
- желтым – дезоксицитидин-5-фосфат
- зеленым – дезоксицитидин-5-фосфат
- синим – дезоксицитидин-5-фосфат
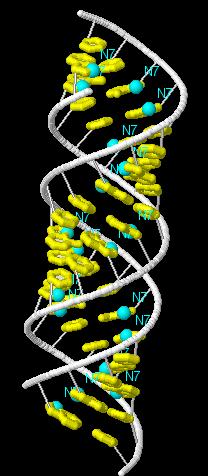
На Рис.8 белым цветом выделен сахорофосфатный остов ДНК. Желтым цветом выделены азотистые основания. Кроме того, светло-голубым цветом выделены и подписаны атомы азота в седьмых положениях в ароматических кольцах аденинов.
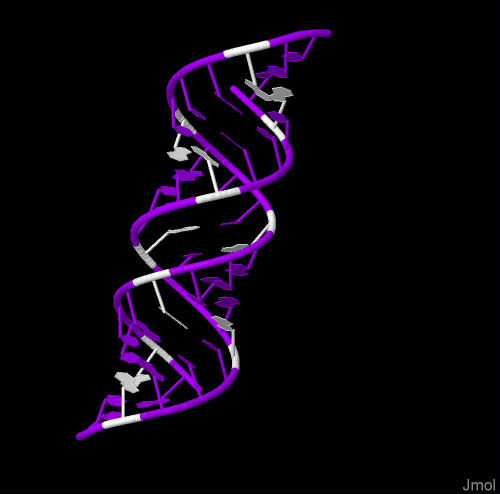
На Рис.9 белым цветом выделены аденины.
Перейдем к работе с PDB- файлами, которые были получены на сайте PDB. С помощью программы JMol было выяснено, что структуры не содержат разрывов.
С помощью программы JMol было выяснено, что структуры не содержат разрывов. На основе используемых файлов были сохранены координаты атомов только ДНК и РНК (DNA.pdb и RNA.pdb, соответственно).
 |
Далее был проведен анализ положения цитозина относительно большой и малой бороздок В-формы ДНК (файл gatc-b.pdb). Для этого был выбран цитозин в положении 36 [C]36. В сторону большой борозды обращены атомы N3, C4, N4, C5 (выделено лиловым цветом). В сторону малой борозды обращены атомы С2, O2, N1, C6 (выделено синим цветом). |
В А-форме положение атомов противоположно положениям в В-форме. В Z-форме расположение атомов аналогично положениям в В-форме.
С помощью инструментов JMol были получены параметры исследуемых трех форм ДНК (Таблица 1)
Таблица 1
| Тип спирали (правая или левая) | Шаг спирали (Å) | Число оснований на виток | Ширина большой бороздки (Å) | Ширина малой бороздки (Å) | |
| А-форма | правая | 28,03 | 10 | 18,5 (фосфаты [C]12-[A]18) | 9,63 (фосфаты [G]5-[G]29) |
| B-форма | левая | 33,75 | 10 | 20,58 (фосфаты [G]9-[T]27) | 13,2 (фосфаты [C]8-[C]36) |
| Z-форма | левая | 38,28 | 10 | 22,49 (фосфаты [G]1-[G]17) | 16,37 (фосфаты [C]6-[G]19) |
Теперь определим торсионные углы в А- и В-формах ДНК.
Торсионный угол – это угол вращения группы атомов относительно связи. Это набор геометрических параметров, который отражает конформацию полинуклеотидной цепи.
Таблица 2
| α P-O5' | β O5'-C5' | γ C5'-C4' | σ C4'-C3' | ε C3'-O3' | ζ O3'-P | χ C1'-N | |
| А-форма | -51,7 | 174,8 | 41,7 | 79,07 | 147,8 | -75,1 | -175,22 |
| B-форма | -29,9 | 136,4 | 31,1 | 133,3 | -140,8 | -160,5 | -99,0 |
| А-форма( из презентации) | -62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма( из презентации) | -63 | 171 | 54 | 123 /131 | 155 | -90 | -117 |
Параметры нуклеиновых кислот можно определить и с помощью инструментов пакета 3DNA. Но так как этот пакет работает только со старым форматом PDB, то для начала создаем файлы RNA_old.pdb и DNA_old.pdb с помощью программы remediator
Далее используем программу find_pair, определяет спаренные основания и положения спиралей в структуре. Полученные данные перенаправляем на вход программе analyze. В результате были созданы ряд файлов с описанием разных параметров структуры.
В файле DNA_old.outs есть информация о торсионных углах. В таблице 3 представлена эта информация, а также средние значения каждого из торсионных углов.
Для РНК можно определить больше параметров, так как в этой структуре можно встретить, например, стебли. Такие стебли можно найти с помощью программы find_pair, которая при работе с RNA_old.pdb дает на выходе файлы с отдельными спиральными структурами:
→RNA_old.inp_0001:
1 цепь: 1-7, 41-44
2 цепь (комплиментарная 1ой): 66-72.
→RNA_old.inp_0002:
1 цепь: 10-12, 7, 17-20
2 цепь (комплиментарная 1ой): 40, 25-20, 11-8.
→RNA_old.inp_0003:
1 цепь: 47-52
2 цепь (комплиментарная 1ой): 67-62.
→RNA_old.inp_0004:
1 цепь: 24-33
2 цепь (комплиментарная 1ой): 35-46.
Стебли:
- Акцепторный: 1-7 и 66-72
- Т-стебель: 49-52 и 62-65
- D-стебель: 10-12 и 23-25
- Антикодоновый: 37-44 и 26-33
Неканонические пары: U-G, C-C.
Есть 2 водородные связи, которые дополнительно стабилизируют третичную структуру тРНК.
В РНК возможно наличие стэкинг-взаимодействий - гидрофобных связей, которые возникают при таком расположении ароматических молекул, которое напоминает расположение монет в стопке и поддерживается ароматическими взаимодействиями (см. Викиверситет). Такие связи стабилизируют структуру.
Получить информацию о таких взаимодействиях можно с помощью программы find_pair, в результате работы которой будет получен файл staking.pdb с преобразованными координатами всех динуклеотидных пар, используемых для получения общепринятого изображения стэкинг-взаимодействий.
В файле RNA_old.out содержится информация о перекрывании пар оснований. Для удобства эту информацию выносим отдельно в таблице 4. в этой таблице синим цветом выделены максимальные значения, бирюзовым – минимальные значения, а зеленым – среднее значение. Стэкинг-взаимодействия будут тем сильнее, чем выше значение перекрывания.
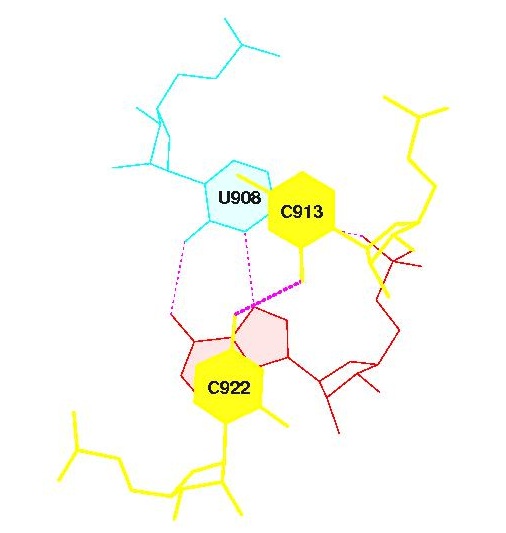
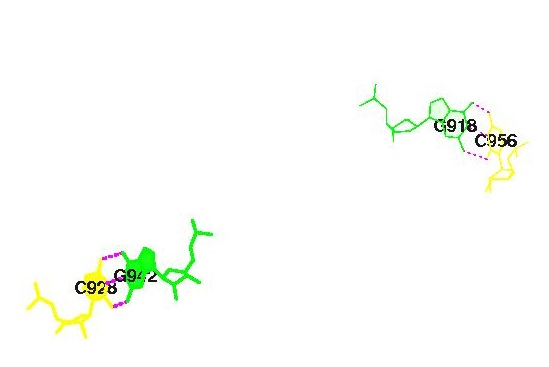
Рассмотрим 12ую пару (максимальное значение: 6,98). Пространственную структуру определяем из файла staking.pdb. Для удобства дальнейшей работы рассматриваемый блок выносим в новый файл stak_max.pdb (Рис. 11).
Таким же образом проанализируем 17ую пару (минимальное значение: 0,00). Рассматриваемый блок выносим в файл stak_min.pdb (Рис. 12).

Получить изображения стэкинг-взаимодействий можно и с помощью команд пакета 3DNA (Рис.13 и Рис.14).