Основные сведения о белке
Name: 2-dehydro-3-deoxyphosphooctonate aldolase
Название: 2-дегидро-3-дезоксифосфооктонат-альдолаза
Ниже приведены ссылки на файлы последовательности белка и его гена:
Таблица 1. Информация о белке 2-дегидро-3-дезоксифосфооктонат-альдолаза из организма Rhodobacter sphaeroides ATCC (идентификатор белка в базе данных RefSeq YP_001041787)
Информация получена с со страниц геномного браузера сайта NCBI:
http://www.ncbi.nlm.nih.gov/genome
| Вид информации | Квалификатор в записи генома | Значение |
| Локус гена в геноме | /locus_tag | Rsph17029_4073 |
| Имя гена | /gene | Rsph17029_4073 |
| Идентификатор гена в базе Gene | /db_xref | GeneID:4894991 |
| Начало гена в геноме | — | 11118 |
| Конец гена в геноме | — | 11954 |
| На какой цепи кодируется ген (прямая или обратная) | — | complement |
| Длина гена (в парах нуклеотидов) | — | 837 |
| Идентификатор белка в базе данных NCBI Protein | /protein_id | YP_001041787.1 |
| Длина белка (в аминокислотных остатках) | — | 278 |
Дополнительные сведения о белке (блок 2 второго семестра)
Таблица 2. Записи белка 2-дегидро-3-дезоксифосфооктонат-альдолаза в базах данных Uniprot, RefSeq, и PDB
| База данных | Идентификатор (ссылка) |
| Uniprot | AC: A3PS35; ID: A3PS35_RHOS1 (текст, web-страница) |
| Uniref50 | Uniref50_B9KXA3 (web-страница) |
| RefSeq proteins | YP_001041787.1 (текст, web-страница) |
| PDB | Точного соответствия Uniprot Retrieve/ID mapping не находит, ближайший аналог: 3FS2 |
История изменений записи A3PS35 Uniprot
Версия 1 датирована 2007-04-03.Последняя, 56 версия датирована 2015-03-11.
В новом описании:
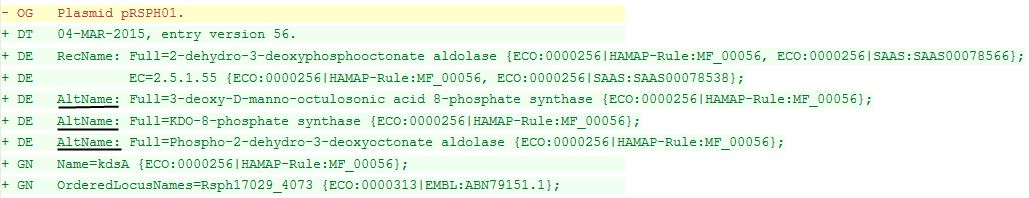
- Добавлено 3 альтернативных имени с сылками на достоверность:
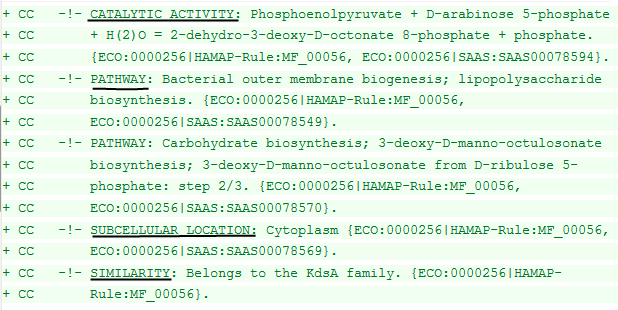
- Добавлено описание католитической активности, путей биосинтеза, расположение белка в клетке, и семейства белка, все со ссылками на достоверность:
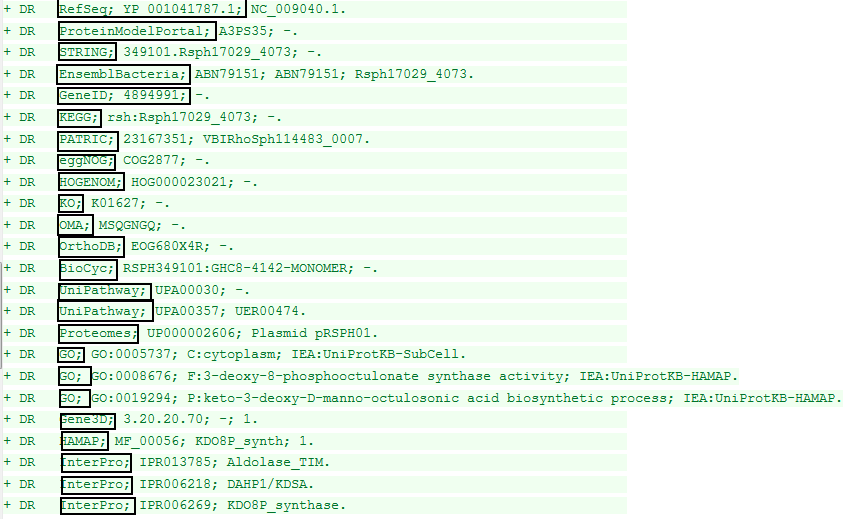
- Добавлено 24 ссылки на другие базы данных:
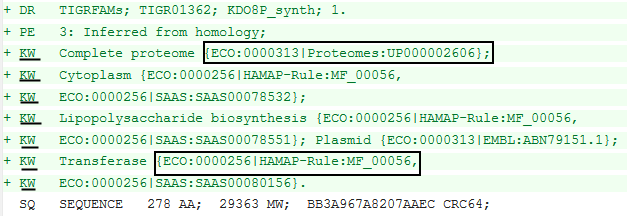
- Также добавлено 7 ключевых слов:
Множество гомологичных белков Uniref50_B9KXA3
Множество состоит из 5 последовательностей кластера Uniref50_B9KXA3 (AC коды: B9KXA3, Q3HKN7, A3PS35, F3U4A2, L1K8F6) и родственного белка, который имеет 3D структуру 3FS2 в базе PDB (AC код: Q8YHF1) и является таким же ферментом но в другой бактерии.Длина пяти последовательностей равна 277-278aa, и только длина опорной последовательности кластера (seed sequence), B9KXA3, равна 669aa.
Excel-таблица с атрибутами последовательностей множества белков: Uniref50_B9KXA3.xls.
Колонки "Features" и "Cross reference (PDB)" оказались пустыми для всех последовательностей кластера Uniref50_B9KXA3.
В колонку "Function [CC]" не попали данные ни из одной записи, тем не менее, в колонки "Catalytic activity" и "Pathway"
была успешно извлечена информация из соответствующих записей комментариев (CC) всех шести последовательностей.
Существенных отличий в найденных атрибутах этих белков нет (кроме длины, см. выше).
Способ аннотации нестандартных аминокислот в записи Uniprot
Поиск в разделе Help сайта Uniprot по ключевому слову selenocysteine находит ссылку на статью
Non-standard residue в которой приведены ссылки
на примеры аннотации позиций селеноцистеина и пирролизина в белке.
Нестандартные аминокислоты обозначаются меткой NON_STD в таблице свойств (feature table, FT строки).
Например, в записи P24183 есть следующая строка:
FT NON_STD 196 196 Selenocysteine.
Она указывает, что 196-я аминокислота белка - селеноцистеин.
В случаях, когда селеноцистеин одновременно является частью активного сайта белка, он аннотируется дважды: как нестандартный остаток и как активный сайт. Например, в записи P07203 это описано так:
FT ACT_SITE 49 49
FT SITE 49 49 Subject to oxidation and hydroselenide
FT loss to dehydroalanine. {ECO:0000250}.
FT NON_STD 49 49 Selenocysteine.
Сравнение аннотаций записей белка в Uniprot и Refseq Proteins
Форматы записей Uniprot и RefSeq сильно оличаются, тем не менее, большая часть информации о белке присутствует в обоих файлах.Файлы записей белка в формате Uniprot и RefSeq Proteins.
Примеры отличий в аннотациях:
- В записи Uniprot приведены альтернативные имена белка:
DE AltName: Full=3-deoxy-D-manno-octulosonic acid 8-phosphate synthase {ECO:0000256|HAMAP-Rule:MF_00056};
DE AltName: Full=KDO-8-phosphate synthase {ECO:0000256|HAMAP-Rule:MF_00056};
DE AltName: Full=Phospho-2-dehydro-3-deoxyoctonate aldolase {ECO:0000256|HAMAP-Rule:MF_00056};
- В записи Uniprot указано семейство, к которому принадлежит белок, его обычное местоположение в клетке, путь биосинтеза в котором он участвует:
CC -!- PATHWAY: Bacterial outer membrane biogenesis; lipopolysaccharide
CC biosynthesis. {ECO:0000256|HAMAP-Rule:MF_00056,
CC ECO:0000256|SAAS:SAAS00078549}.
CC -!- PATHWAY: Carbohydrate biosynthesis; 3-deoxy-D-manno-octulosonate
CC biosynthesis; 3-deoxy-D-manno-octulosonate from D-ribulose 5-
CC phosphate: step 2/3. {ECO:0000256|HAMAP-Rule:MF_00056,
CC ECO:0000256|SAAS:SAAS00078570}.
CC -!- SUBCELLULAR LOCATION: Cytoplasm {ECO:0000256|HAMAP-Rule:MF_00056,
CC ECO:0000256|SAAS:SAAS00078569}.
CC -!- SIMILARITY: Belongs to the KdsA family. {ECO:0000256|HAMAP-
CC Rule:MF_00056}.
- В записи Uniprot указано больше ссылок на другие базы данных (RefSeq содержит только ссылки на InterPro и GeneID).
- В записи RefSeq указана связь с координатами в нуклеотидной последовательности (Uniprot тоже ссылается на NC_009040,
но без координат):
CDS 1..278 /locus_tag="Rsph17029_4073" /coded_by="complement(NC_009040.1:11118..11954)"
- В записи RefSeq есть ссылка на Bioproject:
DBLINK BioProject: PRJNA58449