Для выполнения данного задания было необходимо выбрать один из контигов из сборки Х5 длиной от 20 кб до 100 kb.
Используя команду infoseq (с опциями -only -name -snucleotide1 -length) пакета EMBOSS, я вывелв только названия и длины контигов (для удобства выбора).
Среди контигов нужной длины я выбрала scaffold-51 длины 57241.
Затем получила его нуклеотидную последовательность: fasta-файл
Для работы программе AUQUSTUS требуются либо уже известные гены нашего организма, либо родственного. Чтобы устанавливать примерное таксономическое положение x5, нужно было обратиться к blastx.
Сразу было понятно, что искать весь контиг тяжело. Пришлось искать отдельные фрагменты длиной несколько тысяч п.н. Для начала я использовала фрагмент длиной в 14580 п.н. и оказалось, в последовательности была найдена часть некого гена.
Результаты blastx по запросу части скэффолда scaffold-51:
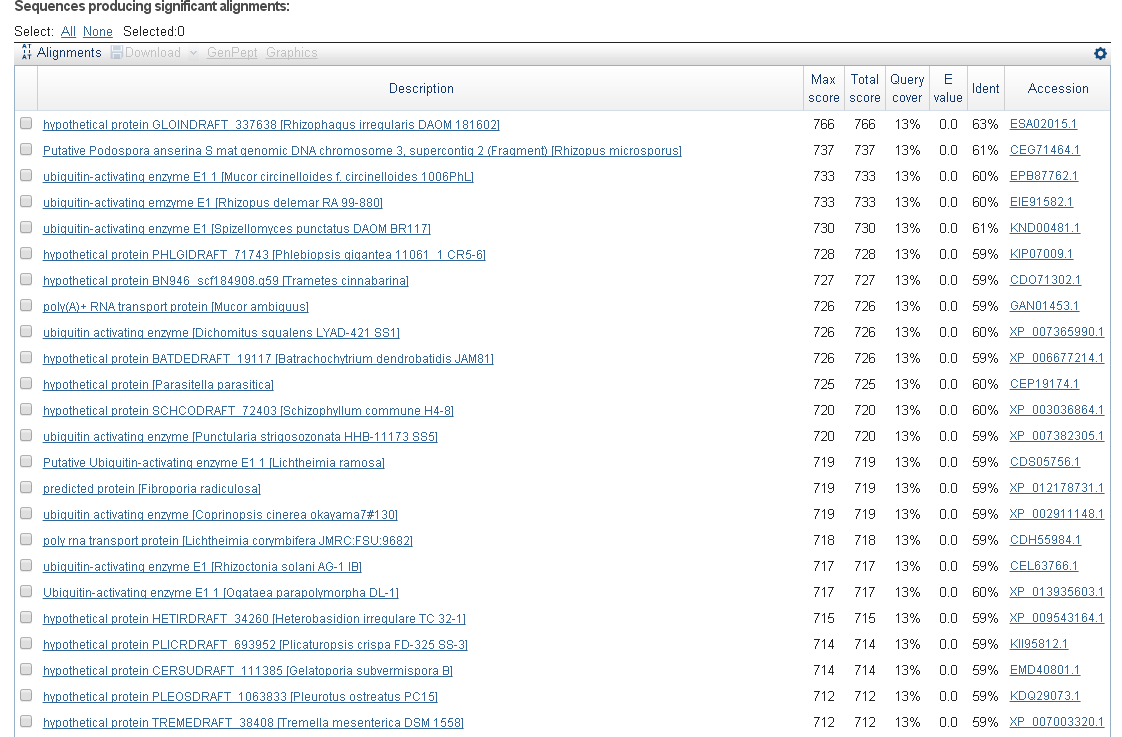
Лучшие находки:
К сожалению, уже после царства Fungi начинаются расхождения.
Пришлось обратиться к поискам по другим фрагментам нашего скэффолда. В целом результаты были похожи.
Я предположила, что подходящим организмом является Rhizopus delemar, т. к. он встречался при всех исследованиях фрагменотов.
Результат программы: на сайте
Архив содержал следующие файлы:
Предсказания генов в формате gff содержатся в файле augustus.gff .Там приведены предсказанные кодирующие нуклеотидные и аминокислотные последовательности, а также экзонно-интеронная структура предсказанных генов.
Проверка предсказания с помощью blastp
Первая находка имеет хороший score и подтверждена экспериментально.
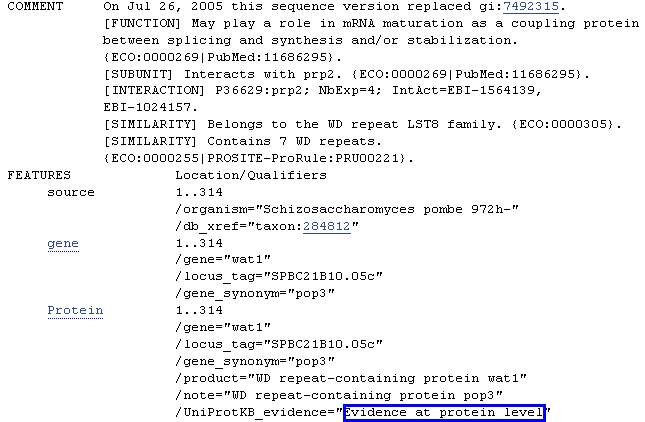
Судя по большому количеству гэпов, экзон-интронная структура предсказана не совсем правильно.
Скорее всего, аминокислотная последовательность гена содержит консервативный домен, принадлежащий суперсемейству Conserved Wat1/Pop3 WD-repeat protein, т.е. продукт гена предположительно участвует в защите целостности микротрубочек.
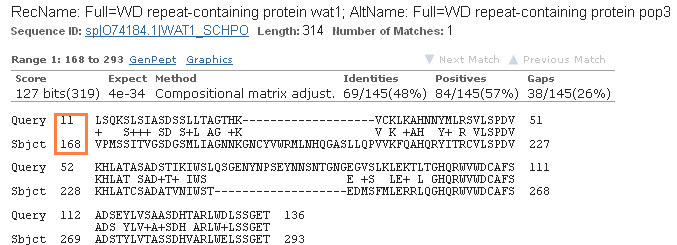
Из рисунка ниже видно, что участок гомологичной последовательности, сходный с анализируемой последовательностью, начинается со 168 аминокислоты (у остальных принмерно также). Поэтому начало гена 1 скорее всего было определено неверно, т.е. длина должна быть больше.
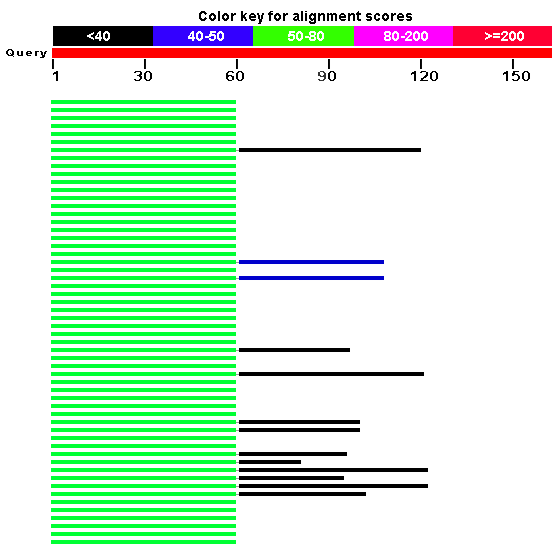
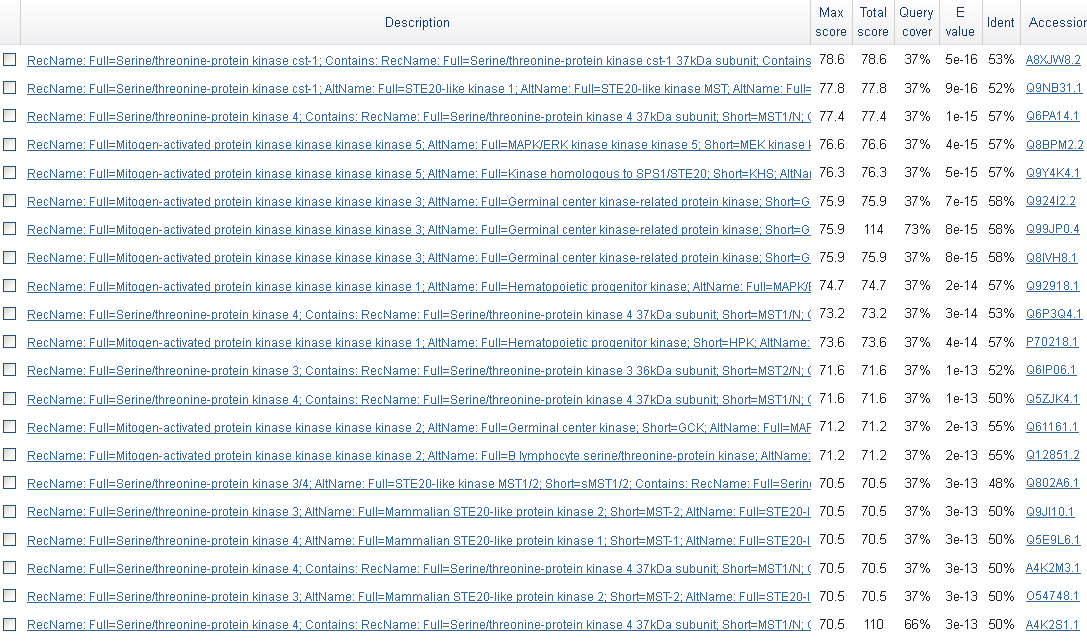
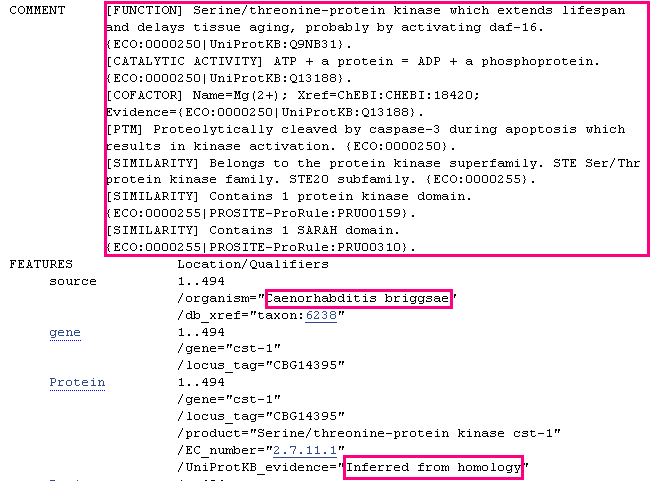
Для второго гена blastр выдал только одну находку, это 1-(5-phosphoribosyl)-5-[(5-phosphoribosylamino)methylideneamino] imidazole-4-carboxamide isomerase. Но находка имеет подтверждение только на уровне гомологии.
Для первой находки, которая имеет подтверждение только на уровне гомологии:
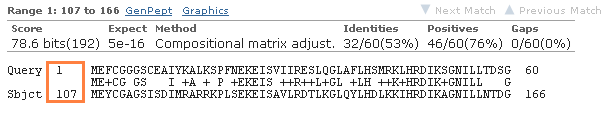
Вторая имеет также хороший score, но зато подтверждена экспериментально.
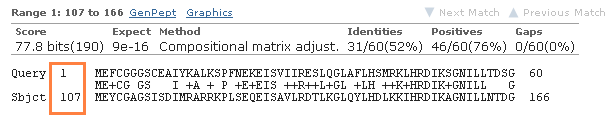
Таким образом, скорее всего ген 2 включает домен Serine/threonine-protein kinase cst-1. Т.е. предположительно yменьшает старение тканей, активируя daf-16.
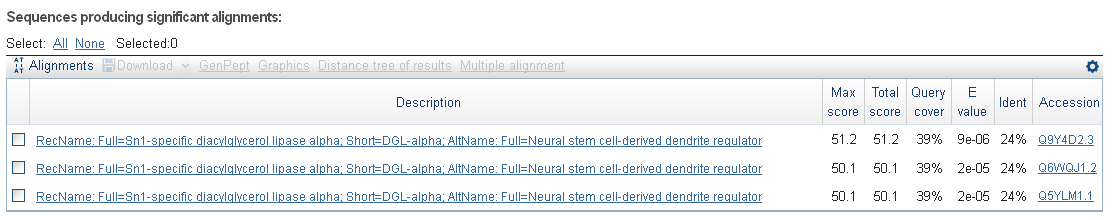
Опять начало гена 23 скорее всего было определено неверно, т.к. участок гомологичной последовательности начинается с 376 аминокислоты (у остальных принмерно также).
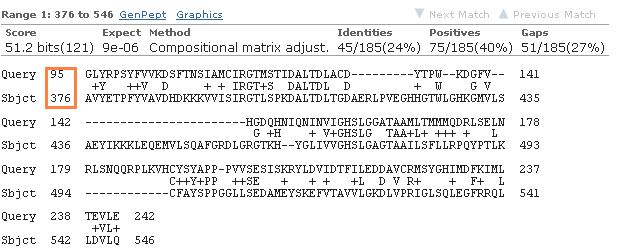
Ген 23 содержит домен, принадлежащий к суперсемейству Sn1-specific diacylglycerol lipase alpha, т.е. катализирует гидролиз диацилглицерола до 2-арахидоноил-глицерол.
Задание 2. Сравнение аннотации Refseq и AUGUSTUS одного гена человека.
Я выбрала ген MTHFR (methylenetetrahydrofolate reductase (NAD(P)H) [ Homo sapiens (human) ]) из NCBI Genes, организм H.sapiens.
Затем я нашла этот ген в UCSC Genome Browser, используя последнюю сборку генома hg38.
Координаты гена: хромосома 11,786,603-11,806,058
Далее требовалось сравнить аннотации Refseq и AUGUSTUS. Для этого я оставила только три трэка: base position, Refseq и AUGUSTUS.
Получила интрон-экзонную структуру в текстовом виде.
Результаты:
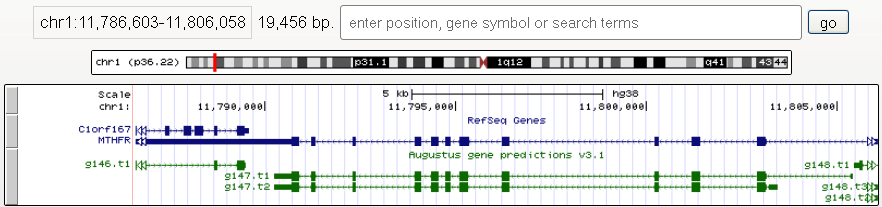
Таблица сравнения аннотаций Refseq и AUGUSTUS.
Выводы:
СПАСИБО ЗА ПРОСМОТР
© Мария Медведева