Практикум 1. Знакомство с PyMOL и электронная плотность
Задание 1. PyMOL
В данном задании работали со структурой, полученной в ходе работы консорциума PanDDA над способами связывания низкомолекулярных фрагментов с главной протеазой коронавируса Sars-COV-2 (PDB: 5rew). Мы искали остатки белка, с которыми потенциально взаимодействует лиганд T4M.
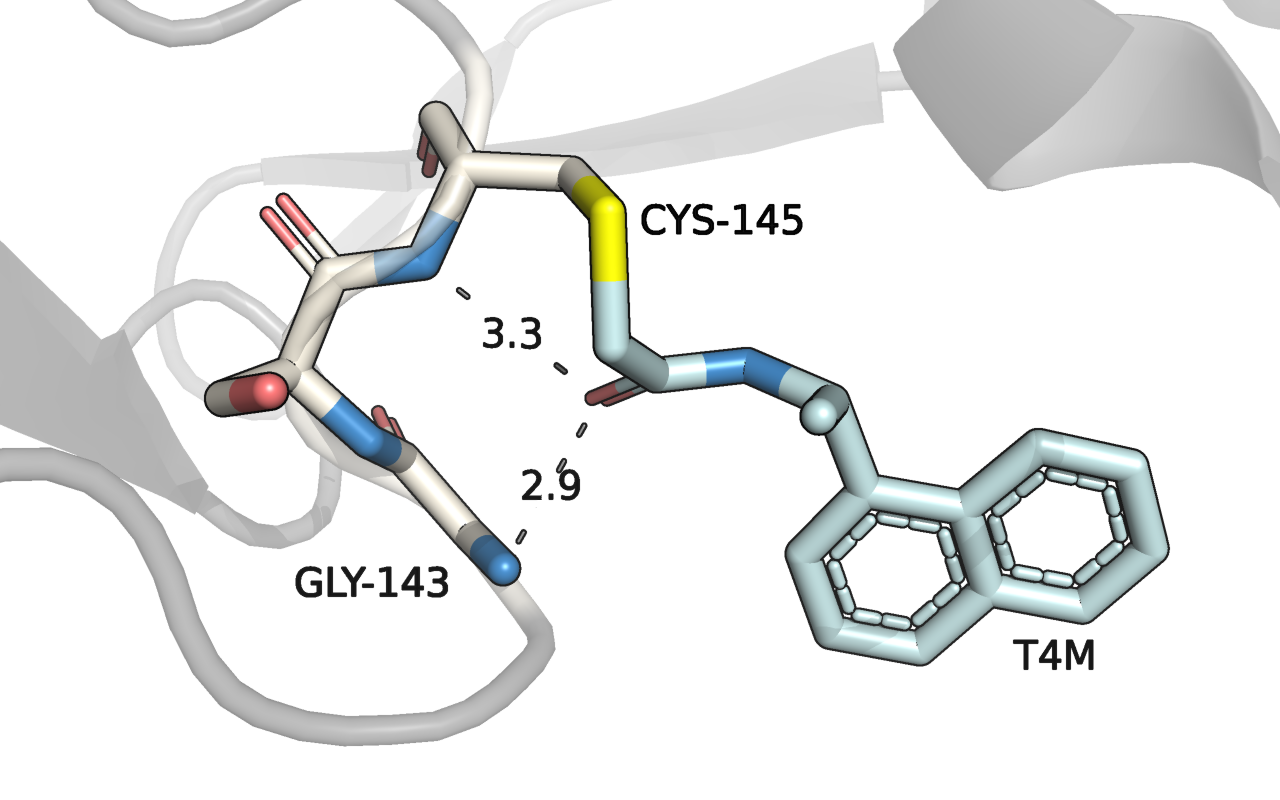
Лиганд T4M ковалентно связывается с Cys-145 протеазы (Рис. 1). Этот остаток - часть активного центра фермента, и наряду с Gly-143 формирует "оксианионный центр" (oxyanion hole), который необходим для стабилизации промежуточного комплекса [1]. Таким образом, связываясь с активным центром и образуя водородные связи с "оксианионной ямкой", лиганд может выступать в качестве потенциального ингибитора цистеиновой протеазы коронавируса.
Можете скачать сессию в PyMOL
Задание 2. ЭП: хорошая и плохая расшифровки.
В данном задании рассматривали кристаллографический расшифровки разного качества одного и того же белка.
Первой особенностью, которая сразу бросается в глаза - у 5yce больше молекул растворителя, чем у 108m. Видимо в структуре 5yce удалось различить больше отдельных молекул растворителя из-за лучшего качества расшифровки.
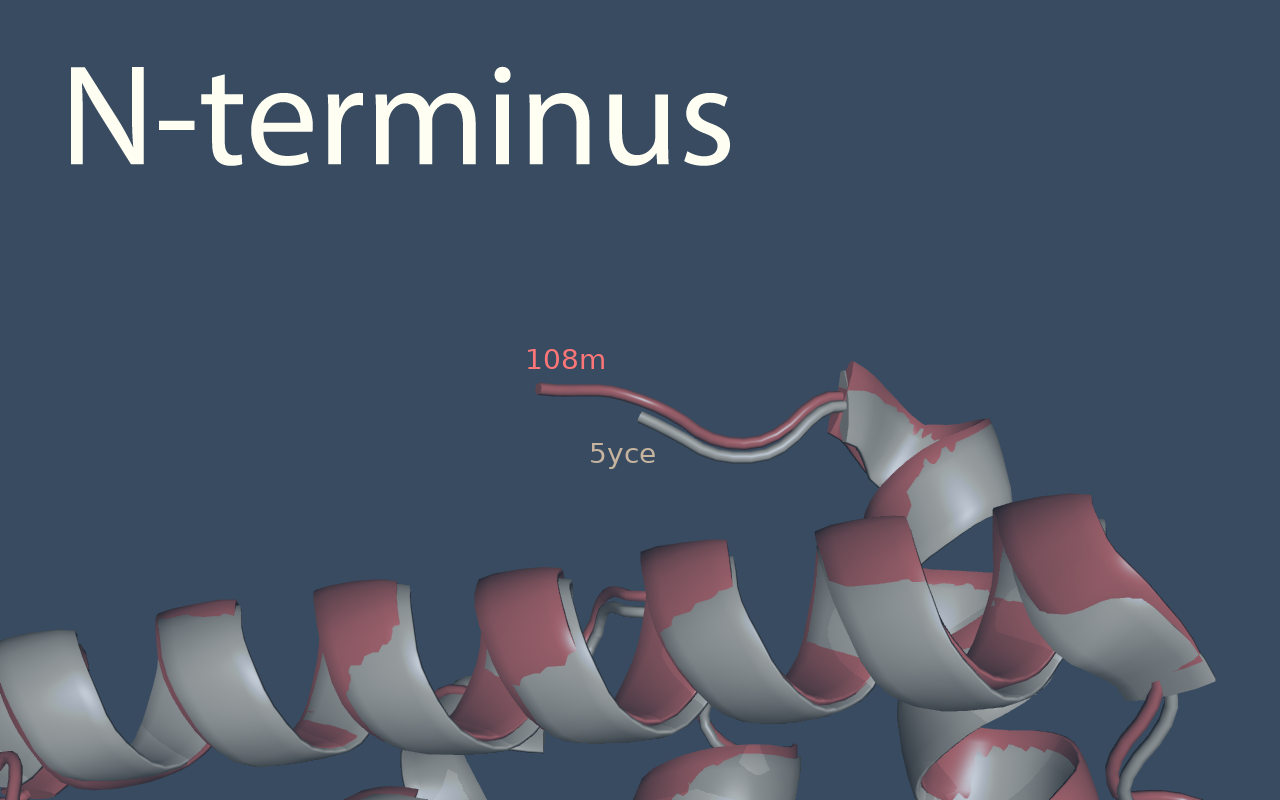
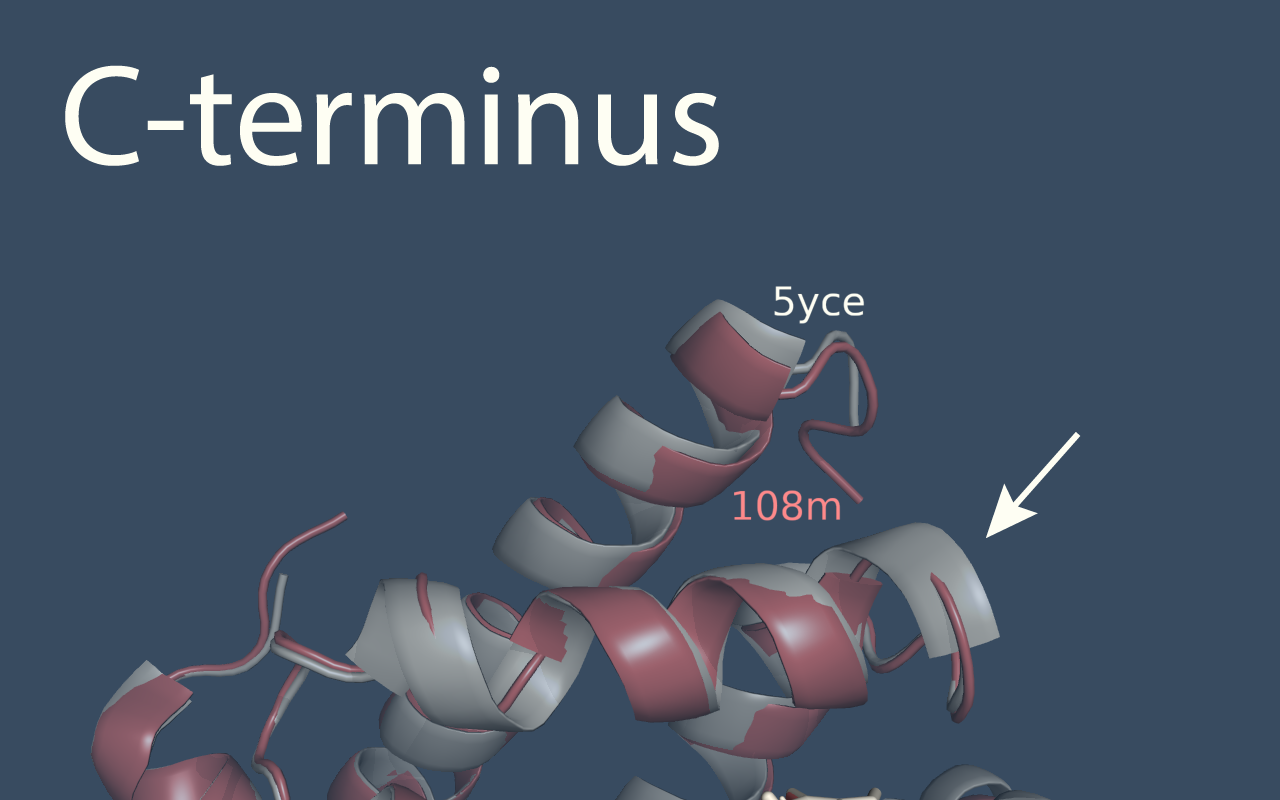
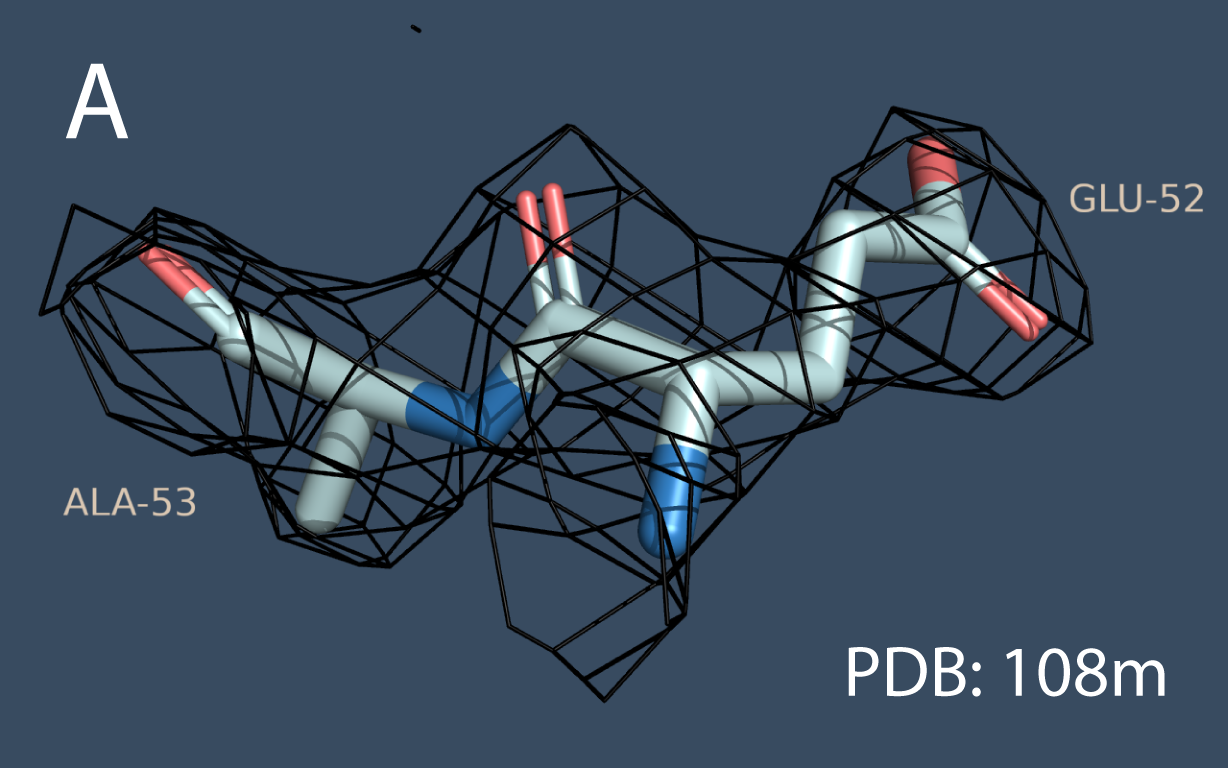
Второй особенностью является длина N- и C-концов белка - у 108m оба конца длинее (Рис.2 и Рис.3).
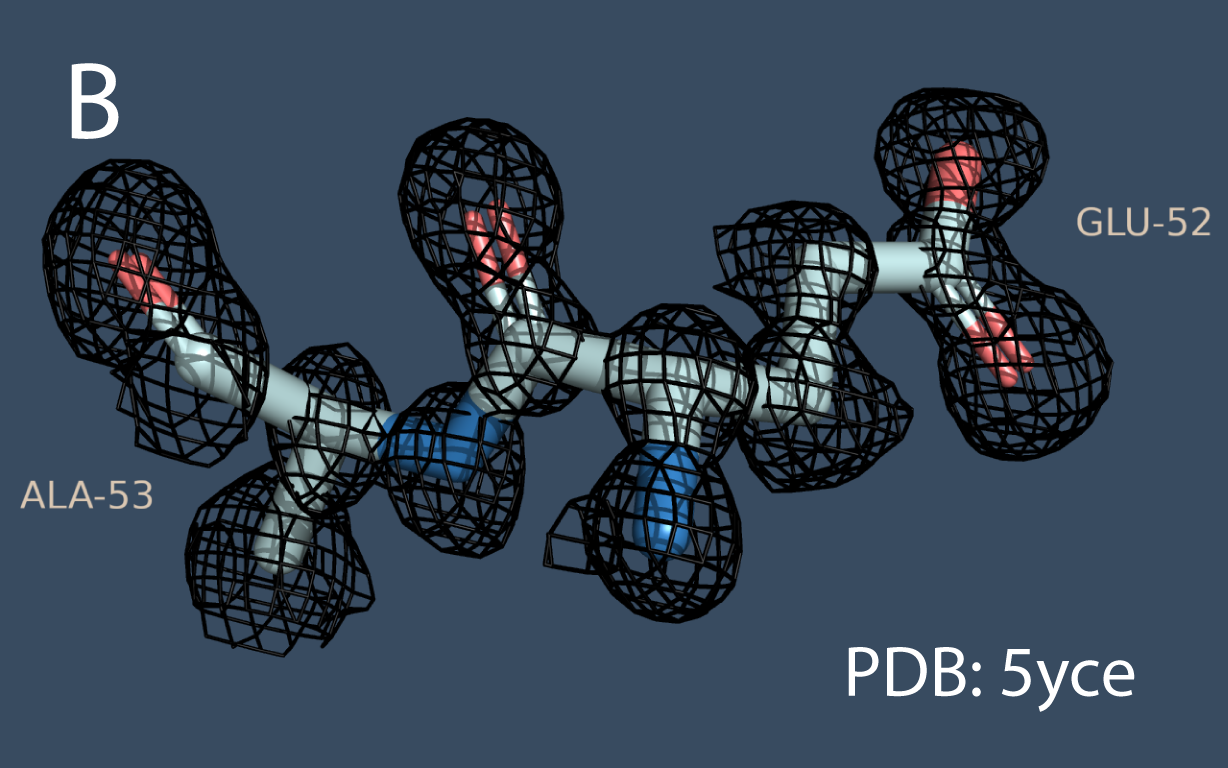
И, наконец, одна из альфа-спиралей длинее у 5yce (Рис. 3).
Открыв карты электронной плотности, мы увидим, что у 5yce лучше читаются формы отдельных атомов и сетка имеет больше полигонов, а 108m меньше полигонов, больше угловатых форм, и в целом карта выглядит неаккуратно. Это говорит в пользу того, что у 5yce разрешение лучше.
Таким образом, я предполагаю, что лучшее разрешение имеет структура 5yce, а не 108m, несмотря на то, что у 108m присутствуют все остатки в структуре. А ещё можно догадаться по номеру pdb, потому что чем больше номер, тем новее структура и скорее всего лучше разрешение. На сайте pdb разрешение 108m составляет 2.67 Å, а для 5yce - это 0.77 Å. Мои выводы полностью совпали с информацией на сайте.
Оба белка - это миоглобин кашалота. Обратите внимание на то, как отличается один и тот же регион в двух структурах. В структуре с лучшим разрешением (PDB: 5yce) больше полигонов, и они образуют чёткие сферы вокруг каждого атома (Рис. 4A). Структура с более плохим разрешением состоит из меньшего числа полигонов, нет чётких сфер вокруг каждого атома, напротив, карбоксильная группа глутаминовой кислоты находится в отдельном "мешочке", что не позволяет достоверно различить кислороды (Рис. 4B).
 |
 |
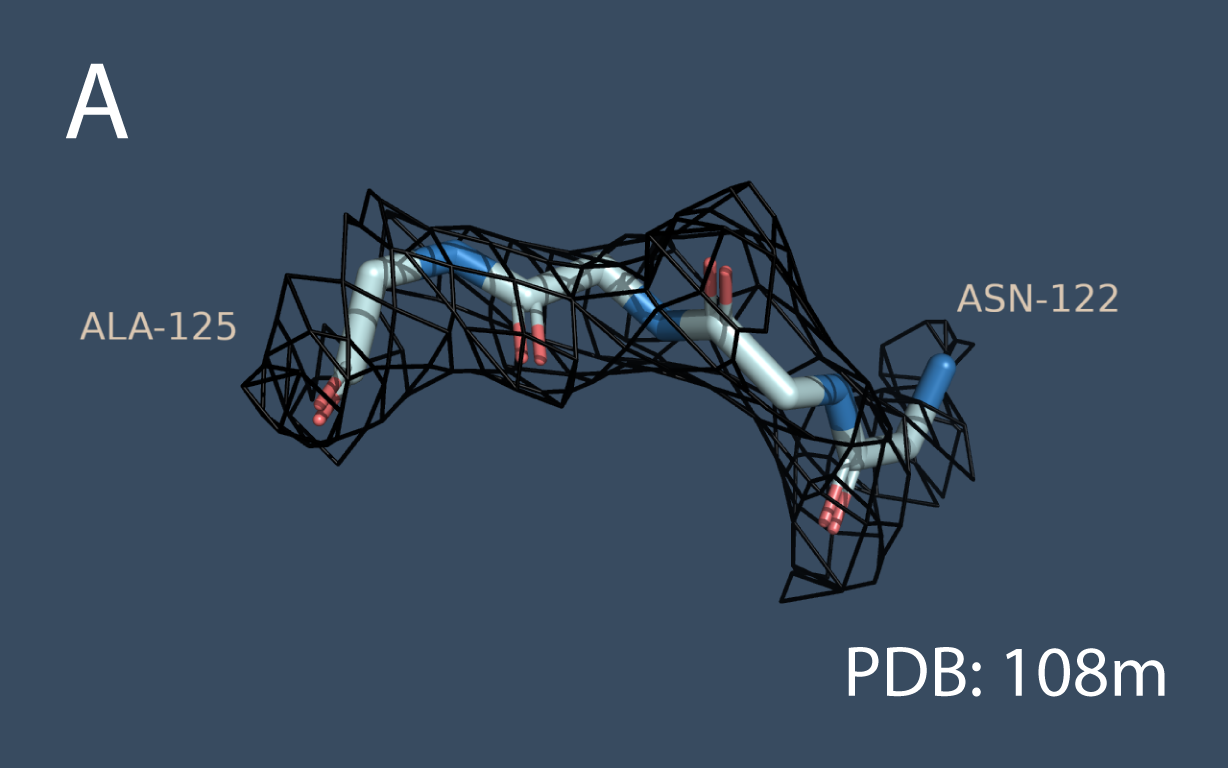
В задании написано, что нужно получить изображения остова. Нужно внимательнее читать задания, поэтому ниже будут правильные картинки (Рис. 5). Выводы аналогичны. Отмечу, что в 122 позиции у 108m точечная мутация - Asn, а у 5yce - Asp, но на изображение это не влияет, потому как ниже показаны только остовные атомы.
 |
 |
Задание 3. ЭП и положение в структуре.
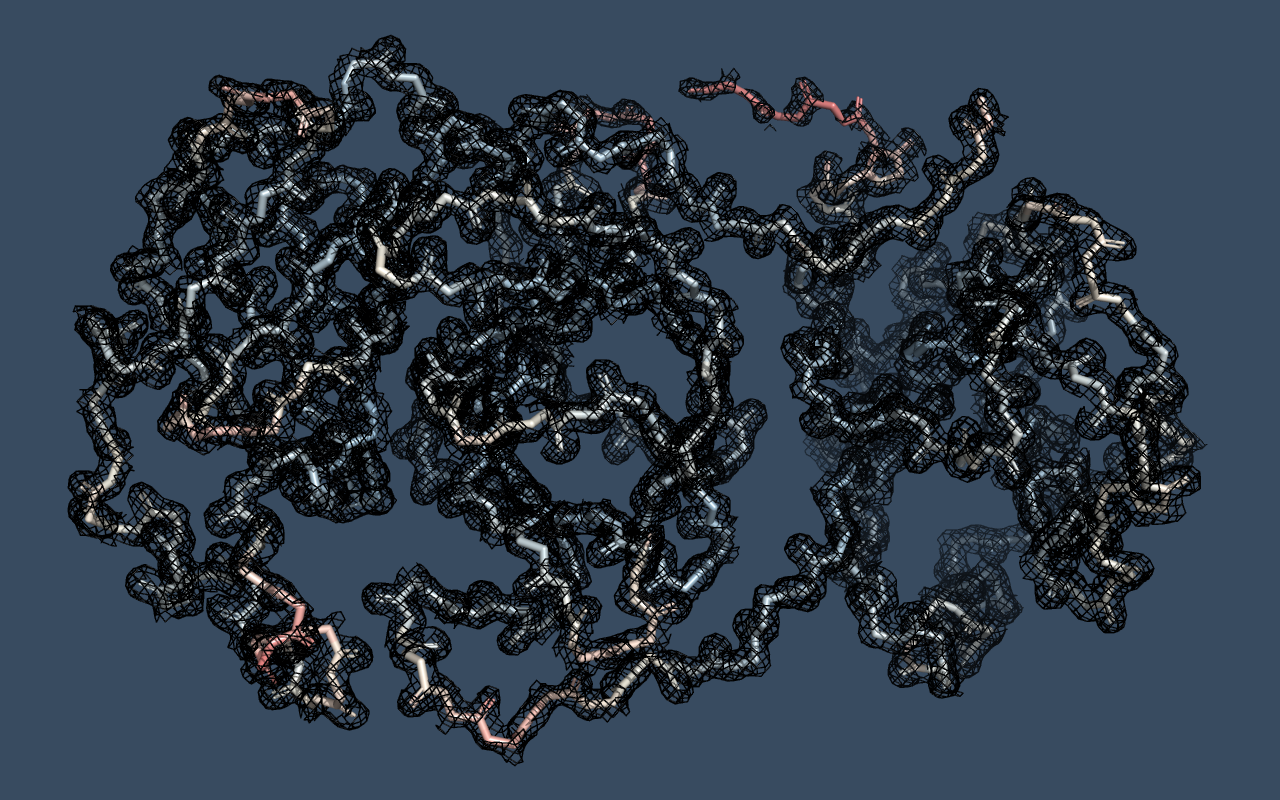
В данном задании использовался фрагмент протеазы SARS-CoV-2 (PDB: 5rew).
Заметим, что при переходе от 1 до 3 уровня подрезки некоторые регионы остова структуры первыми перестают быть покрытыми mesh'ем. Это концы белка и менее структурированные части (НЕ альфа-спирали и НЕ бета-тяжи), т.е. более подвижные части.
Подвижность оценить при помощи B-фактора. Он определяется, как:
Bi = 8π2Ui2, где Ui -
среднеквадратичное отклонение атома i. Т.е. чем более подвижен атом, тем больше "размазана" его электронная плотность и
тем выше B-фактор. Высокий B-фактор может быть обусловлен наличием нескольких стабильных конформаций или нагреванием
кристалла при облучении, которое вызывает тепловое движение. Обычно концы цепей имеют более высокий B-фактор, а плотно
упакованный домен - наоборот [2].
Уровень подрезки - это z-score, где в знаменателе среднеквадратичное отклонение плотности. Чем выше подвижность структуры, тем больше B-фактор и больше ср. отклонение, и тем меньше значение z-score. Поэтому 1) чем меньше z-score, тем больше mesh'а мы видим; 2) сначала перестают покрываться mesh'ем участки с высоким B-фактором (показано красным), что мы и наблюдаем в серии изображений ниже.
Задание 4. ЭП и типы атомов.
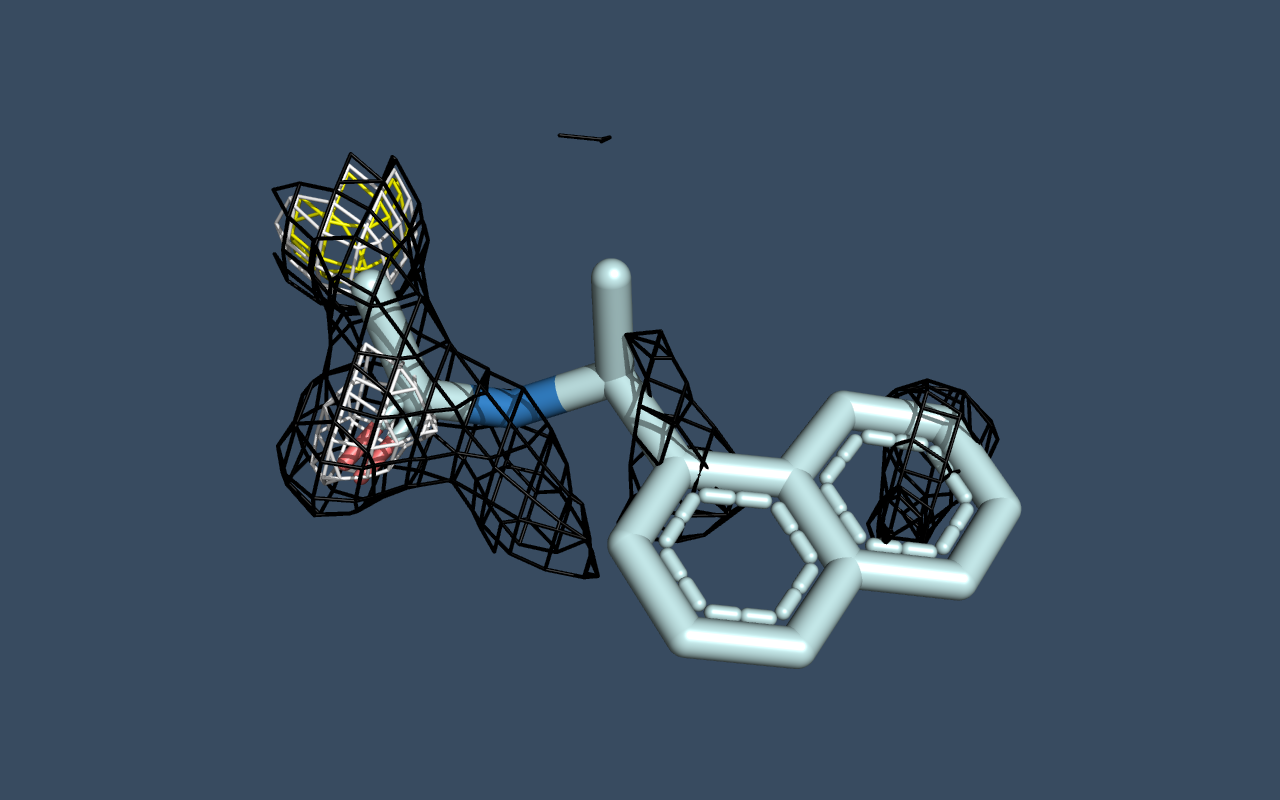
В данном задании продолжаем работать с фрагментом протеазы SARS-CoV-2 (PDB: 5REW), а именно с лигандом, который связывается с протеазой (Рис. 6). На уровне подрезки 1 покрыты не все атомы. Предположительно, это связано с тем, что лиганд связался не с каждой белковой молекулой в кристалле, поэтому суммарный сигнал меньше, чем от всего белка. На уровне подрезки 2 - остаётся mesh на кислороде и сере, которая является частью белка в составе Cys-145, что связано с тем, что кислород и сера, как элементы с высокой электроотрицательностью, оттягивают на себя электронную плотность. На уровне подрезки 3 mesh остаётся только на сере по вышеупомянутой причине. Покрытие при увеличении уровня подрезки, как и в задании 3 уменьшается.
Источники:
- Zhang L. et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors //Science. – 2020. – Т. 368. – №. 6489. – С. 409-412.
- Информация с сайта: https://proteopedia.org/wiki/index.php/Temperature_value
Список некоторых команд, чтобы я могла сориентироваться, как повторить сделанное.