Профили
Для построения профиля была выбраны белки архитектуры Malic_M + malic из таксона Proteobacteria из прошлого раздела.
Последовательности этих белков группируются в четко оформленную кладу на филогенетическом дереве.
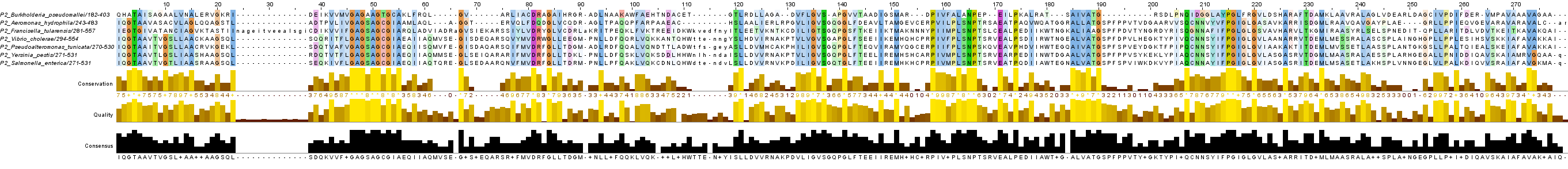
Рисунок 1. Выравнивание выбранных последовательностей домена Malic_M в архитектуре с доменом malic в таксоне Proteobacteria с раскраской ClustalX.
С помощью команды hmm2build по этому выравниванию был построен профиль. Затем профиль был откалиброван программой hmm2calibrate.Полученный профиль.
Затем был получен файл, содержащий все последовательности изучаемого домена (PF0349 Malic_M). Командой hmm2search был проведен поиск профилем по этому файлу.
Результат поиска.
Для оценки эффективности работы профиля был составлен файл ("золотой стандарт"), содержащий ID всех последовательнсотей домена в составе данной архитектуры в этом таксоне.
При пороге e-value = 1e-10 было првоедено сравнение списка находок по профилю с золотым стандартом. Таблица сравнения.
Оказалось, что были найдены все последовательности, входящие в "золотой стнадарт". Таким образом, можно определить следующие характеристики выдачи поиска по профилю:
- TP (true positives) = 176
- FP (false positives) = 1081 - 176 = 905
- FN (false negatives) = 0
Из этих параметров в совю очередь можно определеить чувствительность и избирательность:
- R = TP/(TP+FN) = 176/(176+0) = 1 (чувствительность)
- PPV = TP/(TP+FP) = 176/(176+905) = 0,16 (избирательность)
Такми образом, видно, что построенный профиль обладает 100%-ой чувствительностью (нашлись все последовательности, входящие в "золотой стандарт"), но очень малой избирательностью (нашлось слишком много лишних последовательностей).
© Марк Меерсон, 2014
Последнее обовление: 28.05.2014
