Поиск по сходству. BLAST, E-Value
1. Гомологичность белков
Для выполнения практикума я взяла белок пилин (ANW70673.1)
[1] организма Neisseria meningitidis [2] .
Дальше я провела поиск в BLAST, после чего выбрала 9 последовательностей (не считая изначальной) с достаточным E-value. Первый сеанс поиска
(длина слова - 6 букв, база данных - SwissProt) -выдал всего 7 находок. Второй сеанс (длина слова - 3 буквы, база данных - SwissProt) - уже большее
количество, но большинство из них были с очень низким E-value и идентичностью, названия абсолютно не соответствовали названию изначальной
последовательности. Я решила остановиться на следующих параметрах поиска: длина слова - 6 букв, база данных - RefSeq (первые две последовательности с высоким E-value я нашла в другом таксоне: Neisseria относится к b-proteobacteria, а поиск проводился по a-proteobacteria.
первые 13 результатов - последовательности с очень высокой идентичностью и одинаковым названием (все принадлежат Neisseria meningitidis). Есть подозрение,что это могут быть разные аннотации одого и того же белка, или очень схожие белки разных штаммов одного таксона, поэтому я не включила их в выборку. А вот эти включила:
Таблица 1. Основные данные о последовательностях (BLAST)
| ID |
Name |
Coverage (%) |
Identity (%) |
E-value |
Homology(+/-) |
| WP_060846362.1 |
peptide chain release factor-like protein (Methylobacterium aquaticum) |
61% |
28% |
0.19 |
- |
| WP_051312207.1 |
hypothetical protein (Pseudodonghicola xiamenensis) |
33% |
44% |
9,9 |
- |
| WP_002258782.1 |
pilin family protein |
93% |
97% |
3e-56 |
+ |
| WP_061734064.1 |
pilus assembly protein PilS |
98% |
82% |
6e-52 |
+ |
| WP_061818162.1 |
pilin |
97% |
77% |
7e-48 |
+ |
| WP_002225897.1 |
fimbrial protein MS11-D1 precursor |
98% |
77% |
6e-47 |
+ |
| WP_025460956.1 |
prepilin-type cleavage/methylation domain-containing protein |
94% |
75% |
4e-46 |
+ |
| WP_079758344.1 |
fimbrial protein P9-2 precursor |
98% |
75% |
4e-45 |
+ |
| WP_082299213.1 |
fimbrial protein (Neisseria gonorrhoeae) |
95% |
76% |
1e-44 |
- |
Для установления гомологии я провела множественное выравнивание беков:
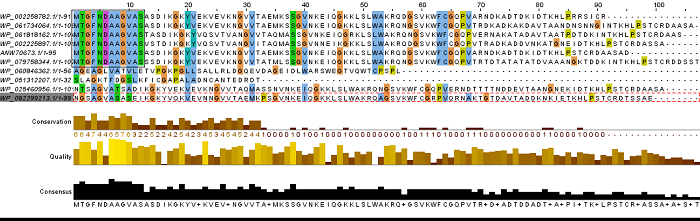 Из выравнивания можно сделать вывод о том, что чётко выделяются пять последовательностей, гомологичных исходной, на что указывает блок. Блок нашёлся всего один, но он имеет довольно чёткие границы, в отличие от остальных "претендентов на блок".
Последовательности в таблице стоят не в том же порядке, что последовательности в выравнивании, гомологичные отмечены "+" в соответствующей графе таблицы.
Из выравнивания можно сделать вывод о том, что чётко выделяются пять последовательностей, гомологичных исходной, на что указывает блок. Блок нашёлся всего один, но он имеет довольно чёткие границы, в отличие от остальных "претендентов на блок".
Последовательности в таблице стоят не в том же порядке, что последовательности в выравнивании, гомологичные отмечены "+" в соответствующей графе таблицы.
Проект выравнивания
2. Перестройки, карты локального сходства
Я выбрала две домаенные архитектуры из семейства LIM с помощью ресурса Pfam . Две доменные архитектуры, которые я сравнивала это:
1.PET and Zinc finger domain containing protein, {ECO:0000313|EMBL:CDJ85745.1}, 470 ак остатков.

2.LIM domain-containing protein jub, {ECO:0000313|EMBL:KRY15501.1}б 849 ак остатков.

E-value = 4e-19
Карта локального сходства выглядит так:
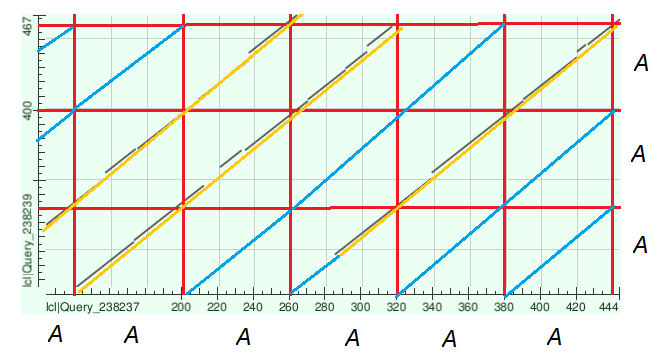
Интерпритация карты:
Скорее всего, карта была построена не по всей длине последовательностей, а по находкам, предположительно по чёрной границе:
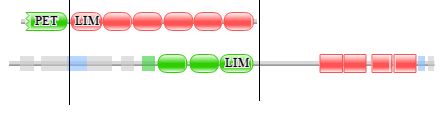
На карте локального сходства оранжевым обозначены находки BLAST, а голубым - то, что по логике вещей должно находиться. Таким образом, домен LIM, состоящий примерно из 60 аминокислотных остатков
и обозначенный на карте как "А", шесть раз встречается в первой доменной архитектуре, три раза - во второй. Это вполне соответствует картинке доменной архитектуры.
Было бы логично предположить, что эволюционное событие, которое иллюстрирует эта карта - это дупликация комплекса из 3х доменов LIM.
Источники:
1. Краткая информация о белке
2. Краткая информация о прокариоте
3. Презентация про BLAST
|