Для построения профиля была определена выборка из домена PF06456 у представителей (ранг: тип). Проводился поиск по Uniprot со следующим запросом: database:(type:pfam id:PF06456) taxonomy:"Chordata [7711]" AND reviewed:yes. Было найдено 18 последовательностей.
2. Построение профиля
Последовательности из Uniprot были выравнены в программе Jalview (Рис.1), выравнивание было отредактировано в программе вручную. Ссылка на проект выравнивания: uniprot.jvp
дальше я строила профиль:
>hmm2build profile.out uniprot.mfa - построение профиля.
>hmm2calibrate profile.out - калибровка профиля.
Полученный профиль: profile.out
3. Поиск по профилю в Swissprot
Необходимо было среди записей банка данных Swissprot найти последовательности, соответствующие построенному профилю. Командная строка:
>hmm2search -E 1000 -T -50 profile.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > find.out
Данные были записаны в таблицу(table.xlsx ). В таблице лист Uniprot содержит изначально найденные последовательности, лист Swissprot все расчеты.
4. Построение ROC кривой Были вычислены коэффициенты Sensitivity (True Positive/ (True Positive + False negative)) и Specificity (True Negative/(False Positive + True Negative)) Для разных выбранных порогов. По Sensitivity и 1-Specificity построена ROC кривая (Рис.1).
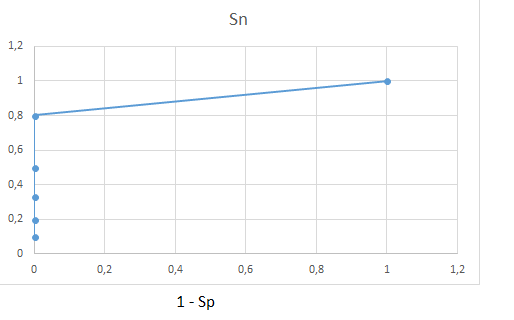
Рис.1 ROC-кривая
Выпадающий скор "выпадает" слишком сильно: -4,4 vs 529 у следующей находки. Я посчитала, что строить гистограмму скоров в данном случае не имеет смысла, порог виден, имеется одна ложная находка.
Вывод: изначально при поиске были указаны "плохие" параметры, из-за этого находки по swissprot и uniprot практически совпали. Поскольку ROC-кривая является отображением корректности классификатора (т.е. в данном случае HMM-профиля), она в данном случае имеет вид практически идеальной ROC-кривой.