Standalone BLAST
Поиск в геноме участков, кодирующих белки, похожие на заданный
Работать будем с белком TRMB_BACSU(AC: O34522), для которого стоит с помощью программы BLAST найти последовательность.
Аминокислотная последовательность trmb_bacsu.fasta была полученна с помощью команды:seqret sw:O34522 trmb_bacsu.fasta данного белка указана ниже:
>TRMB_BACSU O34522 tRNA (guanine-N(7)-)-methyltransferase (2.1.1.33) (BsTrmB)
(tRNA (guanine(46)-N(7))-methyltransferase) (tRNA(m7G46)-methyltransferase)
MRMRHKPWADDFLAENADIAISNPADYKGKWNTVFGNDNPIHIEVGTGKGQFISGMAKQN
PDINYIGIELFKSVIVTAVQKVKDSEAQNVKLLNIDADTLTDVFEPGEVKRVYLNFSDPW
PKKRHEKRRLTYSHFLKKYEEVMGKGGSIHFKTDNRGLFEYSLKSFSEYGLLLTYVSLDL
HNSNLEGNIMTEYEEKFSALGQPIYRAEVEWRT
Далее для файла gt_genome.fasta(файл с полный геном бактерии Geobacillus thermodenitrificans) созданим индексный файл пакеты BLAST+.
Используем команду: makeblastdb -in gt_genome.fasta -dbtype nucl
Теперь проведем поиск в полном геноме бактерии Geobacillus thermodenitrificans участков
кодирующие белки похожие на O34522. Воспользуемся командой tblastn -query trmb_bacsu.fasta -db gl_genome.fasta
-evalue 1e-03 -out t_g.txt и получим файл t_g.txt с информицией об участка.
Результаты приведены в Таблице 1.
 =
=
Таблица 1. Поиск гомологов белка TRMB_BACSU в геноме бактерии Geobacillus thermodenitrificans.
Поиск гомологов некодирующих последовательностей программой BLASTN
По этапам:
blastn -task blastn -query trna_bacsu.fasta -db gt_genome.fasta -evalue 1E-02 -outfmt 6 -out trna_gt.txt
получили файл trna_gt.txt время работы:real 0m0.645s
user 0m0.372s
sys 0m0.224s
grep ">" trna_bacsu.fasta > colon.xls мы поличили колунку состоящую из названий входных последовательностей
Поиск гомологов при изменённых параметрах программы BLASTN
Повторим предыдущии этапы еще два раза, но с другими параметрами. В первый раз изменим весовую матрицу, то есть параметры -reward и -penalty. Установим -reward 5 и -penalty -4 + поменяем параметры -gapopen и -gapextend.
Во второй раз, оставив те же (изменённые по сравнению со значениями по умолчанию) значения параметров -reward, -penalty, -gapopen и -gapextend, поменяем также значение параметра -word_size на минимально возможное(-word_size 4).
blastn -task blastn -query trna_bacsu.fasta -db gt_genome.fasta -evalue 1E-02 -word_size 11 -outfmt 7 -reward 5 -penalty -4 -gapopen 8 -gapextend 6 -out 2tra.txtreal 0m0.780s
user 0m0.480s
sys 0m0.216s
blastn -task blastn -query trna_bacsu.fasta -db gt_genome.fasta -evalue 1E-02 -word_size 4 -outfmt 7 -reward 5 -penalty -4 -gapopen 8 -gapextend 6 -out 2_2tra.txt
real 0m40.808s
user 0m40.488s
sys 0m0.240s
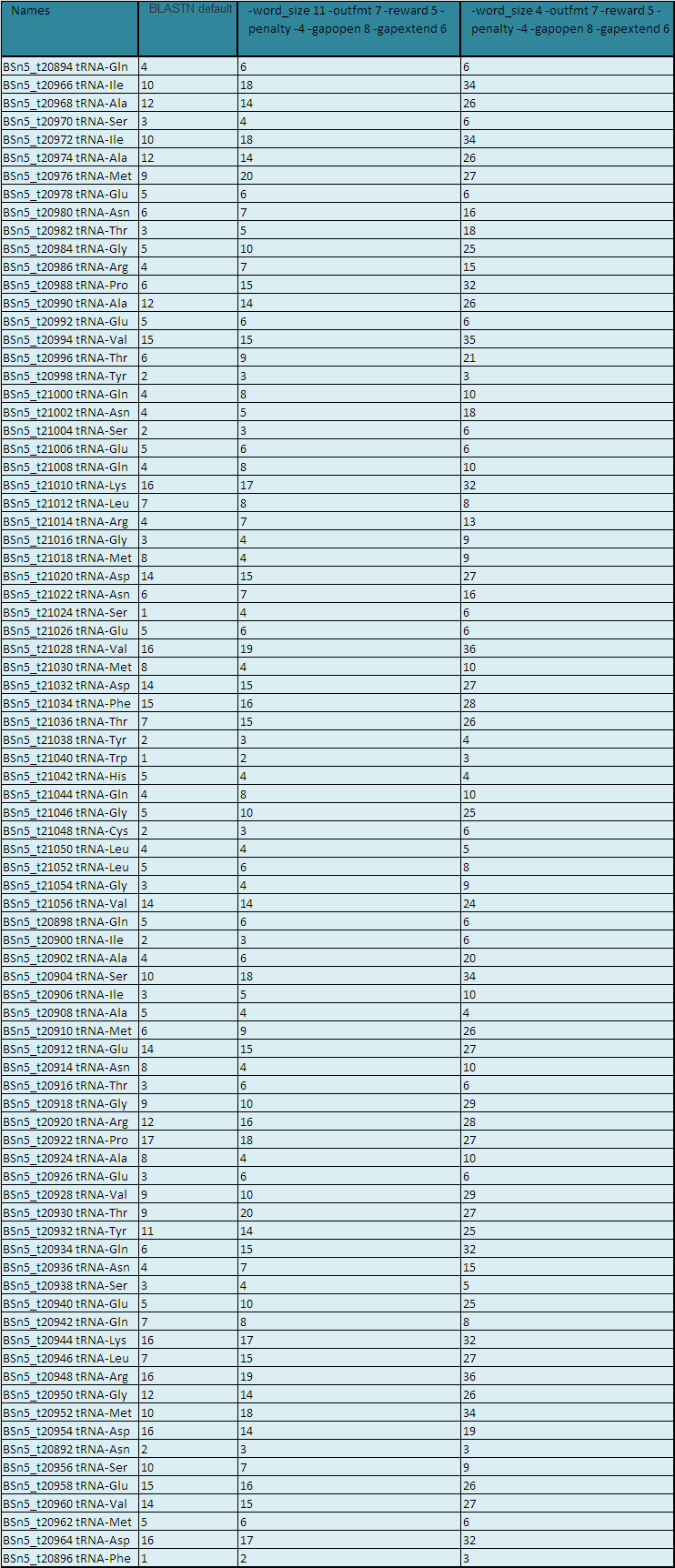
Совместные результаты приведены в Таблице 2.
 =
=
Таблица 2. Поиск гомологов белка TRMB_BACSU в геноме бактерии Geobacillus thermodenitrificans с разными параметрами.
Анализ результатов
Из Таблице 2 видно, что с изменением параметров колличество гомологичных последовательностей увеличивается. Это все благодаря тому, что зменения такого рода приводят к увиличению чувствительности и улучшению поиска(можно найти более далеких гомологов, имеющих короткие участки совпадения последовательностей).
При поиске с -word_size 4 мы получаем гомологи, которые не находятся при поиске с -word_size 11. В ходе поиска использоваласись
следующие команды:
seqret fasta::gt_genome.fasta[155340:155393] fasta::1_golomogy.fasta координаты с 155340 по 155393 в бактерии Geobacillus thermodenitrificans
seqret fasta::trna_bacsu.fasta:BSn5_t20966 fasta::1_trna.fasta
needle 1_golomogy.fasta 1_trna.fasta -out alig.txt полученное выранивние приведено ниже:
# Aligned_sequences: 2
# 1: CP000557
# 2: BSn5_t20966
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 77
# Identity: 42/77 (54.5%)
# Similarity: 42/77 (54.5%)
# Gaps: 23/77 (29.9%)
# Score: 151.5
#
#
#=======================================
CP000557 1 -------tagctcagc--ggtagagcaaccggctgttaaccggtaggtcg 41
||||||||| |.||||||...||.|||.|||.||..||||||
BSn5_t20966 1 gggcctgtagctcagctggttagagcgcacgcctgataagcgtgaggtcg 50
CP000557 42 taggttcgaatcc-------------- 54
..|||||||.|||
BSn5_t20966 51 gtggttcgagtccactcaggcccacca 77
Как и следовало ожидать, совпадающих участков не очень много, но назвать выравнивание плохим, достаточно сложно. Так как нет особо больших консервативных участков (максимально 3 триплета) и большое количество гепов участки на концах(последовательность нуклеотидов, а не белков, у кторох концевые участки в большенсве своем всегда различны), можно пологать что эти последовательности являются очень далекими гомологами друг другу. Проаннотировав, гомологичный участок в поле FT записи EMBL, было получено, что продуктом этого гена(155333..155407) является tRNA-Asn.
Время работы программы BLAST
Как же измененяется время работы программы при изменении параметров? Для этого при запуске на kodomo в командной строке перед всей командой написала слово time(это все есть выше).
Время работы увеличивается при добавлении команд связаных с укорочением длины слова при поиске. При изменении весовой матрицы, работа программы меняется не столько значительно.