Эволюционные домены
Для работы был выбран белок Human carboxypeptidase N (CPN), человеческая карбоксипептидазы N, PDBID: 2NSM, EC: 3.4.17.3, ID UniProtKB: P15169 (CBPN_HUMAN). Выбраный по этому белку домен - Zinc carboxypeptidase, цинк-карбоксипептидазы. Это семейство цинк-зависимые экзопептидазы, отщепляющиих С-концевые аминокислотные остатки от пептидов. Этот домен также называется Peptidase_M14. Ниже представленно изобажение располежения доменов на примере 2NSM.
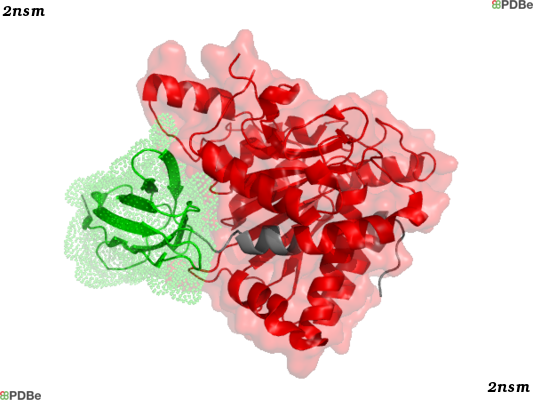
Изображение 1 Структурное представление доменов. На белке P15169 (CBPN_HUMAN), структура 2NSM, красным отмечана область принадлежещая домену PF00246 Peptidase_M14; зеленым отмечана область PF13620, Carboxypeptidase regulatory-like domain, регуляторного домена; серым отмечан область не относящясяя к доменам.
Доменн Peptidase_M14 имеет следующее кратнкое описание:
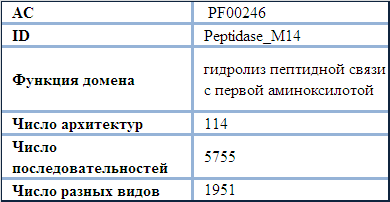
Таблица 1 Описание доменна Peptidase_M14
Далее с помощью JalView было построенно выравнивание белков семейства доменов. Для этой цели использовалась база данных PFAM(Seed), т.к. PFAM(Full) выдавал ошибку. Раскрашивание проводилось по цветовой схеме Clustalx с параметром by conservation=20. К белку CBPN_HUMAN было влючена структура 2NSM, на которой были отмечанны, консервативные позиции. Выравнивание как проект в формате .jar нахожится здесь, как выравнивание в формате fasta здесь.
Далее были выбранны две архитектуры, первая содержит 921 последовательность, вторая содержит 299:
Изображение 2 Архитектура 1. Красным отмечан домен Propep_M14 (PF02244), зеленым Peptidase_M14 (PF00246)
Изображение 3 Архитектура 2. Синим отмечан домен CarboxypepD_reg (PF13620), зеленым Peptidase_M14 (PF00246)
Немного о выбранных архитектурах.

Таблица 2 Характеристика выбранных для работы архитектур Pfam
Для составления сводной таблицы был получен список последовательностей, в которых содержится исследуемый домен. Для этого был использован скрипт swisspfam-to-xls.py:
python swisspfam-to-xls.py -z -h PF00246 -o res.txt
python uniprot-to-taxonomy.py -i flat.txt -o taxonomy.txt
Полученная сводная таблица.
Описание выбранного таксона и его подтаксонов
В качестве таксона были выбраны 1)Eukaryota, субтаксоны - Metazoa, Fungi и 2) Eukaryota, субтаксоны - Metazoa и Bacteria, субтаксон - Bacteroidetes(см. вышеприведенную таблицу(раздел "Выбранные"))
Выбор представителей архитектур
В JalView были загружены выбранные последовательности (18 для превой архитектуры и 11 для второй), выравнены алгоритмом Muscle with Defaults и раскрашены цветовой схемой ClustalX by Conversation (10%). Далее выравнивание было подчищено от пустых колонок и последовательностей, имеющих ярко неправильное выравнивания(в консервативной области - гэпы; где одни гэпы - большой кусок последовательности).
К выравниваиям была добавленна разметка по 3D структуре. В первой архитектуре разметка строилась на белке CBPA1_BOVIN(PDB: 1ARL), во второй архитектуре на CBPN_HUMAN (PDB: 2NSM).
Выравнивние первой архитектрый в формате jar и выравнивниае второй
Из выравнивание видно, что в целом белки достаточно консервативны в облостях вторичных структур. Так же стоит отметить, что в этих местах наблюдается располежение домена Peptidase_M14. Инетесно, что домен Propep_M14(не отмечан на структуре т.к. 1ARL, структра фермента в пост-пропептидном состоянии с открытым активным центром) также находится в менее, но все же немного консервативной области. Другими словами, "затычка" активного центра, схожа у ферментов первой архитектуры. Со второй архитектурой тоже много чего любопятного. Бактериальные белки намного длиннее(по этому не был удален их С-концевой хвост), что определенно выделяет их среди других. Второй домен, CarboxypepD_reg, находится в консервативной области. Из этого можно сделать вывод, что врероятней всего(т.к. большенсто белков регуляторные) домен непосредственно является ведущим в проведении катализа.

