Реконструкция филогенетических деревьев
Таксономия бактерий выбраных в пердыдущем задании.
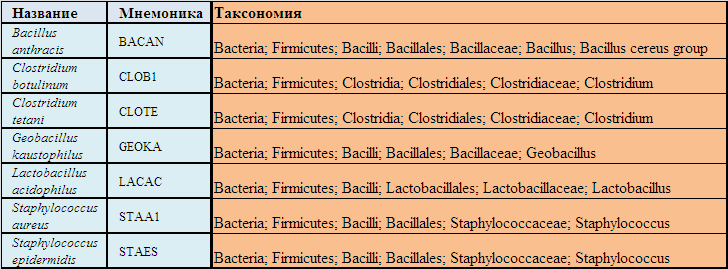
Таблица 1 Таксономия отобраных бактерий.
Для полученого ранее дерева, были отобраны некоторые таксоны: Clostridia и Bacilli(класс(class)); Bacillales и Lactobacillales(порядки (order)); Bacillaceae и Staphylococcaceae(семейства(family)). Их расположение на дереве приведено ниже:
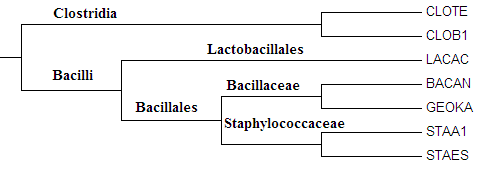
Изображние 1 Таксономия отобраных бактерий
Построение дерева по белку
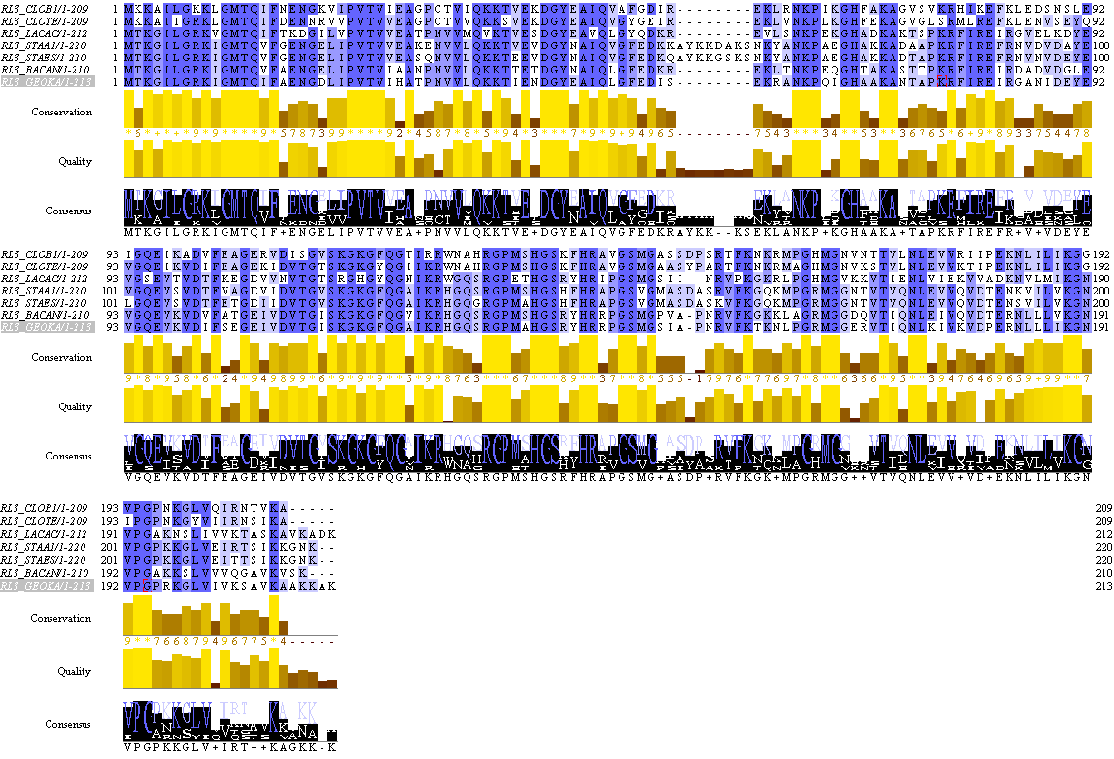
Для реконструкции филогенетического деревы были использованы семейсво рибосомных белоков L3 (Мнемоника: RL3). С помощью мнемоники были получены 7 fasta-последовательностей белков этого семейства, относящихся к выбраным ранее, 7 бактериям. Полученный, объединеный файл всех последовательностей пошел на вход програмам выравнивания: muscle и mafft. Было получено два множестевнных выравнивания, через разные программы. (выравнивания различаются, лишь в одном месте. по RL3_STAA1: c ASP(56) по LYS(67). На этом участке две последовательности(RL3_STAA1 и RL3_STAES) имеют небольшой участок, отсудствующий у других 5 (в 10 аминокислот). Этот участок заканчивается и начинается по разному у каждой программы и отличается на две аминокислоты(одна в лево, одна в право) Выравнивание построенное программой muscleпредставленно ниже.
По данному выравниванию, были построены филогенетические деревью, с помощью 4 различных алгоритмов:
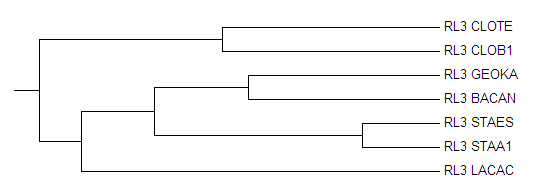
Изображние 3 Дерево построенное при помощи алгоритма "Average distance using % identity"
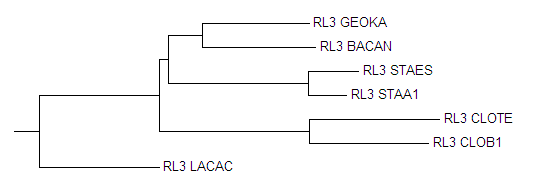
Изображние 4 Дерево построенное при помощи алгоритма "Neighbour joining tree using % identity"
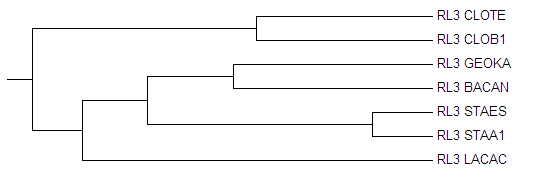
Изображние 5 Дерево построенное при помощи алгоритма "Average distance using blosum62"
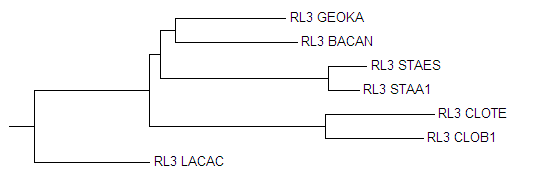
Изображние 6 Дерево построенное при помощи алгоритма "Neighbour joining tree using blosum62"
Как видно, программы дали одинаковые результаты в плане топологии. Можно заметить, что деревья построенные с помощью Neighbour joining tree using % identity с использованием матрицы BLOSUM62, аналогичны тем, что построенны без матрицы. Жель конечно, что программа не укореняет деревья.
Average distance:ветви, котроые появились в новом: нет ; ветви, которые отсудствуют в новом: нет
Neighbour joining :ветви, котроые появились в новом: нет ; ветви, которые отсудствуют в новом: нет
При выравнивании Neighbour joining результат был, топологически схож с Average distance. Но Average distance строит деревья и прововодит укоренение.
Далее с помощью программы Mega методом "Maximum Parsimony", на основе полученного ранее множественного выравнивания, был построен еще один вариант филогенетического дерева.
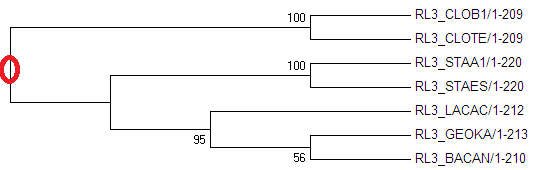
Изображние 7 Дерево построенное при помощи программы Mega, методом "Maximum Parsimony". Красный кружком отмечанно место укоренения
Дерево полученное дерево отличается от оригинального тем, что имеет кладу LACAC, GEOKA, BACAN, которая отсутствовала в оригинальном дереве.