
Занятие 12. Пакет Pftools
1. Необходимо приготовить входной файл, с которым мы работали на прошлом занятии.
Для этого используем команду noreturn -infile neznaika.msf -system unix -outfile neznaika_unix.msf
2. Полученный файл подаем на вход программе pfw:
pfw neznaika_unix.msf > neznaika_w.msf
Получаем файл с рассчетом весов строк (от исходного он отличается тем, что веса здесь уже не равны по единице).
3. Создаем профиль программой pfmake
Подаем полученный программой pfw файл программе pfmake: pfmake neznaika_w.msf /usr/share/pftools23/blosum62.cmp > neznaika_pfmake.txt
Получили файл neznaika_pfmake.txt
4. Подготавливаем файл с последовательностями в fasta-формате, в которых будет проводиться поиск.
Этот пункт был выполнен с помощью программы seqret sw-org:bacteria bacteria.fasta
5. Поиск по профилю
Поиск осуществлялся с помощью программы pfsearch. Поскольку с порогами 1.0 и 2.0 получалось слишком много находок, был выбран порог 3.0.
В итоге файл был получен командой: pfsearch -C3.0 -f neznaika_pfmake.txt bacteria.fasta > my3.pfsearch
6. Анализ результатов
Всего нашлось 1362 находок с весом больше 3
С весом больше 4 - 793
С весом больше 5 - 786
С весом больше 6 - 784
С весом больше 7 - 784
С весом больше 8 - 784
С весом больше 9 - 777
С весом больше 10 - 757
С весом больше 11 - 715
С весом больше 12 - 495
С весом больше 13 - 298
С весом больше 14 - 178
Число верных находок ("True positive hits", TP) 155
Число ложных находок ("False positive hits", FP) 1207
Число ненайденных белков подсемейства ("False negatives", FN) 0
Чувствительность TP/(TP+FN) 1
Селективность TP/(TP+FP) 0.1138
Excel файл
ROC-кривая:
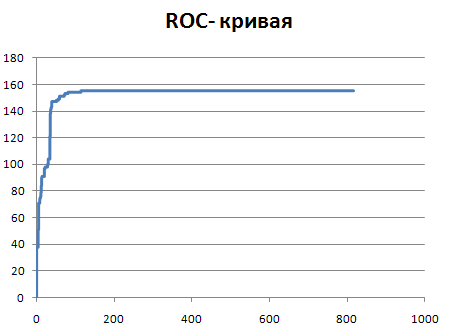
Даже по получившейся ROC-кривой видно, что данный профиль чуть-чуть лучше получившегося в прошлом задании. Также об этом нам говорит небольшая разница в селективности.


© Almukhametov Azat


