A-ДНК
В-ДНК
Z-ДНК
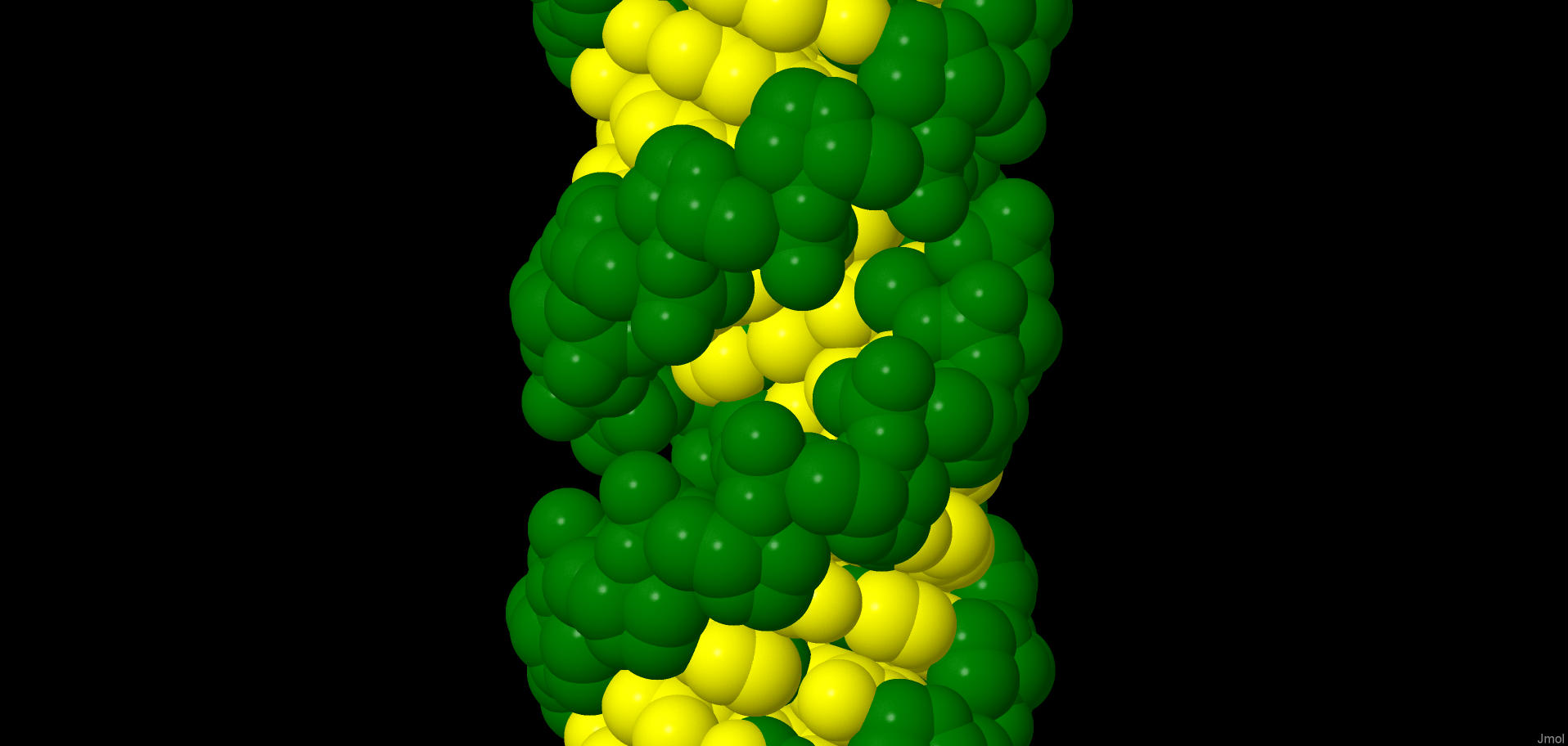
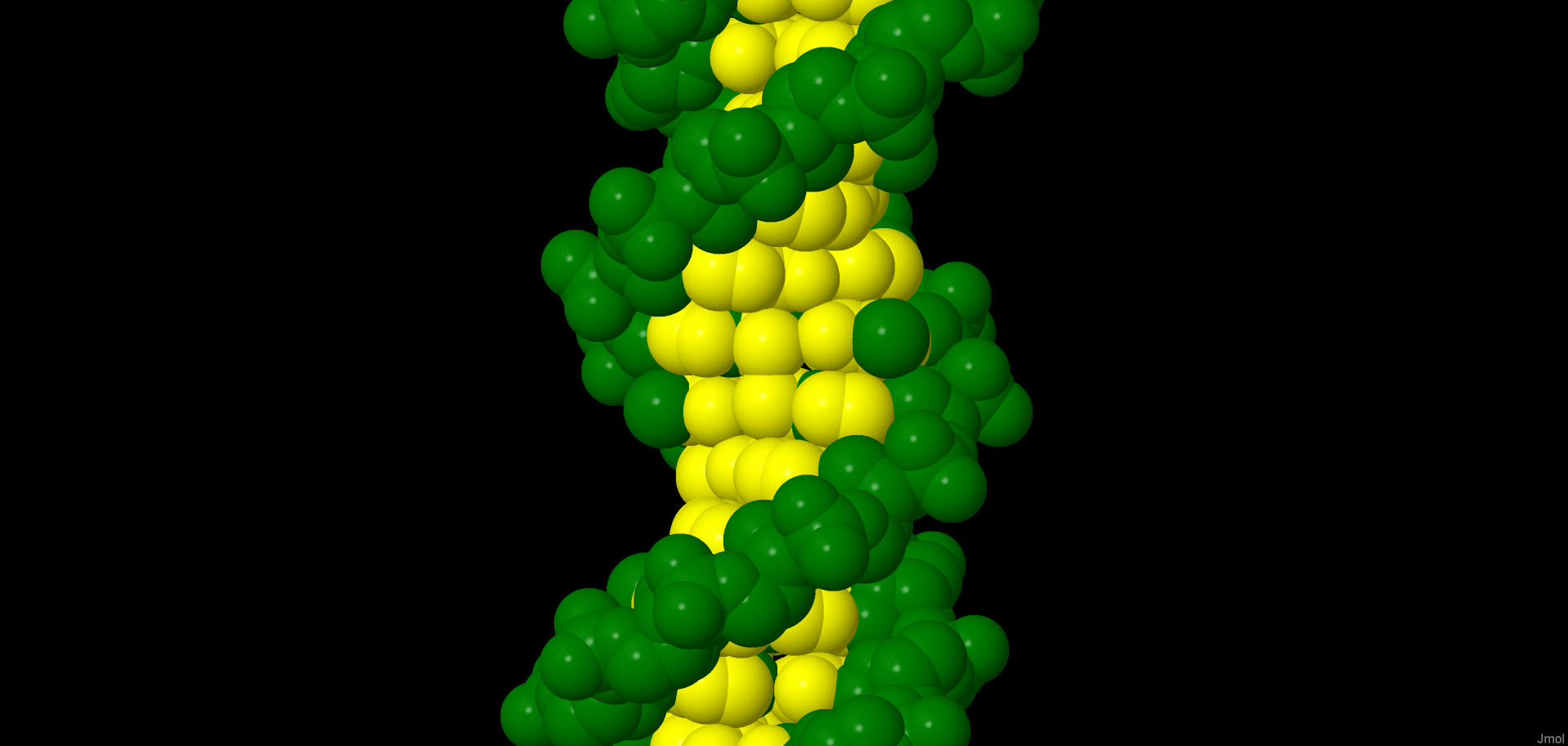
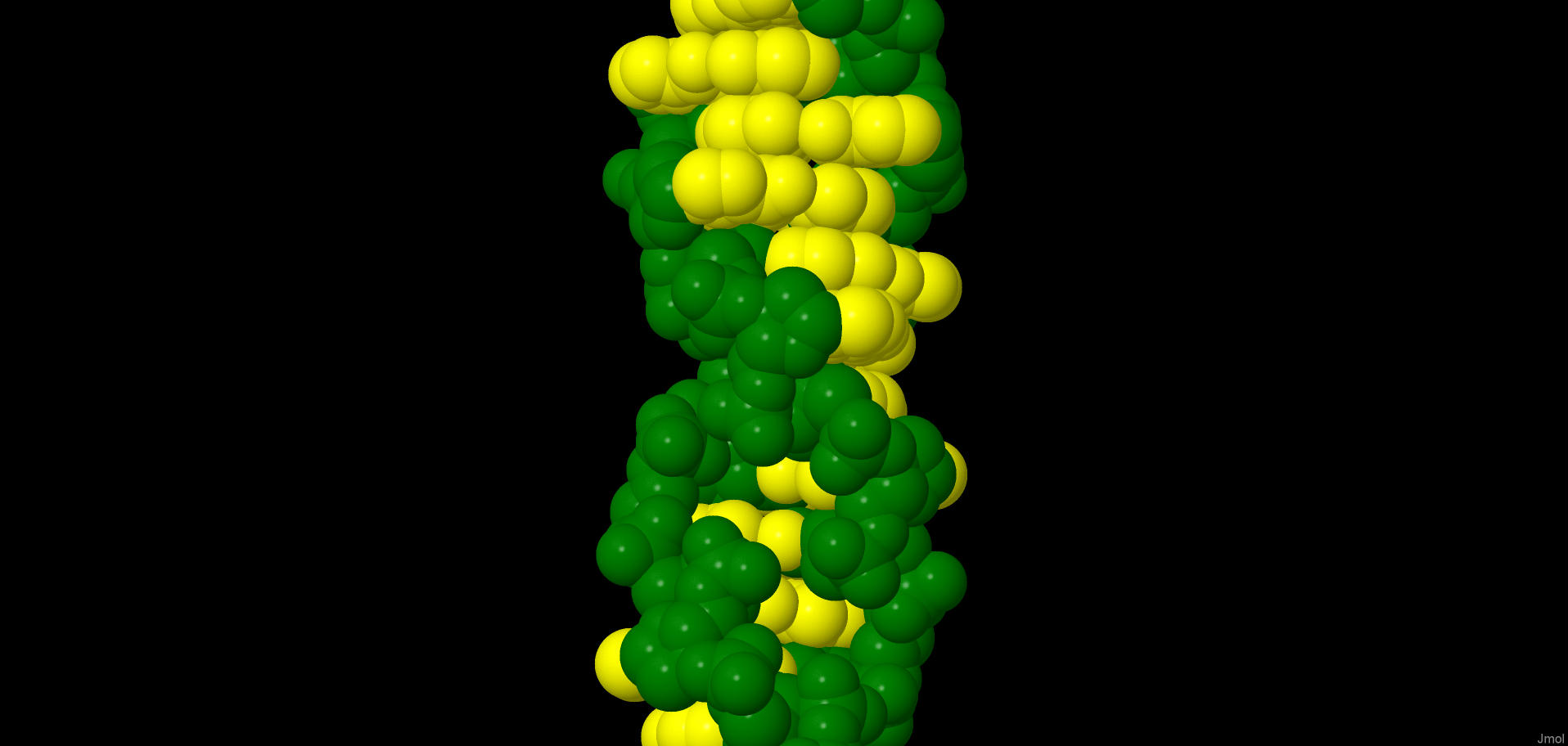
С помощью программы fiber из пакета 3DNA (работающего под операционной системой Linux) были построены три формы дуплекса ДНК: А, В и Z. Последовательность одной нити А-ДНК и В-ДНК представляет собой пять раз повторенную последовательность "GATC". А последовательность одной цепи Z-ДНК имеет вид десяти повтроений "GC", что является особенностью Z-формы.
A-ДНК | В-ДНК | Z-ДНК |
 |  |  |
| A-DNA (PDB) | B-DNA (PDB) | Z-DNA (PDB) |
По полученным в первом задании файлам с формами ДНК в программе Jmol изучались особенности расположения остатков цитозина относительной большой и малой бороздок. В программе MarvinScetch был нарисован цитозин из В-формы ДНК, в котором атомы смотрящие в сторону большой бородки покрашены красным, в сторону малой - синим, а остальные - черным. Для анализа был выбран остаток цитозина №28 и В цепи ДНК. Ниже в таблице представлены номера атомов того же самого цитозина №28, смотрящие в стороны большой и малой бороздок, во всех трех формах ДНК.
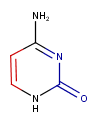
| Таблица №1 | |||
| Направление атомов | А-ДНК | В-ДНК | Z-ДНК |
| В сторону большой бороздки | O2, C2, N3, N1 | C5, C6 | C5, C6, C4, N4 |
| В сторону малой бороздки | C5, C6 | O2, C2, N3 | O2, C2, N1 |
| Остальные | C4, N4 | N1, C4, N4 | N3 |
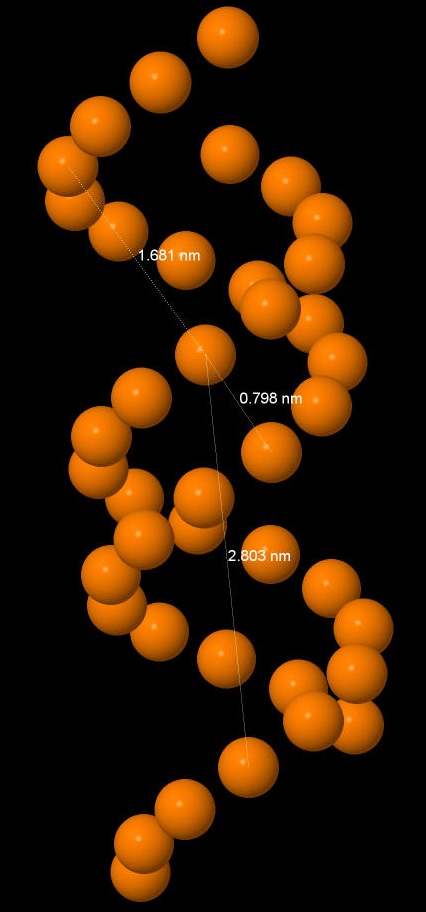
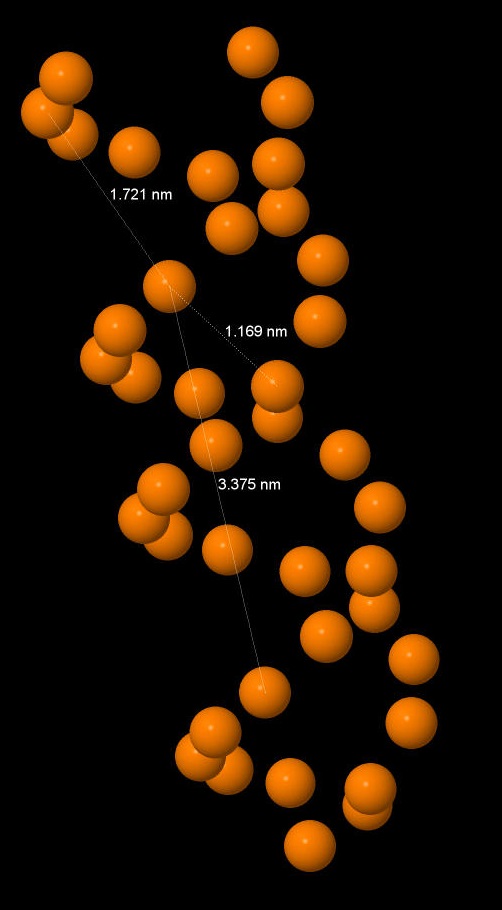
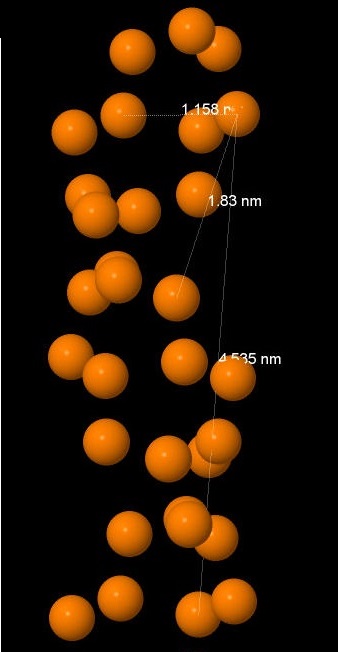
С помощью измерений в Jmol были выяснены представленные в таблице №2 параметры разлных форм ДНК. Все измерения проводились от фосфоров цепи А для А-формы 105, для В-ДНК 310, для Z-формы 64.
| Таблица №2 | |||
| Харатеристика | А-ДНК | B-ДНК | Z-ДНК |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) (между какими нуклеотидами измерялась) | 28,03 (105:А-329:А) | 33,75 (310:А-105:А) | 45,35 (64:А-329:А) |
| Число основания на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) (между какими нуклеотидами измерялась) | 7,98 (105:А-585:В) | 11,69 (310:А-575:В) | 11,58 (64:А-780:В) |
| Ширина малой бороздки (Å) (между какими нуклеотидами измерялась) | 16,81 (105:А-739:В) | 17,21 (310:А-433:В) | 18,3 (64:А-679:В) |
| А-ДНК | B-ДНК | Z-ДНК |
 |  |  |
С помошью программы Jmol (Measurements>Click for torsion (dihedral) measurement) были получены значения для торсионных углов цитозина из А- и В-форм ДНК. После полученные значения были соотнесены с теоретическими значениями данных углов. Как можно заметить, они в основном совпадают.
| Углы | α | β | γ | δ | ε | ς | χ |
| А-ДНК | 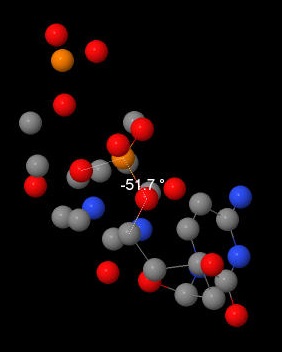 | 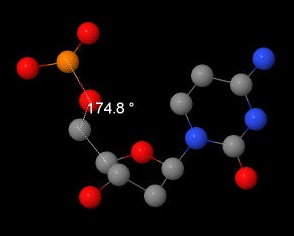 | 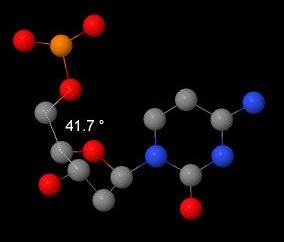 | 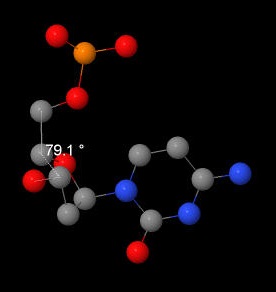 | 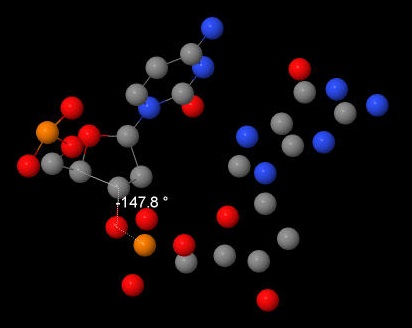 | 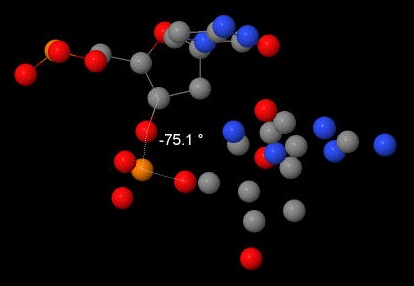 | 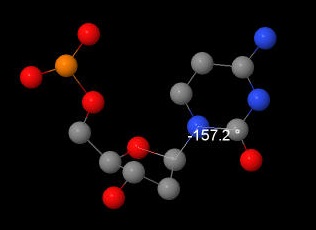 |
| B-ДНК | 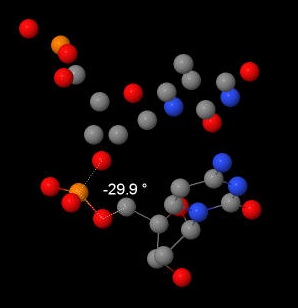 | 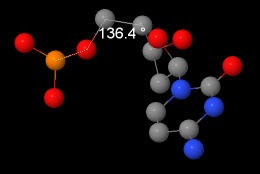 | 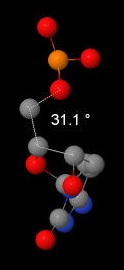 | 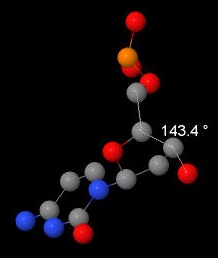 | 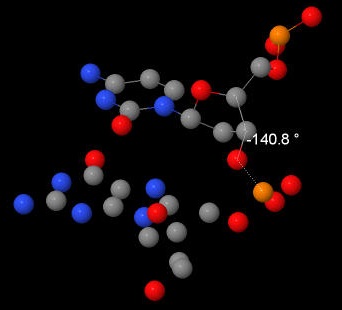 | 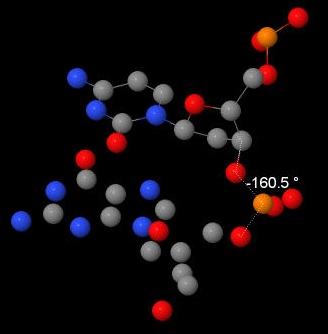 | 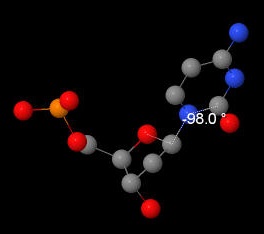 |
| Угол | А-ДНК (Jmol) | А-ДНК (теоритическая) | B-ДНК (Jmol) | В-ДНК (теоритическая) |
| α | -51,78 | 62 | -29,9 | 63 |
| β | 174,8 | 173 | 136,4 | 171 |
| γ | 41,7 | 52 | 31,1 | 54 |
| δ | 79,1 | 88/3 | 143,4 | 131/123 |
| ε | -147,8 | 178 | -140,8 | 155 |
| ς | -75,1 | -50 | -160,5 | -90 |
| χ | -157,2 | -160 | -98,0 | -117 |
С помощью программ find_pair и analyze из пакета 3DNA (find_pair -t XXXX.pdb stdout | analyze ) были определены торсионные углы всех нуклеотидов из тРНК (1qrt.pdb), всех трех форм ДНК (из первого задания) и из ДНК (цепей С, G), взятого из таблицы (1ksx.pdb. С помощью Excel было посчитано среднее арифметиеское каждого из торсионных углов (по всем нуклеотидам исключая концевые). Результаты представлены в таблице №4. Ссылка на файл Excel с рассчетами. Получилось, что для тРНК наиболее "деформированными нуклеотидами оказались: 4G и 21С(по углам α,β,γ) 10G (по углам α,β,γ и χ) 27G (по углам ε,ς) 5U (по углам β,ε) 28G (по углу ς) 11G (по углу χ). Интересно заметить, что углы δ и χ во всех нуклеотидах очень близки к среднему значению, в сравнении остальными углами. А для ДНК из белкого комплекса разброс значений торсионных углов оказался значительно меньше, "деформированными" можно назвать нуклеотиды: 12С (по углам α,γ) 8Т (по углу β) 7Т (по углам β,γ). При том для остальных углов δ,ε,ς,χ столь значительных отклонений в значениях углов не было обнаружено. Полученные средние значения углов представлены в таблице №5.
Судя по значениям торсионных углов структура тРНК больше похожа А-форму ДНК (разные формы ДНК см. в файле Excel).
| Таблица №5 | |||||||
| Углы | α | β | γ | δ | ε | ς | χ |
| tRNA | -40,592 | 22,392 | 53,3 | 85,307 | -118,152 | -63,152 | -121,759 |
| DNA | -20,33125 | 31,325 | 23,33125 | 139,26875 | -135,54375 | -87,6375 | -116,39375 |
Во вторичной структуре тРНК по файлу tRNA.out было найдено 4 стебля (stems).
Стебель №1: с нуклеотидов 2:В---71:В по нуклеотиды 7:В---66:В
Стебель №2: с нуклеотидов 49:В---65:В по нуклеотиды 53:В---61:В
Стебель №3: с нуклеотидов 37:В---33:В по нуклеотиды 44:В---26:В
Стебель №4: с нуклеотидов 10:В---25:В по нуклеотиды 12:В---23:В
Были обнаружены следующие неканонические пары: 55U---18G; 38U---32U; 44C---26A; 13A---45A; 14A---21A; 46U---47U.
Дополнительные водородные связи, стабилизирующие структуру, представлены в следующих парах: 54U---58A; 55U---18G; 13A---45A; 14A---21A; 15G---48C; 19G---56C.
Можно заметить, что часть дополнительных водородных связей как раз образованы неканоническими парами.
В том же файле tRNA.out было обнаружено, что самое большое значение площади перекрывани 12.26( 7.36) у двух последовательно идущих пар Section #0020 GC/AC, тогда как самое маленькое значение площали перекрывания 0.00( 0.00) у Section #0013 UA/UG; Section #0021 CG/CA; Section #0024 CA/AG; Section #0027 GG/CC; Section #0028 GU/UC.
Для пар №20 и 21 (с наибольшей и одной из наименьших форм перекрывания) с помощью программ (ex_str -20 stacking.pdb step20.pdb и stack2img -cdolt step20.pdb step20.ps) были получены 20PDB, 21PDB и 20PS, 21PS файлы соответсвенно.
Ниже представлены изображения этих пар.
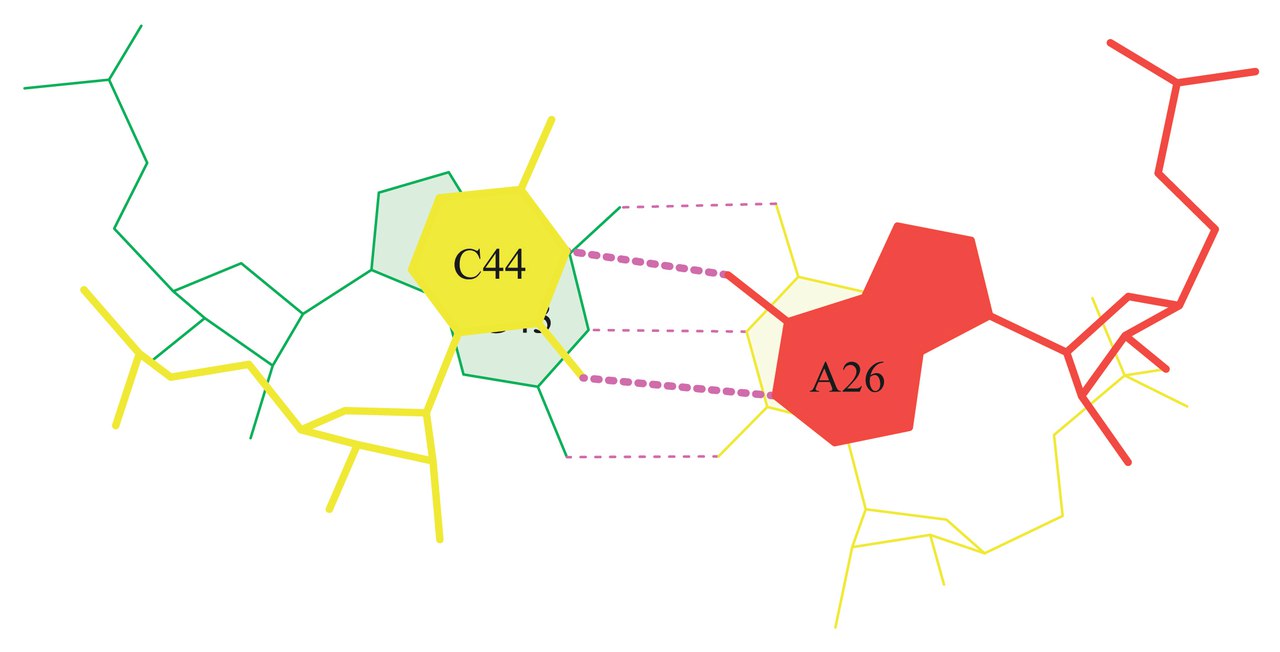 | 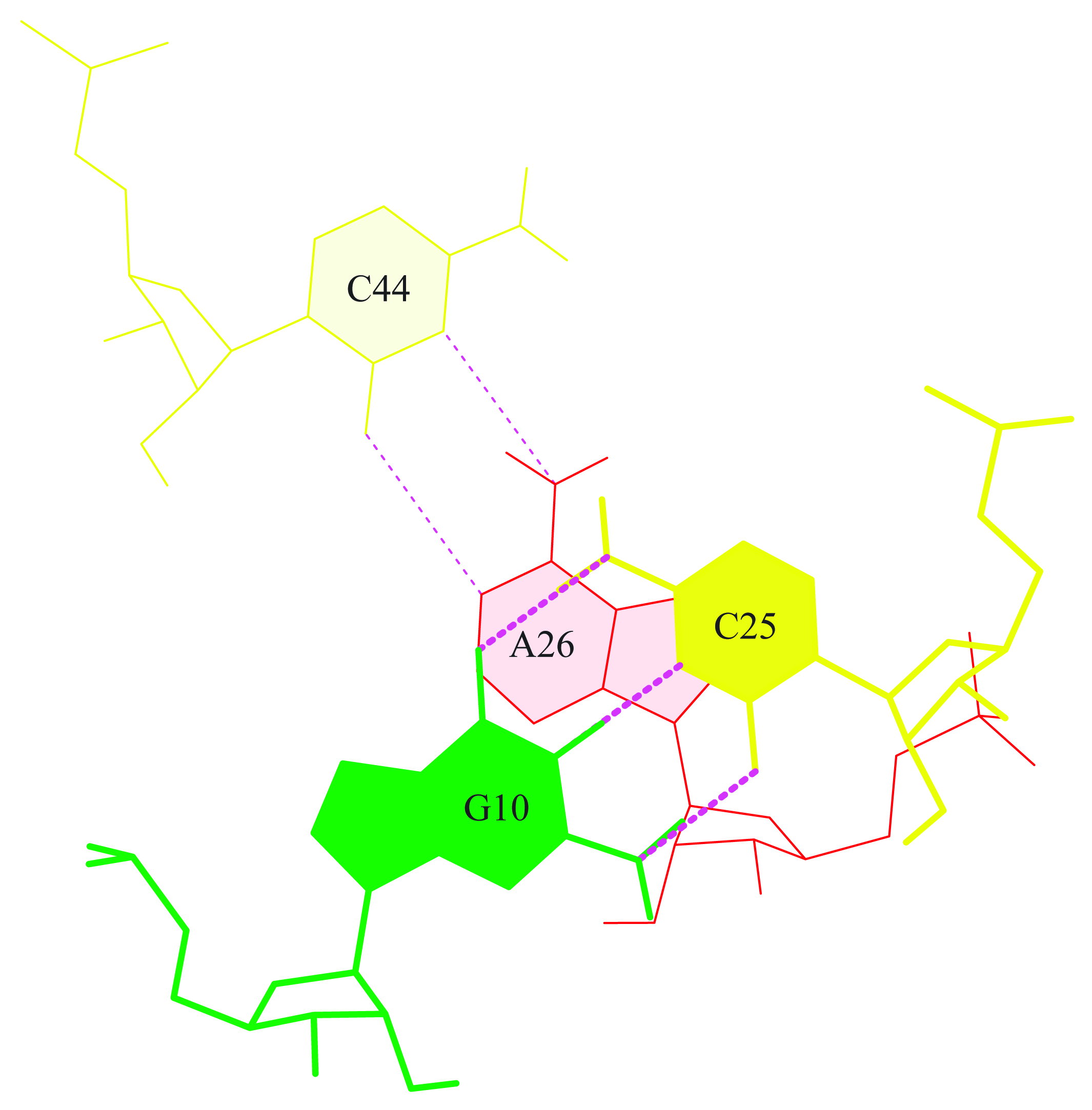 |