 [2]
[2]В данном практикуме анализировалась плазмида Rhodobacter sphaeroides strain MBTLJ-8 (индетификатор CP012965) длинной 107170 пар оснований.
Классификация Rhodobacter sphaeroides
Геном Rhodobacter sphaeroides имеет следующие характеристики:
Rhodobacter sphaeroides - вид пурпурных бактерий, которые могут получать энергию с помощью фотосинтеза и являются одним из основных организмов для изучения бактериального фотосинтеза. Вызывает интерес регуляция данного процесса. У R.sphaeroides есть сложная система определения давления кислорода. Геном представлен двумя хромосомами примерно по 3 Mb и 900 Kb и пятью плазмидами. Многие гены дублируются на обеих хромосомах, но регулируются по-разному. Многие открытые рамки считывания (ORFs) кодируют белки с неизвестными функциями. Обитают в глубоких озерах и стоячих водах.[1]
Для получения последовательности плазмиды в формате fasta была использована команда seqret из пакета EMBOSS (из базы данных embl):
seqret embl:CP012965 CP012965.fasta Файл
Для получения формата с особенностями была использована та же команда с параметром - feature:
seqret embl:СP012965 -feature CP012965.gff Файл
С помошью скрипта из файла с особенностями (.gff) была извлечена информация о белоккодирующих последовательностях: ориентация (+/- цепь), координаты начала и конца.
Полученный файл Файл с анотированными генами gff (I)
Далее эти гены были предсказаны с помощью программы Prodigal, для чего она была скачана на компьютер и запущена из командной строки (под far); -i входной файл в формате .fasta, -o выходной файл:
prodigal.windows.exe -i cp012965.fasta -o cp012965_gene
Полученный файл Файл с генами предсказанными prodigal (II)
Далее аннотированные и прдесказанные гены (файлы I и II) были сравнены сравнены с помощью Скрипта
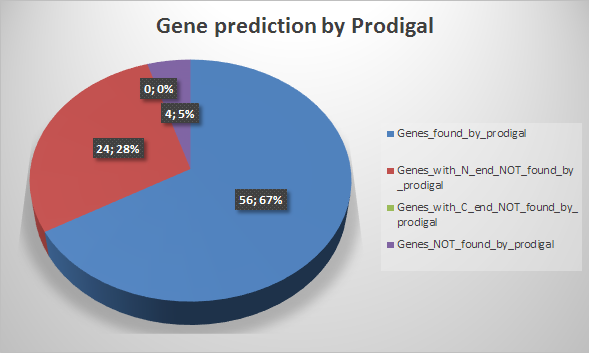
Данный скрип поочереди читает данные из gff и prodigal, сортируя каждую координату гена в свой список в зависимости от ориентации ("+" или "-" в файле gff и "complete" в выдаче prodigal). Потом соответствующие списки сравниваются и координаты сортируются по трем новым спискам: абсолютно совпадающие координаты, координаты генов с совпадающими С концами, координаты генов с совпадающими N концами. Затем считает все аннотированные гены (.gff), координаты которых не были найдены среди выдачи prodigal. В итоге она создает два выходных файла: 1ый - Results.txt с полученными координатами генов по трем спискам (совпадающим абсолютно, по С концам, по N концам) и количеством не найденных генов; 2ой - Stats.txt с требуемой статистикой по найденным генам (на основе него строилась диаграмма).
Полученные файлы: Results.txt Stats.txt
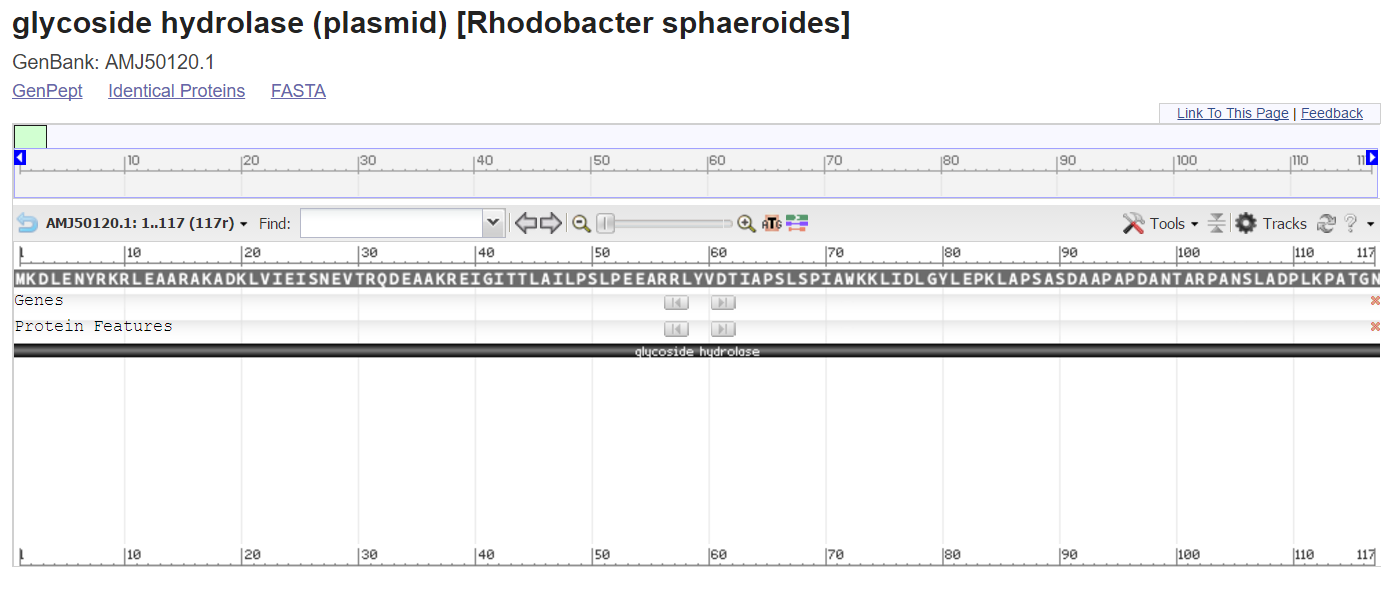
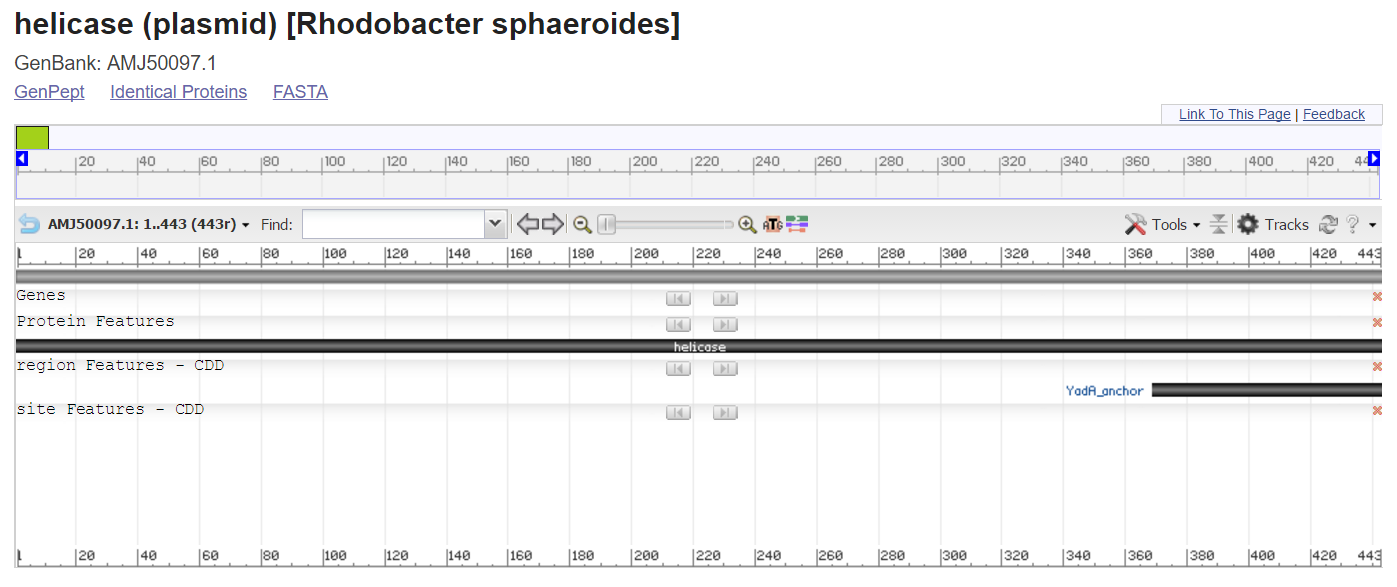
К примеру программой prodigal был не верно предсказан ген 18796..20127 (был предсказан как 18754..20127), кодирующий хеликазу (ID AMJ50097.1) по таблице генетического кода 11. Если использовать параметр -s в программе prodigal, который позволяет записать в файл все варианты предсказания гена, но то можно убедиться, что правильный вараинт там тоже есть, с тем же стартовым кодоном, но немного меньшим весом (см. рис ниже).

Аналогично был не верно прдесказан ген 47309..47662 (как 47216..47662), кодирующий гликозидную гидролазу (ID AMJ50120.1) тоже по таблице 11. И если обратиться к файлу со всеми вариантами предсказания генов, можно увидеть аналогичную картину, что нужный ген с таким же стартовым кодоном рассматривался, но был посчитан с меньшим весом (см. рис. ниже)