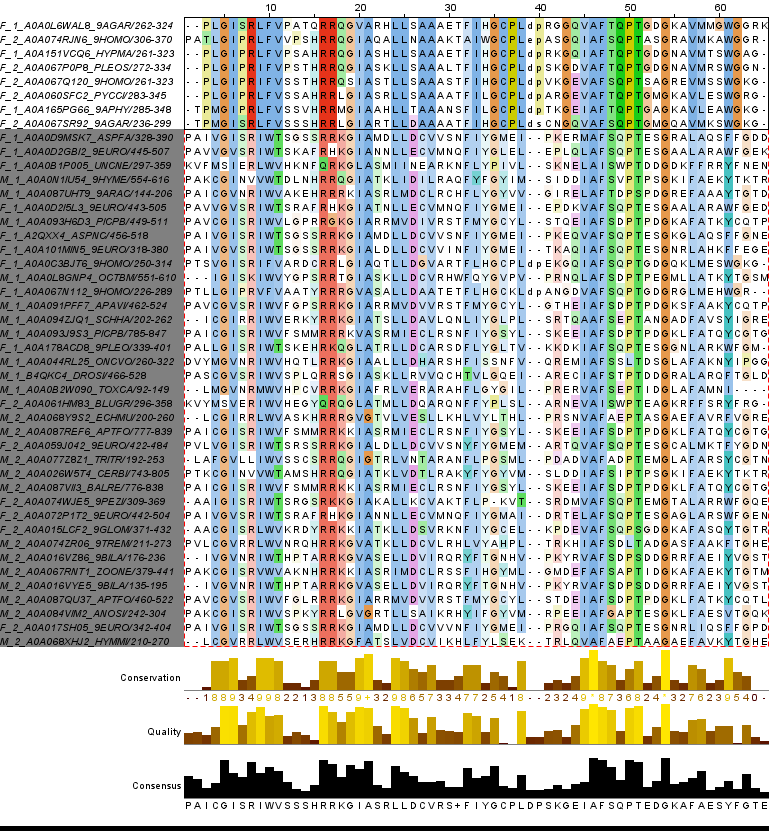
Для данного практикума было выбрано подсемейство белков, которое представляет собой отдельную кладу на дереве, имееет общие, диагностические черты в выравнивании, принадлежит к царству Fungi, правда представлено белками с двумя разными архитектурами. Его выравнивание и расположение на дереве представлено на рис1. и рис2. В данном семействе оказалось 8 белков.

Рис1. Выравнивание подсемейства
Рис2. Расположение подсемейства на дереве
Выравнивание последовательностей подсемейства в формате fasta
Далее был построен профиль этого подсемейства с использованием пакета HMMER. Он использует скрытые модели Маркова.
Построение профиля
| hmm2build profile.out family.fa |
Калибровка профиля
| hmm2calibrate profile.out |
Были получены все белки Uniprot, включаюшие мой домен, в формате fasta. Файл. По этим белкам был проведен поиск с помощью откалиброванного профиля.
| hmm2search profile.out uniprot-PF13880.fasta > find.out |
Полученный файл с результатами
Анализ полученный данных проводился средствами Excel. Файл Были посчитаные такие параметры как число истино положительных находок (TP), истинно отрицательных (TN), ложно положительных (FP), ложно отрицательных (FN). На основе них высчитвается чувствительность и специфичность.
В резльтате была построена ROC-кривая, прдестваляющая собой график в осях чувствительность (SE) и 1-специфичность (SP) (рис3.) Также была построена гистограмма весов находок (рис4.)
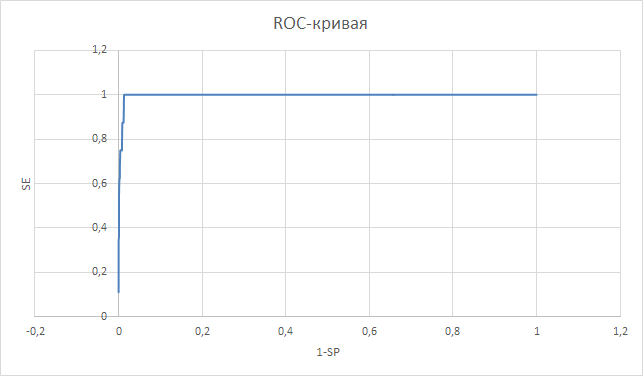
Рис3. ROC-кривая
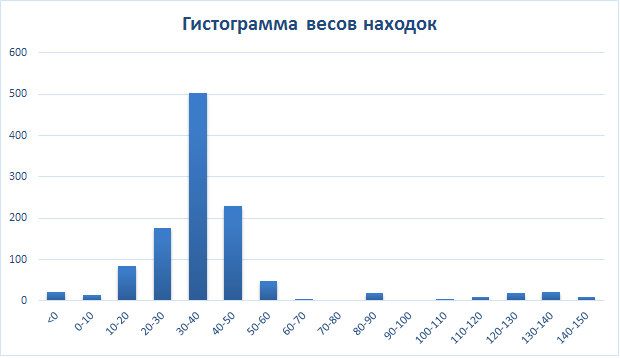
Рис4. Гистограмма весов находок
Порог был выбран таким образом, чтобы чувствительность и специфичность были одновременно максимальные max(SP+SE-1). Таким образом пороговый Score = 134,4. При нем SP=0,987 SE=1. Данный профиль позволяет хорошо выделять подсемейтво.
Таблица1. Характеристики порога
| На самом деле | Принадлежит подсемейству | Не принадлежит подсемейству | Сумма |
| Выше порога по профилю | 8 | 15 | 23 |
| Ниже порога по профилю | 0 | 1142 | 1142 |
| Сумма | 8 | 1157 | 1165 |