Домены и профили
Выбор домена и доменной архитектуры
Для анализа из базы данных Pfam был выбран ATP-grasp-like domain. Данный домен содержит сайт свяызвания АТФ и у некоторых бактерий входит в состав фермента, участвующего в синтезе глутатиона.
| ID | AC | seed | full | Uniprot | average length | average id% |
| ATP-grasp_6 | PF18419 | 28 | 307 | 1874 | 54 | 49 |
Была выбрана доменная архитектура: Glu_cys_ligase(PF04262), ATP-grasp_6 (Рис.1). Она характерна для 29 последовательностей, список AC.

После этого был скачан файл с последовательностями full.fasta, создан файл с последовательностями белков с выбранной архитектурой и построены гистограммы длин последовательностей.
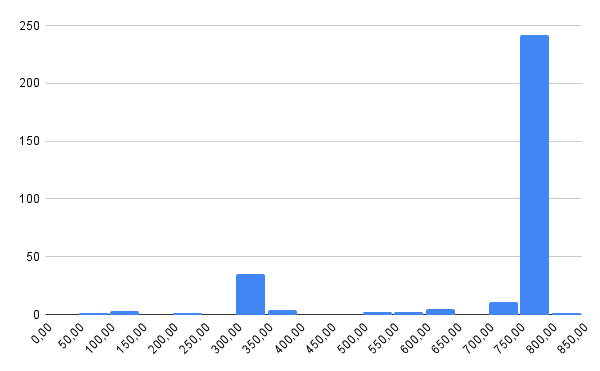
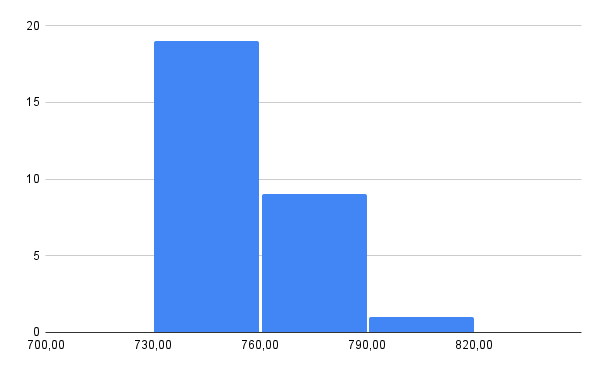
Выбранные белки имеют длину 730-820 аминокислот.
Построение HMM профиля
Последовательности с выбранной доменной архитектурой были выровнены в Jalview алгоритмом muscle. Далее была проведена ревизия - удалены высокосходные последовательности (Remove redundancy 80%), участок после второго домена (границы доменов в выравнивании 15-605), и ещё несколько последовательностей. Осталось 20 из 29 последовательностей.
С помощью пакета HMMER по данным последовательностям был построен и откалиброван HMM-профиль:
hmm2build -g out_hmm.txt align_domen_after.fasta hmm2calibrate out_hmm.txtДалее по этому профилю выполнялся поиск в файле со всеми последовательностями:
hmm2search out_hmm.txt full.fasta --cpu 1 > search_out.txtРезультаты.
После этого была составлена таблица, содержащая информацию о находках. Были посчитаны значения specificity, 1 - sensitivity и F1, построена ROC-кривая и график F1:
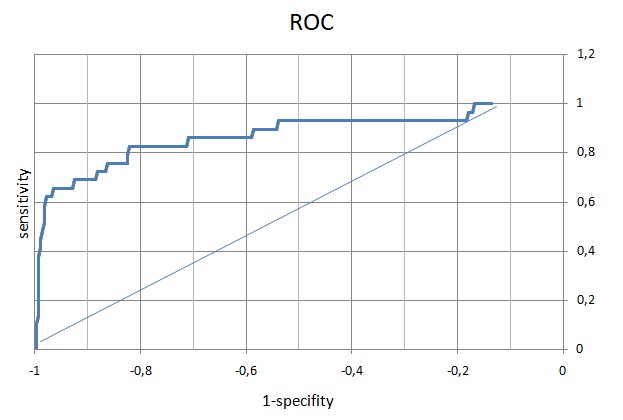
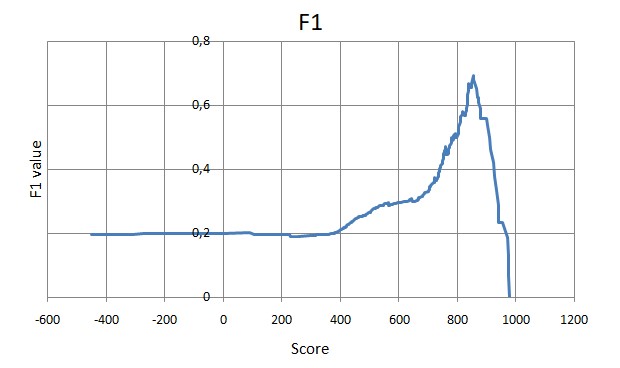
Порог веса по ROC-кривой 838,4. При таком пороге находятся 19 из 29 последовательностей с двудоменной архитектурой. Максимум F1 достигается при весе 853,7 - этому соответствует 18 из 19 белков. Такой порог мне кажется более удачным, так как в первом случае помимо одной правильной находки добавляется три неправильные.