Для построения HMM-профиля был использован пакет HMMER. Были использованы следующие команды:
hmm2build -g hmmout.txt seq_arch_align_revision.fa
hmm2calibrate hmmout.txt
В результате работы программы был получен HMM-профиль, имеющий длину 451 а.о. Далее с помощью HMMER был произведен поиск по HMM-профилю в выборке full с помощью следующей команды:
hmm2search --cpu=1 hmmout.txt PF11471_full_105.fasta > hmm_search_result.txt
Файл с результатом выдачи содержит 102 находки.
В итоге был создан файл с двумя листами. Лист 'final_table' содержит таблицу со следующими колонками: AC всех белков и информацию о том, имеет ли белок выбранную архитектуру, использовался ли белок для построения HMM-профиля, входит ли белок в список находок, вес находки и E-value находки. Лист 'search' содержит результаты выдачи поиска по HMM-профилю в выборке full и количество белков выборке full, количество находок и количество белков с выбранной архетиктурой.
Проверка HMM-профиля.
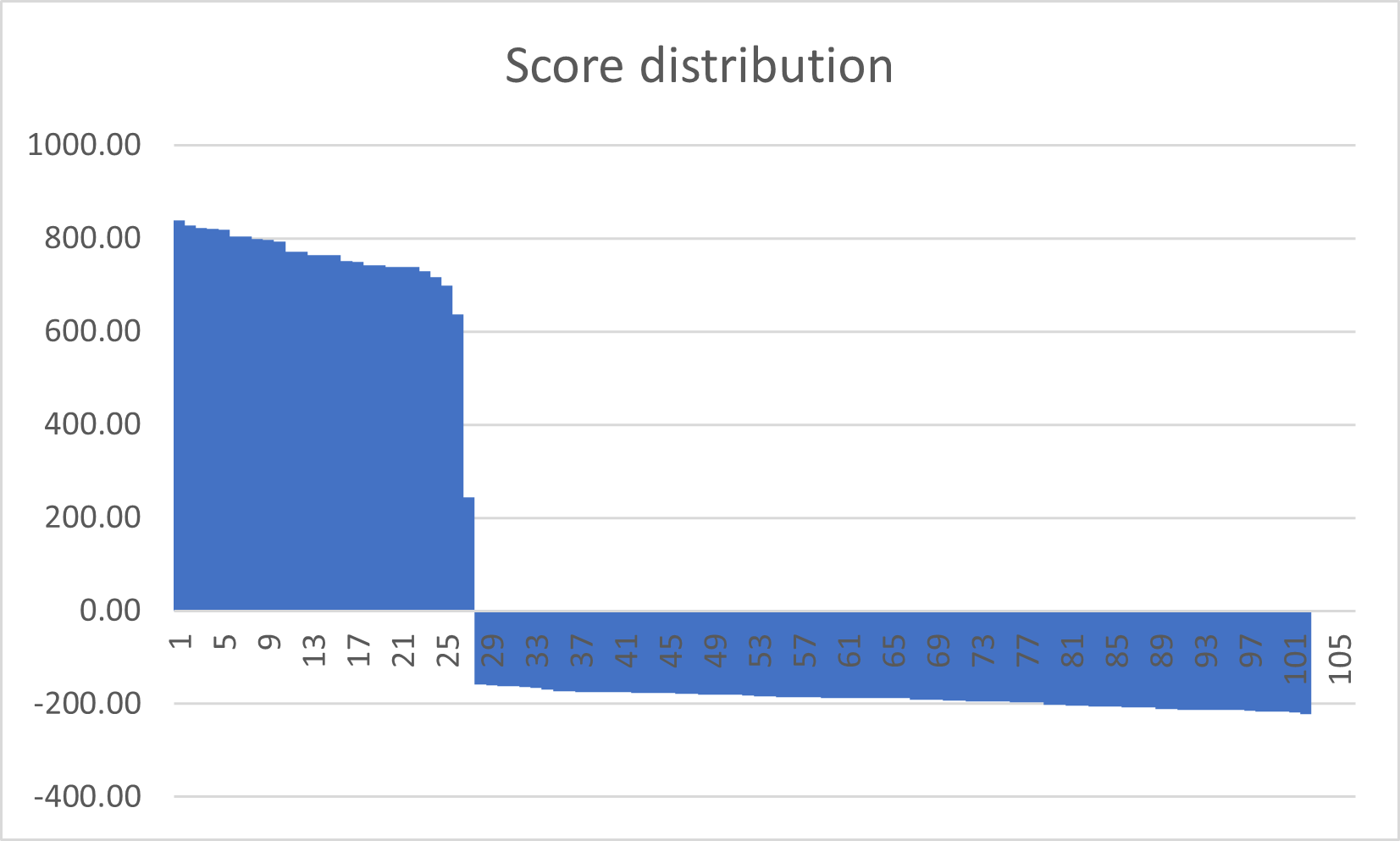
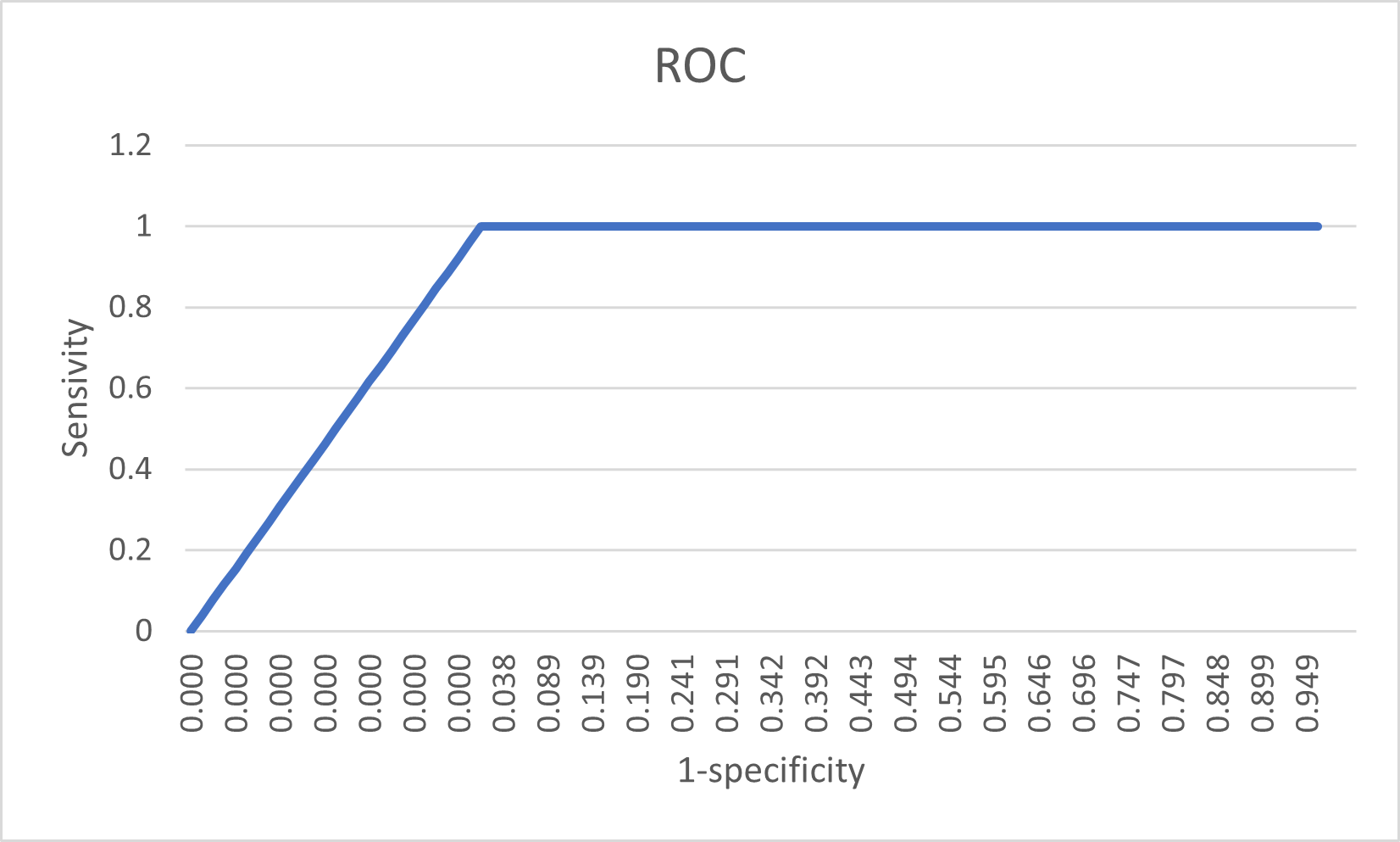
Cначала с помощью Excel был построен график распределения весов (рис. 1). На диаграмме можно наблюдать 'ступеньку падения' веса между -159.1 и 244.7 значениями веса. Можно предположить, что порог веса находится в данном интервале. С помощью построения ROC-кривой оценим данное предположение. В таблицу из листа 'search' была добавлена колонка с информацией о принадлежности белка к выбранной архетиктуре ('y' принадлежит, 'n' нет). Далее для каждого значения веса (считаем что каждый вес является порогом) были вычислены 1-специфичность и чувствительность и затем построена ROC-кривая (рис.2). Для определения порога с оптимальными значениями чувствительности и специфичности для каждого веса было определено значение F1. Максимальное значение F1 равное 1 достигается при значении веса 244.70, данное значение как раз и является порогом, разделяющим белки с выбранной архетиктурой и без архетиктуры.