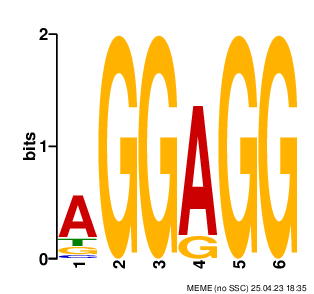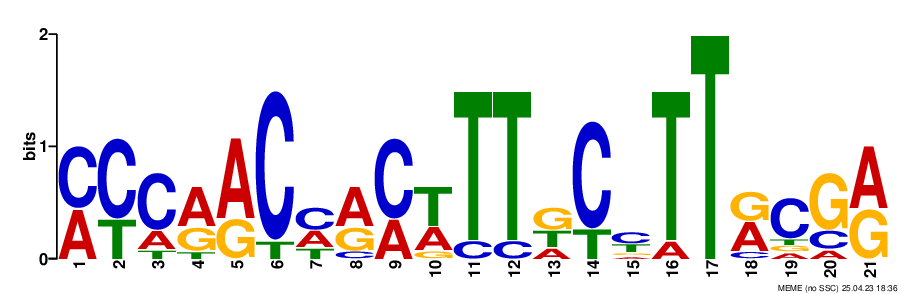Запуск MEME.
Для поиска мотивов был использован локальный meme на сервере kodomo. Программа была запущена следующей командой:
meme house_keeping_promoters.txt -dna -mod zoops -nmotifs 3 -minw 6 -maxw 50
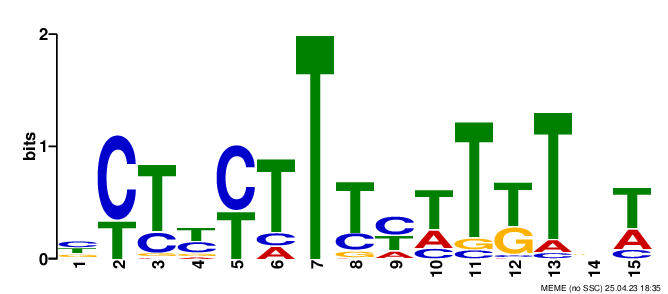
Программа нашла три мотива, и постороила их Logo, представленные на рисунках 1, 2, 3. С текстовой выдачей meme можно ознакомиться по ссылке. Только первый и второй мотивы имеют p-value меньше 0.05, с ними я и буду работать. Как видно из рисунка 4, первый мотив находится в 45-ти последовательностях, при этом 44-х из них в конце последовательности промотора (8-6 нуклеотидов от инициаторного кодона). Также по LOGO можно понять, что это высококонсеравтивный участок, а сама структура мотива aGGAGG очень сильно схожа с последовательностью Шайна — Дальгарно 1. P-value второго мотива равно 3.2e-008 и 10 из этих последовательностей находятся в -35 регионе, тем не менее визуально на данном Logo я не смог распознать эелементы -10 -35 консенсуса. С расположением мотивов в промоторной области можно ознакомиться по ссылке.