
Для выполнения данного практикума в качестве эукариотического организма рассматриваю организм Panthera uncia из 7 практикума. В ходе выполнения заданий использовала файлы из 3 задания предыдущего практикума.
С помощью текстового поиска в файле с последовательностями белков Panthera uncia нашла белок, аннотированный как δ-субъединица АТФ-синтазы (поиск нашёл данный белок по ключевой фразе "ATP synthase subunit delta", поиск по другим ключевым фразам ничего не выдавал). Белок является митохондриальным. Последовательность данного белка (XP_049480088.1) в fasta формате доступна по ссылке.
Идентификатор нуклеотидной записи (NW_026059575.1), к которой относится ген, кодирующий данный белок, был найден в файле с последовательностями генома с аннотацией по идентификатору этого белка (XP_049480088.1) в строчке LOCUS.
Ссылка на файл последовательностьи гена с небольшой окрестностью до и после самого гена в fasta-формате.
В этом разделе мне было интересно найти гомологов δ-субъединицу АТФ-синтазы у дальних родственников Panthera uncia, например, у первичноротых животных, так как Panthera uncia относится ко вторичноротым животным. Выбор был среди наиболее всем известных представителей первичноротых животных: Пауков (Araneae) и Пчёл (Apoidea). Я выбрала Пчёл, так как Пауки мне не очень приятны.
Поиск гомологов осуществлялся за счёт разных алгоритов BLAST на сайте NCBI. В качестве базы данных для поиска выбрала известную RefSeq Genome Database, так как в ней находятся только аннотированные последовательности. Всего оказалось 29 таких сборок для Пчёл в базе RefSeq Genome Database, в которых, предполагаем, есть δ-субъединица АТФ-синтаза, поэтому ожидаемое число гомологов 29.
Первый алгоритм, который был примён - это blastn, так как он ориентирован на поиск схожести даже у сильно различающихся последовательностей. На вход программе подавался файл последовательностьи исследуемого гена с небольшой окрестностью до и после самого гена в fasta-формате (никакие параметры не изменялись при поиске, в том числе Word size, равный 11).
Всего было обнаружено 8 находок, выравнивание этих 8 находок произошло на одном и том же участке, Query Cover составляет всего лишь 1%, но среди находок нет генов именно δ-субъединицы. Ссылка на файл с результатами.

В целом, были получены ожидаемые результаты от алгоритма blastn, так как он рабоает с небольшими участками для того, чтобы найти хоть какую-то схожесть у последовательностей. Также данный алгоритм не учитывает вырожденность генетического кода.
Следующий, применённный алгоритм - это tblastn, так как сравнивает запрос, представленный белком, с транслированными последовательностями из базы данных. На вход программе подавлся файл с последовательностью исследуемого белка (никакие параметры не изменялись при поиске, в том числе Word size, равный 5).
Всего было обнаружено 25 находок, у некоторых находок выровнялось 78% последовательности с последовательность исследуемого белка (минимальное выравнивание среди всех находок составило 39%), но среди находок опять нет генов именно δ-субъединицы. Ссылка на файл с результатами.
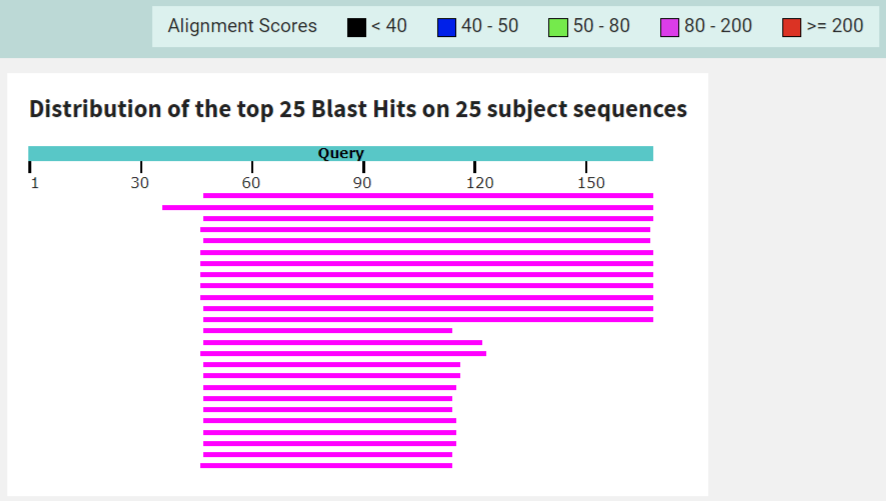
В целом, алгоритма tblastn сработал хорошо, он нашёл гомолги, судя по проценту покрытия. Однако эти находки в большинстве своём были транслированы с "unplaced genomic scaffold", тогда, опираясь на процент покрытия, последовательности, закодированные в этих scaffolds, являеются не чем-то неизвестным, а конкретно отвечают за образование δ-субъединицу АТФ-синтазы.
В этом разделе был проведён поиск гомологов 16S рРНК и 23S рРНК Escherichia coli у Panthera uncia.
16S рРНК входит в состав малой субъединицы (30S) прокариотической рибосомы (70S). Выполняет следующие функции:
23S рРНК входит в состав большой субъединицы (50S) прокариотической рибосомы (70S). Выполняет следующие функции:
Для начала было проведено индексирование последовательности генома Panthera uncia на собственном компьютере за счёт программы makeblastdb пакета BLAST+. Предварительно данный пакет был установлен на компьютер в соответствие с инструкцией установки BLAST+ для Windows (оказалось, что было достаточно следовать инструкции до пункта Installation включительно, потому что последующие шаги из инструкции приводили к тому, что программа просто не работала).
makeblastdb -in Punciagenomic.fna -dbtype nucl
пояснение: Punciagenomic.fna - это переименнованный файл GCF_023721935.1_Puncia_PCG_1.0_genomic.fna из предыдущего практикума
Далее был запущен blastn (данный алгоритм подходит для поиска похожих некодирующих белки нуклеотидных последовательностей даже среди неблизкородственных организмов):
blastn -task blastn -query 16S.fasta -db Punciagenomic.fna -out 16Stable.out -outfmt 7
blastn -task blastn -query 16S.fasta -db Punciagenomic.fna -out 16Stext.out
blastn -task blastn -query 23S.fasta -db Punciagenomic.fna -out 23Stable.out -outfmt 7
blastn -task blastn -query 23S.fasta -db Punciagenomic.fna -out 23Stext.out
Параметр -outfmt7 задаёт выдачу файла в виде таблице с комментариями. Без параметра - текстовый файл.
Результаты выдачи: таблица находок для 16S рРНК, текстовый файл с находками для 16S рРНК, таблица находок для 23S рРНК, текстовый файл с находок для 23S рРНК.
Для 16S рРНК было обнаружено 11 находок, а для 23S рРНК - тоже 11. Причём в таблице находок для 23S рРНК можно заметить, что 3 находки абсолютно идентичны, но одна из них обнаружена в митохондриальном геноме, другая - в хромосоме B4, а последняя - в Scaffold. При этом данная митохондриальная находка отвечает за синтез 16S рРНК, что можно понять, если искать данную находку по её же локусу - NC_010638.1 - в файл gffb из прошлого практикума, а другая часть последовательности данного локуса, отвечающая за синтез 12S рРНК, является находкой для 16S рРНК, что странно, потому что ожидаемо, что последовательность, отвечающая за синтез митохондриальной 16S рРНК Panthera uncia, окажется похожей на бактериальную 16S рРНК Escherichia coli, так как теория симбиогенеза гласит, что митохондрии - это некогда бывшие бактерии, поэтому могу преположить, что мною не были заданы параметры программе blastn, которые бы позволили выровнять последовательность митохондриальной 16S рРНК Panthera uncia относительно 16S рРНК Escherichia coli.
Я относила все находки с E-value больше 1 к случайным находкам, то есть не к гомологам, при этом ориентировалась ещё на аннотацию у найденных участков в сборке, если аннотации не было, то данные находки также оставляла под вопросом. Для 16S рРНК подошли 3 находки, 2 из которых отвечают за синтез 5.8S рРНК, однако у одной из них E-value был равен 6.00, но при этом она была аннотирована как часть последовательности 5.8S рРНК (остальные 2 находки с хорошим E-value были не аннотированы), другая находка имела E-value, также равеное 6.00, но была аннотирована как часть последовательности 12S рРНК (о ней говорилось выше). Для 23S рРНК - 1 митохондриальная находка (о ней писала в предыдущем абзаце).